Hinweis
Gehen Sie zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Release Highlights für scikit-learn 1.4#
Wir freuen uns, die Veröffentlichung von scikit-learn 1.4 bekannt zu geben! Viele Fehlerbehebungen und Verbesserungen wurden hinzugefügt, sowie einige neue Hauptfunktionen. Nachfolgend erläutern wir einige der wichtigsten Funktionen dieser Veröffentlichung. **Für eine vollständige Liste aller Änderungen** konsultieren Sie bitte die Release Notes.
Um die neueste Version zu installieren (mit pip)
pip install --upgrade scikit-learn
oder mit conda
conda install -c conda-forge scikit-learn
HistGradientBoosting unterstützt nativ kategorische DTypes in DataFrames#
ensemble.HistGradientBoostingClassifier und ensemble.HistGradientBoostingRegressor unterstützen nun direkt DataFrames mit kategorischen Merkmalen. Hier haben wir einen Datensatz mit einer Mischung aus kategorischen und numerischen Merkmalen
from sklearn.datasets import fetch_openml
X_adult, y_adult = fetch_openml("adult", version=2, return_X_y=True)
# Remove redundant and non-feature columns
X_adult = X_adult.drop(["education-num", "fnlwgt"], axis="columns")
X_adult.dtypes
age int64
workclass category
education category
marital-status category
occupation category
relationship category
race category
sex category
capital-gain int64
capital-loss int64
hours-per-week int64
native-country category
dtype: object
Durch Setzen von categorical_features="from_dtype" behandelt der Gradient Boosting-Klassifikator die Spalten mit kategorischen DTypes als kategorische Merkmale im Algorithmus
from sklearn.ensemble import HistGradientBoostingClassifier
from sklearn.metrics import roc_auc_score
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X_adult, y_adult, random_state=0)
hist = HistGradientBoostingClassifier(categorical_features="from_dtype")
hist.fit(X_train, y_train)
y_decision = hist.decision_function(X_test)
print(f"ROC AUC score is {roc_auc_score(y_test, y_decision)}")
ROC AUC score is 0.9280768177329133
Polars-Ausgabe in set_output#
Die Transformer von scikit-learn unterstützen nun die Polars-Ausgabe mit der set_output API.
import polars as pl
from sklearn.compose import ColumnTransformer
from sklearn.preprocessing import OneHotEncoder, StandardScaler
df = pl.DataFrame(
{"height": [120, 140, 150, 110, 100], "pet": ["dog", "cat", "dog", "cat", "cat"]}
)
preprocessor = ColumnTransformer(
[
("numerical", StandardScaler(), ["height"]),
("categorical", OneHotEncoder(sparse_output=False), ["pet"]),
],
verbose_feature_names_out=False,
)
preprocessor.set_output(transform="polars")
df_out = preprocessor.fit_transform(df)
df_out
print(f"Output type: {type(df_out)}")
Output type: <class 'polars.dataframe.frame.DataFrame'>
Unterstützung für fehlende Werte in Random Forest#
Die Klassen ensemble.RandomForestClassifier und ensemble.RandomForestRegressor unterstützen nun fehlende Werte. Beim Trainieren jedes einzelnen Baumes evaluiert der Splitting-Algorithmus jeden potenziellen Schwellenwert, wobei fehlende Werte zum linken und rechten Knoten gehen. Mehr Details im Benutzerhandbuch.
import numpy as np
from sklearn.ensemble import RandomForestClassifier
X = np.array([0, 1, 6, np.nan]).reshape(-1, 1)
y = [0, 0, 1, 1]
forest = RandomForestClassifier(random_state=0).fit(X, y)
forest.predict(X)
array([0, 0, 1, 1])
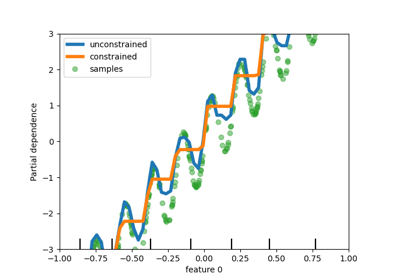
Unterstützung für monotone Einschränkungen in baumbasierten Modellen hinzufügen#
Während wir in scikit-learn 0.23 die Unterstützung für monotone Einschränkungen in histogrammbasierten Gradient Boosting hinzugefügt haben, unterstützen wir diese Funktion nun für alle anderen baumbasierten Modelle wie Bäume, Random Forests, Extra-Trees und exaktes Gradient Boosting. Hier zeigen wir diese Funktion für Random Forest bei einem Regressionsproblem.
import matplotlib.pyplot as plt
from sklearn.ensemble import RandomForestRegressor
from sklearn.inspection import PartialDependenceDisplay
n_samples = 500
rng = np.random.RandomState(0)
X = rng.randn(n_samples, 2)
noise = rng.normal(loc=0.0, scale=0.01, size=n_samples)
y = 5 * X[:, 0] + np.sin(10 * np.pi * X[:, 0]) - noise
rf_no_cst = RandomForestRegressor().fit(X, y)
rf_cst = RandomForestRegressor(monotonic_cst=[1, 0]).fit(X, y)
disp = PartialDependenceDisplay.from_estimator(
rf_no_cst,
X,
features=[0],
feature_names=["feature 0"],
line_kw={"linewidth": 4, "label": "unconstrained", "color": "tab:blue"},
)
PartialDependenceDisplay.from_estimator(
rf_cst,
X,
features=[0],
line_kw={"linewidth": 4, "label": "constrained", "color": "tab:orange"},
ax=disp.axes_,
)
disp.axes_[0, 0].plot(
X[:, 0], y, "o", alpha=0.5, zorder=-1, label="samples", color="tab:green"
)
disp.axes_[0, 0].set_ylim(-3, 3)
disp.axes_[0, 0].set_xlim(-1, 1)
disp.axes_[0, 0].legend()
plt.show()

Erweiterte Schätzer-Anzeigen#
Die Schätzer-Anzeigen wurden erweitert: Wenn wir uns forest ansehen, das oben definiert ist
forest
Man kann auf die Dokumentation des Schätzers zugreifen, indem man auf das Symbol "?" oben rechts im Diagramm klickt.
Zusätzlich ändert sich die Anzeige von orange zu blau, wenn der Schätzer angepasst ist. Diese Information kann auch durch Hovern über das Symbol "i" erhalten werden.
from sklearn.base import clone
clone(forest) # the clone is not fitted
Unterstützung für Metadaten-Routing#
Viele Meta-Schätzer und Kreuzvalidierungsroutinen unterstützen nun das Metadaten-Routing, das im Benutzerhandbuch aufgeführt ist. Dies ist beispielsweise, wie Sie eine verschachtelte Kreuzvalidierung mit Stichprobengewichten und GroupKFold durchführen können
import sklearn
from sklearn.datasets import make_regression
from sklearn.linear_model import Lasso
from sklearn.metrics import get_scorer
from sklearn.model_selection import GridSearchCV, GroupKFold, cross_validate
# For now by default metadata routing is disabled, and need to be explicitly
# enabled.
sklearn.set_config(enable_metadata_routing=True)
n_samples = 100
X, y = make_regression(n_samples=n_samples, n_features=5, noise=0.5)
rng = np.random.RandomState(7)
groups = rng.randint(0, 10, size=n_samples)
sample_weights = rng.rand(n_samples)
estimator = Lasso().set_fit_request(sample_weight=True)
hyperparameter_grid = {"alpha": [0.1, 0.5, 1.0, 2.0]}
scoring_inner_cv = get_scorer("neg_mean_squared_error").set_score_request(
sample_weight=True
)
inner_cv = GroupKFold(n_splits=5)
grid_search = GridSearchCV(
estimator=estimator,
param_grid=hyperparameter_grid,
cv=inner_cv,
scoring=scoring_inner_cv,
)
outer_cv = GroupKFold(n_splits=5)
scorers = {
"mse": get_scorer("neg_mean_squared_error").set_score_request(sample_weight=True)
}
results = cross_validate(
grid_search,
X,
y,
cv=outer_cv,
scoring=scorers,
return_estimator=True,
params={"sample_weight": sample_weights, "groups": groups},
)
print("cv error on test sets:", results["test_mse"])
# Setting the flag to the default `False` to avoid interference with other
# scripts.
sklearn.set_config(enable_metadata_routing=False)
cv error on test sets: [-0.30929661 -0.38510355 -0.2852123 -0.16594534 -0.26202999]
Verbesserte Speicher- und Laufzeiteffizienz für PCA bei dünnen Daten#
PCA kann nun dünne Matrizen nativ für den arpack-Solver handhaben, indem es scipy.sparse.linalg.LinearOperator nutzt, um die Materialisierung großer dünner Matrizen bei der Durchführung der Eigenwertzerlegung der Kovarianzmatrix des Datensatzes zu vermeiden.
from time import time
import scipy.sparse as sp
from sklearn.decomposition import PCA
X_sparse = sp.random(m=1000, n=1000, random_state=0)
X_dense = X_sparse.toarray()
t0 = time()
PCA(n_components=10, svd_solver="arpack").fit(X_sparse)
time_sparse = time() - t0
t0 = time()
PCA(n_components=10, svd_solver="arpack").fit(X_dense)
time_dense = time() - t0
print(f"Speedup: {time_dense / time_sparse:.1f}x")
Speedup: 3.7x
Gesamtlaufzeit des Skripts: (0 Minuten 1,890 Sekunden)
Verwandte Beispiele