Hinweis
Zum Ende springen, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Release Highlights für scikit-learn 0.23#
Wir freuen uns, die Veröffentlichung von scikit-learn 0.23 bekannt zu geben! Viele Fehlerbehebungen und Verbesserungen wurden hinzugefügt, sowie einige neue wichtige Funktionen. Nachfolgend detaillieren wir einige der wichtigsten Funktionen dieser Version. **Für eine vollständige Liste aller Änderungen** siehe die Release Notes.
Um die neueste Version zu installieren (mit pip)
pip install --upgrade scikit-learn
oder mit conda
conda install -c conda-forge scikit-learn
Generalisierte Lineare Modelle und Poisson-Verlust für Gradient Boosting#
Lang erwartete generalisierte lineare Modelle mit nicht-normalen Verlustfunktionen sind nun verfügbar. Insbesondere wurden drei neue Regressoren implementiert: PoissonRegressor, GammaRegressor und TweedieRegressor. Der Poisson-Regressor kann zur Modellierung von positiven Ganzzahlzählungen oder relativen Häufigkeiten verwendet werden. Lesen Sie mehr im Benutzerhandbuch. Zusätzlich unterstützt HistGradientBoostingRegressor einen neuen 'poisson'-Verlust.
import numpy as np
from sklearn.ensemble import HistGradientBoostingRegressor
from sklearn.linear_model import PoissonRegressor
from sklearn.model_selection import train_test_split
n_samples, n_features = 1000, 20
rng = np.random.RandomState(0)
X = rng.randn(n_samples, n_features)
# positive integer target correlated with X[:, 5] with many zeros:
y = rng.poisson(lam=np.exp(X[:, 5]) / 2)
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=rng)
glm = PoissonRegressor()
gbdt = HistGradientBoostingRegressor(loss="poisson", learning_rate=0.01)
glm.fit(X_train, y_train)
gbdt.fit(X_train, y_train)
print(glm.score(X_test, y_test))
print(gbdt.score(X_test, y_test))
0.35776189065725783
0.42425183539869415
Umfassende visuelle Darstellung von Schätzern#
Schätzer können nun in Notebooks visualisiert werden, indem die Option display='diagram' aktiviert wird. Dies ist besonders nützlich, um die Struktur von Pipelines und anderen zusammengesetzten Schätzern zusammenzufassen, mit Interaktivität zur Bereitstellung von Details. Klicken Sie auf das Beispielbild unten, um Pipeline-Elemente zu erweitern. Sehen Sie sich Visualizing Composite Estimators an, um zu erfahren, wie Sie diese Funktion nutzen können.
from sklearn import set_config
from sklearn.compose import make_column_transformer
from sklearn.impute import SimpleImputer
from sklearn.linear_model import LogisticRegression
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import OneHotEncoder, StandardScaler
set_config(display="diagram")
num_proc = make_pipeline(SimpleImputer(strategy="median"), StandardScaler())
cat_proc = make_pipeline(
SimpleImputer(strategy="constant", fill_value="missing"),
OneHotEncoder(handle_unknown="ignore"),
)
preprocessor = make_column_transformer(
(num_proc, ("feat1", "feat3")), (cat_proc, ("feat0", "feat2"))
)
clf = make_pipeline(preprocessor, LogisticRegression())
clf
Skalierbarkeits- und Stabilitätsverbesserungen für KMeans#
Der KMeans-Schätzer wurde komplett überarbeitet und ist nun deutlich schneller und stabiler. Außerdem ist der Elkan-Algorithmus nun kompatibel mit dünnbesetzten Matrizen. Der Schätzer verwendet OpenMP-basierte Parallelität anstelle von joblib, sodass der Parameter n_jobs keine Auswirkung mehr hat. Weitere Details zur Steuerung der Anzahl der Threads finden Sie in unseren Parallelism-Hinweisen.
import numpy as np
import scipy
from sklearn.cluster import KMeans
from sklearn.datasets import make_blobs
from sklearn.metrics import completeness_score
from sklearn.model_selection import train_test_split
rng = np.random.RandomState(0)
X, y = make_blobs(random_state=rng)
X = scipy.sparse.csr_matrix(X)
X_train, X_test, _, y_test = train_test_split(X, y, random_state=rng)
kmeans = KMeans(n_init="auto").fit(X_train)
print(completeness_score(kmeans.predict(X_test), y_test))
0.8483587861238999
Verbesserungen an den histogrammbasierten Gradient Boosting-Schätzern#
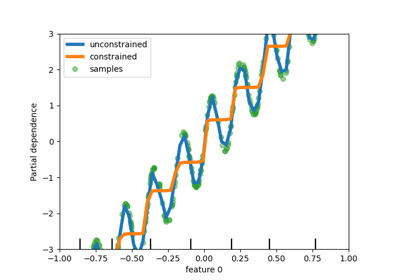
Es wurden verschiedene Verbesserungen an HistGradientBoostingClassifier und HistGradientBoostingRegressor vorgenommen. Neben dem oben erwähnten Poisson-Verlust unterstützen diese Schätzer nun Stichprobengewichte. Außerdem wurde ein automatisches Kriterium für Early-Stopping hinzugefügt: Early-Stopping ist standardmäßig aktiviert, wenn die Anzahl der Stichproben 10k überschreitet. Schließlich können Benutzer nun monotone Einschränkungen definieren, um die Vorhersagen basierend auf den Variationen spezifischer Merkmale einzuschränken. Im folgenden Beispiel konstruieren wir ein Ziel, das generell positiv mit dem ersten Merkmal korreliert ist, mit etwas Rauschen. Die Anwendung monontoner Einschränkungen ermöglicht es der Vorhersage, den globalen Effekt des ersten Merkmals zu erfassen, anstatt das Rauschen anzupassen. Ein Anwendungsbeispiel finden Sie unter Features in Histogram Gradient Boosting Trees.
import numpy as np
from matplotlib import pyplot as plt
from sklearn.ensemble import HistGradientBoostingRegressor
# from sklearn.inspection import plot_partial_dependence
from sklearn.inspection import PartialDependenceDisplay
from sklearn.model_selection import train_test_split
n_samples = 500
rng = np.random.RandomState(0)
X = rng.randn(n_samples, 2)
noise = rng.normal(loc=0.0, scale=0.01, size=n_samples)
y = 5 * X[:, 0] + np.sin(10 * np.pi * X[:, 0]) - noise
gbdt_no_cst = HistGradientBoostingRegressor().fit(X, y)
gbdt_cst = HistGradientBoostingRegressor(monotonic_cst=[1, 0]).fit(X, y)
# plot_partial_dependence has been removed in version 1.2. From 1.2, use
# PartialDependenceDisplay instead.
# disp = plot_partial_dependence(
disp = PartialDependenceDisplay.from_estimator(
gbdt_no_cst,
X,
features=[0],
feature_names=["feature 0"],
line_kw={"linewidth": 4, "label": "unconstrained", "color": "tab:blue"},
)
# plot_partial_dependence(
PartialDependenceDisplay.from_estimator(
gbdt_cst,
X,
features=[0],
line_kw={"linewidth": 4, "label": "constrained", "color": "tab:orange"},
ax=disp.axes_,
)
disp.axes_[0, 0].plot(
X[:, 0], y, "o", alpha=0.5, zorder=-1, label="samples", color="tab:green"
)
disp.axes_[0, 0].set_ylim(-3, 3)
disp.axes_[0, 0].set_xlim(-1, 1)
plt.legend()
plt.show()

Stichprobengewichtsunterstützung für Lasso und ElasticNet#
Die beiden linearen Regressoren Lasso und ElasticNet unterstützen nun Stichprobengewichte.
import numpy as np
from sklearn.datasets import make_regression
from sklearn.linear_model import Lasso
from sklearn.model_selection import train_test_split
n_samples, n_features = 1000, 20
rng = np.random.RandomState(0)
X, y = make_regression(n_samples, n_features, random_state=rng)
sample_weight = rng.rand(n_samples)
X_train, X_test, y_train, y_test, sw_train, sw_test = train_test_split(
X, y, sample_weight, random_state=rng
)
reg = Lasso()
reg.fit(X_train, y_train, sample_weight=sw_train)
print(reg.score(X_test, y_test, sw_test))
0.999791942438998
Gesamtlaufzeit des Skripts: (0 Minuten 0,551 Sekunden)
Verwandte Beispiele