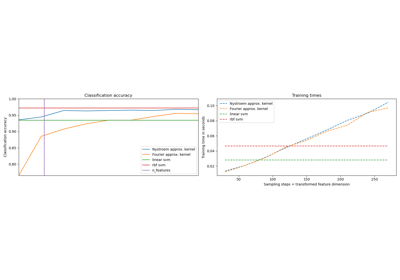
Nystroem#
- class sklearn.kernel_approximation.Nystroem(kernel='rbf', *, gamma=None, coef0=None, degree=None, kernel_params=None, n_components=100, random_state=None, n_jobs=None)[Quelle]#
Approximiert eine Kernel-Abbildung mittels einer Teilmenge der Trainingsdaten.
Konstruiert eine annähernde Feature-Map für einen beliebigen Kernel unter Verwendung einer Teilmenge der Daten als Basis.
Lesen Sie mehr im Benutzerhandbuch.
Hinzugefügt in Version 0.13.
- Parameter:
- kernelstr oder aufrufbar, Standard='rbf'
Kernel-Map, die angenähert werden soll. Ein aufrufbares Objekt sollte zwei Argumente und die an dieses Objekt übergebenen Schlüsselwortargumente als
kernel_paramsakzeptieren und eine Gleitkommazahl zurückgeben.- gammafloat, Standard=None
Gamma-Parameter für die RBF-, Laplacesche, Polynom-, exponentielle Chi2- und Sigmoid-Kernel. Die Interpretation des Standardwerts überlässt es dem Kernel; siehe Dokumentation für sklearn.metrics.pairwise. Von anderen Kerneln ignoriert.
- coef0float, Standard=None
Null-Koeffizient für Polynom- und Sigmoid-Kernel. Ignoriert von anderen Kerneln.
- degreefloat, Standard=None
Grad des Polynom-Kernels. Ignoriert von anderen Kerneln.
- kernel_paramsdict, Standard=None
Zusätzliche Parameter (Schlüsselwortargumente) für die als aufrufbares Objekt übergebene Kernel-Funktion.
- n_componentsint, Standardwert=100
Anzahl der zu konstruierenden Features. Wie viele Datenpunkte werden verwendet, um die Abbildung zu konstruieren.
- random_stateint, RandomState-Instanz oder None, default=None
Pseudo-Zufallszahlengenerator zur Steuerung der gleichmäßigen Stichprobe ohne Wiederholung von
n_componentsder Trainingsdaten zur Konstruktion des Basis-Kernels. Übergeben Sie eine Ganzzahl für reproduzierbare Ergebnisse über mehrere Funktionsaufrufe hinweg. Siehe Glossar.- n_jobsint, default=None
Die Anzahl der zu verwendenden Jobs für die Berechnung. Dies geschieht durch Aufteilung der Kernel-Matrix in
n_jobsgleichmäßige Scheiben und deren parallele Berechnung.Nonebedeutet 1, außer in einemjoblib.parallel_backendKontext.-1bedeutet die Verwendung aller Prozessoren. Siehe Glossar für weitere Details.Hinzugefügt in Version 0.24.
- Attribute:
- components_ndarray der Form (n_components, n_features)
Teilmenge der Trainingspunkte, die zur Konstruktion der Feature-Map verwendet wird.
- component_indices_ndarray der Form (n_components)
Indizes von
components_im Trainingsdatensatz.- normalization_ndarray der Form (n_components, n_components)
Normalisierungsmatrix, die für das Einbetten benötigt wird. Quadratwurzel der Kernel-Matrix auf
components_.- n_features_in_int
Anzahl der während des fits gesehenen Merkmale.
Hinzugefügt in Version 0.24.
- feature_names_in_ndarray mit Form (
n_features_in_,) Namen der während fit gesehenen Merkmale. Nur definiert, wenn
XMerkmalnamen hat, die alle Zeichenketten sind.Hinzugefügt in Version 1.0.
Siehe auch
AdditiveChi2SamplerApproximiert eine Merkmalsabbildung für den additiven Chi2-Kernel.
PolynomialCountSketchPolynomialer Kernel-Approximation mittels Tensor Sketch.
RBFSamplerApproximiert eine RBF-Kernel-Merkmalsabbildung mittels zufälliger Fourier-Merkmale.
SkewedChi2SamplerApproximiert eine Merkmalsabbildung für den "skewed chi-squared"-Kernel.
sklearn.metrics.pairwise.kernel_metricsListe der integrierten Kernel.
Referenzen
Williams, C.K.I. und Seeger, M. „Using the Nystroem method to speed up kernel machines“, Advances in neural information processing systems 2001
T. Yang, Y. Li, M. Mahdavi, R. Jin und Z. Zhou „Nystroem Method vs Random Fourier Features: A Theoretical and Empirical Comparison“, Advances in Neural Information Processing Systems 2012
Beispiele
>>> from sklearn import datasets, svm >>> from sklearn.kernel_approximation import Nystroem >>> X, y = datasets.load_digits(n_class=9, return_X_y=True) >>> data = X / 16. >>> clf = svm.LinearSVC() >>> feature_map_nystroem = Nystroem(gamma=.2, ... random_state=1, ... n_components=300) >>> data_transformed = feature_map_nystroem.fit_transform(data) >>> clf.fit(data_transformed, y) LinearSVC() >>> clf.score(data_transformed, y) 0.9987...
- fit(X, y=None)[Quelle]#
Schätzer an Daten anpassen.
Entnimmt eine Teilmenge von Trainingspunkten, berechnet den Kernel für diese und berechnet die Normalisierungsmatrix.
- Parameter:
- Xarray-ähnlich, Form (n_samples, n_features)
Trainingsdaten, wobei
n_samplesdie Anzahl der Stichproben undn_featuresdie Anzahl der Merkmale ist.- yarray-ähnlich, Form (n_samples,) oder (n_samples, n_outputs), Standardwert=None
Zielwerte (None für unüberwachte Transformationen).
- Gibt zurück:
- selfobject
Gibt die Instanz selbst zurück.
- fit_transform(X, y=None, **fit_params)[Quelle]#
An Daten anpassen, dann transformieren.
Passt den Transformer an
Xundymit optionalen Parameternfit_paramsan und gibt eine transformierte Version vonXzurück.- Parameter:
- Xarray-like der Form (n_samples, n_features)
Eingabestichproben.
- yarray-like der Form (n_samples,) oder (n_samples, n_outputs), Standardwert=None
Zielwerte (None für unüberwachte Transformationen).
- **fit_paramsdict
Zusätzliche Fit-Parameter. Nur übergeben, wenn der Estimator zusätzliche Parameter in seiner
fit-Methode akzeptiert.
- Gibt zurück:
- X_newndarray array der Form (n_samples, n_features_new)
Transformiertes Array.
- get_feature_names_out(input_features=None)[Quelle]#
Holt die Ausgabemerkmale für die Transformation.
Die Feature-Namen werden mit dem kleingeschriebenen Klassennamen präfixiert. Wenn der Transformer z.B. 3 Features ausgibt, dann sind die Feature-Namen:
["klassenname0", "klassenname1", "klassenname2"].- Parameter:
- input_featuresarray-like von str oder None, default=None
Wird nur verwendet, um die Feature-Namen mit den in
fitgesehenen Namen zu validieren.
- Gibt zurück:
- feature_names_outndarray von str-Objekten
Transformierte Merkmalnamen.
- get_metadata_routing()[Quelle]#
Holt das Metadaten-Routing dieses Objekts.
Bitte prüfen Sie im Benutzerhandbuch, wie der Routing-Mechanismus funktioniert.
- Gibt zurück:
- routingMetadataRequest
Ein
MetadataRequest, der Routing-Informationen kapselt.
- get_params(deep=True)[Quelle]#
Holt Parameter für diesen Schätzer.
- Parameter:
- deepbool, default=True
Wenn True, werden die Parameter für diesen Schätzer und die enthaltenen Unterobjekte, die Schätzer sind, zurückgegeben.
- Gibt zurück:
- paramsdict
Parameternamen, zugeordnet ihren Werten.
- set_output(*, transform=None)[Quelle]#
Ausgabecontainer festlegen.
Siehe Einführung in die set_output API für ein Beispiel zur Verwendung der API.
- Parameter:
- transform{“default”, “pandas”, “polars”}, default=None
Konfiguriert die Ausgabe von
transformundfit_transform."default": Standardausgabeformat eines Transformers"pandas": DataFrame-Ausgabe"polars": Polars-AusgabeNone: Die Transformationskonfiguration bleibt unverändert
Hinzugefügt in Version 1.4: Die Option
"polars"wurde hinzugefügt.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
- set_params(**params)[Quelle]#
Setzt die Parameter dieses Schätzers.
Die Methode funktioniert sowohl bei einfachen Schätzern als auch bei verschachtelten Objekten (wie
Pipeline). Letztere haben Parameter der Form<component>__<parameter>, so dass es möglich ist, jede Komponente eines verschachtelten Objekts zu aktualisieren.- Parameter:
- **paramsdict
Schätzer-Parameter.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
- transform(X)[Quelle]#
Feature-Map auf X anwenden.
Berechnet eine annähernde Feature-Map unter Verwendung des Kernels zwischen einigen Trainingspunkten und X.
- Parameter:
- Xarray-like der Form (n_samples, n_features)
Zu transformierende Daten.
- Gibt zurück:
- X_transformedndarray der Form (n_samples, n_components)
Transformierte Daten.
Galeriebeispiele#
Visualisierung der probabilistischen Vorhersagen eines VotingClassifier

Fehlende Werte mit Varianten von IterativeImputer imputieren

One-Class SVM vs. One-Class SVM mittels Stochastic Gradient Descent

Vergleich von Anomalieerkennungsalgorithmen zur Ausreißererkennung auf Toy-Datensätzen