GaussianNB#
- class sklearn.naive_bayes.GaussianNB(*, priors=None, var_smoothing=1e-09)[Quelle]#
Gaußscher Naiv Bayes (GaussianNB).
Kann Online-Updates von Modellparametern über
partial_fitdurchführen. Details zum Algorithmus, der zur Online-Aktualisierung von Merkmalsmittelwerten und -varianzen verwendet wird, finden Sie unter Stanford CS Tech Report STAN-CS-79-773 von Chan, Golub und LeVeque.Lesen Sie mehr im Benutzerhandbuch.
- Parameter:
- priorsarray-like von Form (n_classes,), default=None
Prior-Wahrscheinlichkeiten der Klassen. Wenn angegeben, werden die Priors nicht entsprechend den Daten angepasst.
- var_smoothingfloat, Standard=1e-9
Anteil der größten Varianz aller Merkmale, der zu den Varianzen für die Berechnungstabilität addiert wird.
Hinzugefügt in Version 0.20.
- Attribute:
- class_count_ndarray der Form (n_classes,)
Anzahl der in jeder Klasse beobachteten Trainingsstichproben.
- class_prior_ndarray der Form (n_classes,)
Wahrscheinlichkeit jeder Klasse.
- classes_ndarray der Form (n_classes,)
Klassenbezeichnungen, die dem Klassifikator bekannt sind.
- epsilon_float
Absoluter additiver Wert zu den Varianzen.
- n_features_in_int
Anzahl der während des fits gesehenen Merkmale.
Hinzugefügt in Version 0.24.
- feature_names_in_ndarray mit Form (
n_features_in_,) Namen der während fit gesehenen Merkmale. Nur definiert, wenn
XMerkmalnamen hat, die alle Zeichenketten sind.Hinzugefügt in Version 1.0.
- var_ndarray der Form (n_classes, n_features)
Varianz jedes Merkmals pro Klasse.
Hinzugefügt in Version 1.0.
- theta_ndarray der Form (n_classes, n_features)
Mittelwert jedes Merkmals pro Klasse.
Siehe auch
BernoulliNBNaiv-Bayes-Klassifikator für multivariate Bernoulli-Modelle.
CategoricalNBNaiv-Bayes-Klassifikator für kategorische Merkmale.
ComplementNBKomplementärer Naive Bayes-Klassifikator.
MultinomialNBNaiv-Bayes-Klassifikator für multinomielle Modelle.
Beispiele
>>> import numpy as np >>> X = np.array([[-1, -1], [-2, -1], [-3, -2], [1, 1], [2, 1], [3, 2]]) >>> Y = np.array([1, 1, 1, 2, 2, 2]) >>> from sklearn.naive_bayes import GaussianNB >>> clf = GaussianNB() >>> clf.fit(X, Y) GaussianNB() >>> print(clf.predict([[-0.8, -1]])) [1] >>> clf_pf = GaussianNB() >>> clf_pf.partial_fit(X, Y, np.unique(Y)) GaussianNB() >>> print(clf_pf.predict([[-0.8, -1]])) [1]
- fit(X, y, sample_weight=None)[Quelle]#
Trainiert den Gaußschen Naive Bayes gemäß X, y.
- Parameter:
- Xarray-like der Form (n_samples, n_features)
Trainingsvektoren, wobei
n_samplesdie Anzahl der Stichproben undn_featuresdie Anzahl der Merkmale ist.- yarray-like von Form (n_samples,)
Zielwerte.
- sample_weightarray-like der Form (n_samples,), Standardwert=None
Gewichte, die auf einzelne Stichproben angewendet werden (1. für ungewichtet).
Hinzugefügt in Version 0.17: Gaußscher Naive Bayes unterstützt das Training mit sample_weight.
- Gibt zurück:
- selfobject
Gibt die Instanz selbst zurück.
- get_metadata_routing()[Quelle]#
Holt das Metadaten-Routing dieses Objekts.
Bitte prüfen Sie im Benutzerhandbuch, wie der Routing-Mechanismus funktioniert.
- Gibt zurück:
- routingMetadataRequest
Ein
MetadataRequest, der Routing-Informationen kapselt.
- get_params(deep=True)[Quelle]#
Holt Parameter für diesen Schätzer.
- Parameter:
- deepbool, default=True
Wenn True, werden die Parameter für diesen Schätzer und die enthaltenen Unterobjekte, die Schätzer sind, zurückgegeben.
- Gibt zurück:
- paramsdict
Parameternamen, zugeordnet ihren Werten.
- partial_fit(X, y, classes=None, sample_weight=None)[Quelle]#
Inkrementelles Training auf einem Stapel von Stichproben.
Diese Methode wird voraussichtlich mehrmals hintereinander auf verschiedenen Teilen eines Datensatzes aufgerufen, um Out-of-Core- oder Online-Lernen zu implementieren.
Dies ist besonders nützlich, wenn der gesamte Datensatz zu groß ist, um gleichzeitig in den Speicher zu passen.
Diese Methode hat einige Leistungseinbußen und numerische Stabilitätsprobleme, daher ist es besser, partial_fit auf möglichst großen Datenstapeln aufzurufen (solange sie in das Speicherbudget passen), um die Overhead-Kosten zu verbergen.
- Parameter:
- Xarray-like der Form (n_samples, n_features)
Trainingsvektoren, wobei
n_samplesdie Anzahl der Stichproben undn_featuresdie Anzahl der Merkmale ist.- yarray-like von Form (n_samples,)
Zielwerte.
- classesarray-like der Form (n_classes,), Standard=None
Liste aller Klassen, die im y-Vektor vorkommen können.
Muss beim ersten Aufruf von partial_fit bereitgestellt werden, kann bei nachfolgenden Aufrufen weggelassen werden.
- sample_weightarray-like der Form (n_samples,), Standardwert=None
Gewichte, die auf einzelne Stichproben angewendet werden (1. für ungewichtet).
Hinzugefügt in Version 0.17.
- Gibt zurück:
- selfobject
Gibt die Instanz selbst zurück.
- predict(X)[Quelle]#
Führt die Klassifizierung auf einem Array von Testvektoren X durch.
- Parameter:
- Xarray-like der Form (n_samples, n_features)
Die Eingabestichproben.
- Gibt zurück:
- Cndarray der Form (n_samples,)
Vorhergesagte Zielwerte für X.
- predict_joint_log_proba(X)[Quelle]#
Gibt die Gelenk-Log-Wahrscheinlichkeitsschätzungen für den Testvektor X zurück.
Für jede Zeile x von X und jede Klasse y gilt: Gelenk-Log-Wahrscheinlichkeit ist
log P(x, y) = log P(y) + log P(x|y),wobeilog P(y)die Prior-Wahrscheinlichkeit der Klasse undlog P(x|y)die klassenbedingte Wahrscheinlichkeit ist.- Parameter:
- Xarray-like der Form (n_samples, n_features)
Die Eingabestichproben.
- Gibt zurück:
- Cndarray von Form (n_samples, n_classes)
Gibt die Gelenk-Log-Wahrscheinlichkeit der Stichproben für jede Klasse im Modell zurück. Die Spalten entsprechen den Klassen in sortierter Reihenfolge, wie sie im Attribut classes_ erscheinen.
- predict_log_proba(X)[Quelle]#
Gibt Log-Wahrscheinlichkeitsschätzungen für den Testvektor X zurück.
- Parameter:
- Xarray-like der Form (n_samples, n_features)
Die Eingabestichproben.
- Gibt zurück:
- Carray-like der Form (n_samples, n_classes)
Gibt die Log-Wahrscheinlichkeit der Stichproben für jede Klasse im Modell zurück. Die Spalten entsprechen den Klassen in sortierter Reihenfolge, wie sie im Attribut classes_ erscheinen.
- predict_proba(X)[Quelle]#
Gibt Wahrscheinlichkeitsschätzungen für den Testvektor X zurück.
- Parameter:
- Xarray-like der Form (n_samples, n_features)
Die Eingabestichproben.
- Gibt zurück:
- Carray-like der Form (n_samples, n_classes)
Gibt die Wahrscheinlichkeit der Stichproben für jede Klasse im Modell zurück. Die Spalten entsprechen den Klassen in sortierter Reihenfolge, wie sie im Attribut classes_ erscheinen.
- score(X, y, sample_weight=None)[Quelle]#
Gibt die Genauigkeit für die bereitgestellten Daten und Bezeichnungen zurück.
Bei der Multi-Label-Klassifizierung ist dies die Subset-Genauigkeit, eine strenge Metrik, da für jede Stichprobe verlangt wird, dass jede Label-Menge korrekt vorhergesagt wird.
- Parameter:
- Xarray-like der Form (n_samples, n_features)
Teststichproben.
- yarray-like der Form (n_samples,) oder (n_samples, n_outputs)
Wahre Bezeichnungen für
X.- sample_weightarray-like der Form (n_samples,), Standardwert=None
Stichprobengewichte.
- Gibt zurück:
- scorefloat
Mittlere Genauigkeit von
self.predict(X)in Bezug aufy.
- set_fit_request(*, sample_weight: bool | None | str = '$UNCHANGED$') GaussianNB[Quelle]#
Konfiguriert, ob Metadaten für die
fit-Methode angefordert werden sollen.Beachten Sie, dass diese Methode nur relevant ist, wenn dieser Schätzer als Unter-Schätzer innerhalb eines Meta-Schätzers verwendet wird und Metadaten-Routing mit
enable_metadata_routing=Trueaktiviert ist (siehesklearn.set_config). Bitte lesen Sie das Benutzerhandbuch, um zu erfahren, wie der Routing-Mechanismus funktioniert.Die Optionen für jeden Parameter sind
True: Metadaten werden angefordert und, falls vorhanden, anfitübergeben. Die Anforderung wird ignoriert, wenn keine Metadaten vorhanden sind.False: Metadaten werden nicht angefordert und der Meta-Schätzer übergibt sie nicht anfit.None: Metadaten werden nicht angefordert und der Meta-Schätzer löst einen Fehler aus, wenn der Benutzer sie bereitstellt.str: Metadaten sollten mit diesem Alias an den Meta-Schätzer übergeben werden und nicht mit dem ursprünglichen Namen.
Der Standardwert (
sklearn.utils.metadata_routing.UNCHANGED) behält die bestehende Anforderung bei. Dies ermöglicht es Ihnen, die Anforderung für einige Parameter zu ändern und für andere nicht.Hinzugefügt in Version 1.3.
- Parameter:
- sample_weightstr, True, False, oder None, Standardwert=sklearn.utils.metadata_routing.UNCHANGED
Metadaten-Routing für den Parameter
sample_weightinfit.
- Gibt zurück:
- selfobject
Das aktualisierte Objekt.
- set_params(**params)[Quelle]#
Setzt die Parameter dieses Schätzers.
Die Methode funktioniert sowohl bei einfachen Schätzern als auch bei verschachtelten Objekten (wie
Pipeline). Letztere haben Parameter der Form<component>__<parameter>, so dass es möglich ist, jede Komponente eines verschachtelten Objekts zu aktualisieren.- Parameter:
- **paramsdict
Schätzer-Parameter.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
- set_partial_fit_request(*, classes: bool | None | str = '$UNCHANGED$', sample_weight: bool | None | str = '$UNCHANGED$') GaussianNB[Quelle]#
Konfiguriert, ob Metadaten für die
partial_fit-Methode angefordert werden sollen.Beachten Sie, dass diese Methode nur relevant ist, wenn dieser Schätzer als Unter-Schätzer innerhalb eines Meta-Schätzers verwendet wird und Metadaten-Routing mit
enable_metadata_routing=Trueaktiviert ist (siehesklearn.set_config). Bitte lesen Sie das Benutzerhandbuch, um zu erfahren, wie der Routing-Mechanismus funktioniert.Die Optionen für jeden Parameter sind
True: Metadaten werden angefordert und anpartial_fitübergeben, wenn sie bereitgestellt werden. Die Anforderung wird ignoriert, wenn keine Metadaten bereitgestellt werden.False: Metadaten werden nicht angefordert und der Meta-Schätzer übergibt sie nicht anpartial_fit.None: Metadaten werden nicht angefordert und der Meta-Schätzer löst einen Fehler aus, wenn der Benutzer sie bereitstellt.str: Metadaten sollten mit diesem Alias an den Meta-Schätzer übergeben werden und nicht mit dem ursprünglichen Namen.
Der Standardwert (
sklearn.utils.metadata_routing.UNCHANGED) behält die bestehende Anforderung bei. Dies ermöglicht es Ihnen, die Anforderung für einige Parameter zu ändern und für andere nicht.Hinzugefügt in Version 1.3.
- Parameter:
- klassenstr, True, False, oder None, Standard=sklearn.utils.metadata_routing.UNCHANGED
Metadaten-Routing für den Parameter
klasseninpartial_fit.- sample_weightstr, True, False, oder None, Standardwert=sklearn.utils.metadata_routing.UNCHANGED
Metadaten-Routing für den Parameter
sample_weightinpartial_fit.
- Gibt zurück:
- selfobject
Das aktualisierte Objekt.
- set_score_request(*, sample_weight: bool | None | str = '$UNCHANGED$') GaussianNB[Quelle]#
Konfiguriert, ob Metadaten für die
score-Methode angefordert werden sollen.Beachten Sie, dass diese Methode nur relevant ist, wenn dieser Schätzer als Unter-Schätzer innerhalb eines Meta-Schätzers verwendet wird und Metadaten-Routing mit
enable_metadata_routing=Trueaktiviert ist (siehesklearn.set_config). Bitte lesen Sie das Benutzerhandbuch, um zu erfahren, wie der Routing-Mechanismus funktioniert.Die Optionen für jeden Parameter sind
True: Metadaten werden angefordert und, falls vorhanden, anscoreübergeben. Die Anforderung wird ignoriert, wenn keine Metadaten vorhanden sind.False: Metadaten werden nicht angefordert und der Meta-Schätzer übergibt sie nicht anscore.None: Metadaten werden nicht angefordert und der Meta-Schätzer löst einen Fehler aus, wenn der Benutzer sie bereitstellt.str: Metadaten sollten mit diesem Alias an den Meta-Schätzer übergeben werden und nicht mit dem ursprünglichen Namen.
Der Standardwert (
sklearn.utils.metadata_routing.UNCHANGED) behält die bestehende Anforderung bei. Dies ermöglicht es Ihnen, die Anforderung für einige Parameter zu ändern und für andere nicht.Hinzugefügt in Version 1.3.
- Parameter:
- sample_weightstr, True, False, oder None, Standardwert=sklearn.utils.metadata_routing.UNCHANGED
Metadaten-Routing für den Parameter
sample_weightinscore.
- Gibt zurück:
- selfobject
Das aktualisierte Objekt.
Galeriebeispiele#

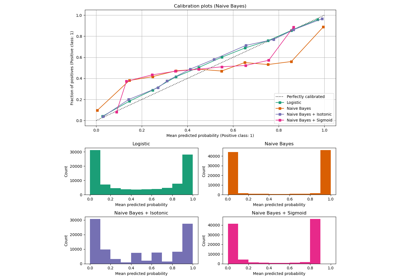
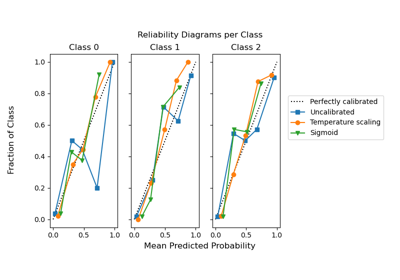
Wahrscheinlichkeitskalibrierung von Klassifikatoren

Lernkurven plotten und die Skalierbarkeit von Modellen prüfen