Birch#
- class sklearn.cluster.Birch(*, threshold=0.5, branching_factor=50, n_clusters=3, compute_labels=True)[Quelle]#
Implementiert den BIRCH-Clustering-Algorithmus.
Es ist ein speichereffizienter Online-Lernalgorithmus, der als Alternative zu
MiniBatchKMeansbereitgestellt wird. Er konstruiert eine Baumdatenstruktur, deren Clusterzentroide an den Blättern abgelesen werden. Diese können entweder die endgültigen Clusterzentroide sein oder als Eingabe für einen anderen Clustering-Algorithmus wieAgglomerativeClusteringverwendet werden.Mehr dazu im Benutzerhandbuch.
Hinzugefügt in Version 0.16.
- Parameter:
- thresholdfloat, default=0.5
Der Radius des Teilclusters, der durch das Zusammenführen einer neuen Stichprobe und des nächstgelegenen Teilclusters entsteht, sollte kleiner als der Schwellenwert sein. Andernfalls wird ein neuer Teilcluster gestartet. Ein sehr niedriger Wert fördert die Aufteilung, und umgekehrt.
- branching_factorint, default=50
Maximale Anzahl von CF-Teilclustern in jedem Knoten. Wenn eine neue Stichprobe eintrifft, die die Anzahl der Teilcluster über den Verzweigungsfaktor hinaus erhöht, wird dieser Knoten in zwei Knoten aufgeteilt, wobei die Teilcluster in jedem neu verteilt werden. Der übergeordnete Teilcluster dieses Knotens wird entfernt und zwei neue Teilcluster werden als Eltern der 2 aufgeteilten Knoten hinzugefügt.
- n_clustersint, Instanz eines sklearn.cluster-Modells oder None, default=3
Anzahl der Cluster nach dem finalen Clustering-Schritt, der die Teilcluster aus den Blättern als neue Stichproben behandelt.
None: Der finale Clustering-Schritt wird nicht durchgeführt und die Teilcluster werden wie sie sind zurückgegeben.sklearn.clusterEstimator : Wenn ein Modell bereitgestellt wird, wird das Modell trainiert, wobei die Teilcluster als neue Stichproben behandelt werden, und die ursprünglichen Daten werden der Bezeichnung des nächstgelegenen Teilclusters zugeordnet.int: Das trainierte Modell istAgglomerativeClusteringmitn_clusters, das gleich dem int ist.
- compute_labelsbool, default=True
Ob Labels für jede Anpassung berechnet werden sollen oder nicht.
- Attribute:
- root__CFNode
Wurzel des CFTree.
- dummy_leaf__CFNode
Startzeiger auf alle Blätter.
- subcluster_centers_ndarray
Zentroiden aller Teilcluster, die direkt aus den Blättern gelesen werden.
- subcluster_labels_ndarray
Labels, die den Teilcluster-Zentroiden nach ihrer globalen Clusterbildung zugewiesen werden.
- labels_ndarray der Form (n_samples,)
Array von Labels, die den Eingabedaten zugewiesen werden. Wenn partial_fit anstelle von fit verwendet wird, werden sie den letzten Datenstapel zugewiesen.
- n_features_in_int
Anzahl der während des fits gesehenen Merkmale.
Hinzugefügt in Version 0.24.
- feature_names_in_ndarray mit Form (
n_features_in_,) Namen der während fit gesehenen Merkmale. Nur definiert, wenn
XMerkmalnamen hat, die alle Zeichenketten sind.Hinzugefügt in Version 1.0.
Siehe auch
MiniBatchKMeansAlternative Implementierung, die inkrementelle Aktualisierungen der Positionen der Zentren mit Mini-Batches durchführt.
Anmerkungen
Die Baumdatenstruktur besteht aus Knoten, wobei jeder Knoten eine Anzahl von Teilclustern enthält. Die maximale Anzahl von Teilclustern in einem Knoten wird durch den Verzweigungsfaktor bestimmt. Jeder Teilcluster speichert eine lineare Summe, eine quadratische Summe und die Anzahl der Stichproben in diesem Teilcluster. Darüber hinaus kann jeder Teilcluster einen Knoten als Kind haben, wenn der Teilcluster kein Mitglied eines Blattknotens ist.
Für einen neuen Punkt, der die Wurzel betritt, wird er mit dem Teilcluster zusammengeführt, der ihm am nächsten liegt, und die lineare Summe, die quadratische Summe und die Anzahl der Stichproben dieses Teilclusters werden aktualisiert. Dies geschieht rekursiv, bis die Eigenschaften des Blattknotens aktualisiert sind.
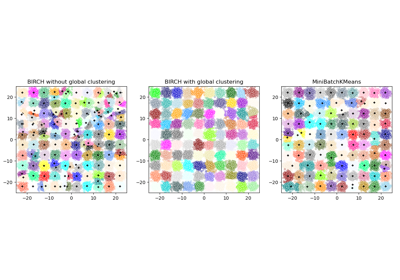
Siehe Vergleich von BIRCH und MiniBatchKMeans für einen Vergleich mit
MiniBatchKMeans.Referenzen
Tian Zhang, Raghu Ramakrishnan, Maron Livny BIRCH: An efficient data clustering method for large databases. https://www.cs.sfu.ca/CourseCentral/459/han/papers/zhang96.pdf
Roberto Perdisci JBirch - Java implementation of BIRCH clustering algorithm https://code.google.com/archive/p/jbirch
Beispiele
>>> from sklearn.cluster import Birch >>> X = [[0, 1], [0.3, 1], [-0.3, 1], [0, -1], [0.3, -1], [-0.3, -1]] >>> brc = Birch(n_clusters=None) >>> brc.fit(X) Birch(n_clusters=None) >>> brc.predict(X) array([0, 0, 0, 1, 1, 1])
Für einen Vergleich des BIRCH-Clustering-Algorithmus mit anderen Clustering-Algorithmen siehe Vergleich verschiedener Clustering-Algorithmen auf Spielzeugdatensätzen
- fit(X, y=None)[Quelle]#
Erstellen Sie einen CF-Baum für die Eingabedaten.
- Parameter:
- X{array-like, sparse matrix} der Form (n_samples, n_features)
Eingabedaten.
- yIgnoriert
Nicht verwendet, hier zur API-Konsistenz durch Konvention vorhanden.
- Gibt zurück:
- self
Angepasster Schätzer.
- fit_predict(X, y=None, **kwargs)[Quelle]#
Führt Clustering auf
Xdurch und gibt Cluster-Labels zurück.- Parameter:
- Xarray-like der Form (n_samples, n_features)
Eingabedaten.
- yIgnoriert
Wird nicht verwendet, ist aber aus Gründen der API-Konsistenz per Konvention vorhanden.
- **kwargsdict
Argumente, die an
fitübergeben werden sollen.Hinzugefügt in Version 1.4.
- Gibt zurück:
- labelsndarray der Form (n_samples,), dtype=np.int64
Clusterbeschriftungen.
- fit_transform(X, y=None, **fit_params)[Quelle]#
An Daten anpassen, dann transformieren.
Passt den Transformer an
Xundymit optionalen Parameternfit_paramsan und gibt eine transformierte Version vonXzurück.- Parameter:
- Xarray-like der Form (n_samples, n_features)
Eingabestichproben.
- yarray-like der Form (n_samples,) oder (n_samples, n_outputs), Standardwert=None
Zielwerte (None für unüberwachte Transformationen).
- **fit_paramsdict
Zusätzliche Fit-Parameter. Nur übergeben, wenn der Estimator zusätzliche Parameter in seiner
fit-Methode akzeptiert.
- Gibt zurück:
- X_newndarray array der Form (n_samples, n_features_new)
Transformiertes Array.
- get_feature_names_out(input_features=None)[Quelle]#
Holt die Ausgabemerkmale für die Transformation.
Die Feature-Namen werden mit dem kleingeschriebenen Klassennamen präfixiert. Wenn der Transformer z.B. 3 Features ausgibt, dann sind die Feature-Namen:
["klassenname0", "klassenname1", "klassenname2"].- Parameter:
- input_featuresarray-like von str oder None, default=None
Wird nur verwendet, um die Feature-Namen mit den in
fitgesehenen Namen zu validieren.
- Gibt zurück:
- feature_names_outndarray von str-Objekten
Transformierte Merkmalnamen.
- get_metadata_routing()[Quelle]#
Holt das Metadaten-Routing dieses Objekts.
Bitte prüfen Sie im Benutzerhandbuch, wie der Routing-Mechanismus funktioniert.
- Gibt zurück:
- routingMetadataRequest
Ein
MetadataRequest, der Routing-Informationen kapselt.
- get_params(deep=True)[Quelle]#
Holt Parameter für diesen Schätzer.
- Parameter:
- deepbool, default=True
Wenn True, werden die Parameter für diesen Schätzer und die enthaltenen Unterobjekte, die Schätzer sind, zurückgegeben.
- Gibt zurück:
- paramsdict
Parameternamen, zugeordnet ihren Werten.
- partial_fit(X=None, y=None)[Quelle]#
Online-Lernen. Verhindert den Neuaufbau des CFTree von Grund auf.
- Parameter:
- X{array-like, sparse matrix} von shape (n_samples, n_features), Standardwert=None
Eingabedaten. Wenn X nicht angegeben wird, wird nur der globale Clustering-Schritt durchgeführt.
- yIgnoriert
Nicht verwendet, hier zur API-Konsistenz durch Konvention vorhanden.
- Gibt zurück:
- self
Angepasster Schätzer.
- predict(X)[Quelle]#
Daten mithilfe der
centroids_von Teilclustern vorhersagen.Vermeiden Sie die Berechnung der Zeilennormen von X.
- Parameter:
- X{array-like, sparse matrix} der Form (n_samples, n_features)
Eingabedaten.
- Gibt zurück:
- labelsndarray von shape(n_samples,)
Beschriftete Daten.
- set_output(*, transform=None)[Quelle]#
Ausgabecontainer festlegen.
Siehe Einführung in die set_output API für ein Beispiel zur Verwendung der API.
- Parameter:
- transform{“default”, “pandas”, “polars”}, default=None
Konfiguriert die Ausgabe von
transformundfit_transform."default": Standardausgabeformat eines Transformers"pandas": DataFrame-Ausgabe"polars": Polars-AusgabeNone: Die Transformationskonfiguration bleibt unverändert
Hinzugefügt in Version 1.4: Die Option
"polars"wurde hinzugefügt.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
- set_params(**params)[Quelle]#
Setzt die Parameter dieses Schätzers.
Die Methode funktioniert sowohl bei einfachen Schätzern als auch bei verschachtelten Objekten (wie
Pipeline). Letztere haben Parameter der Form<component>__<parameter>, so dass es möglich ist, jede Komponente eines verschachtelten Objekts zu aktualisieren.- Parameter:
- **paramsdict
Schätzer-Parameter.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
- transform(X)[Quelle]#
Transformieren Sie X in die Dimension der Teilcluster-Zentroiden.
Jede Dimension repräsentiert den Abstand vom Stichprobenpunkt zu jedem Cluster-Zentroiden.
- Parameter:
- X{array-like, sparse matrix} der Form (n_samples, n_features)
Eingabedaten.
- Gibt zurück:
- X_trans{array-like, sparse matrix} of shape (n_samples, n_clusters)
Transformierte Daten.
Galeriebeispiele#
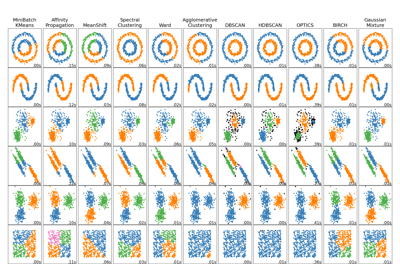
Vergleich verschiedener Clustering-Algorithmen auf Toy-Datensätzen