PCA#
- class sklearn.decomposition.PCA(n_components=None, *, copy=True, whiten=False, svd_solver='auto', tol=0.0, iterated_power='auto', n_oversamples=10, power_iteration_normalizer='auto', random_state=None)[Quelle]#
Hauptkomponentenanalyse (PCA).
Lineare Dimensionsreduktion mittels Singular Value Decomposition (SVD) der Daten, um sie in einen niederdimensionalen Raum zu projizieren. Die Eingabedaten werden vor der Anwendung der SVD für jedes Merkmal zentriert, aber nicht skaliert.
Es wird die LAPACK-Implementierung der vollständigen SVD oder eine randomisierte abgeschnittene SVD nach der Methode von Halko et al. 2009 verwendet, abhängig von der Form der Eingabedaten und der Anzahl der zu extrahierenden Komponenten.
Bei sparsen Eingaben kann die ARPACK-Implementierung der abgeschnittenen SVD verwendet werden (d.h. über
scipy.sparse.linalg.svds). Alternativ kannTruncatedSVDin Betracht gezogen werden, bei der die Daten nicht zentriert werden.Beachten Sie, dass diese Klasse sparsen Eingaben nur für bestimmte Solver wie „arpack“ und „covariance_eigh“ unterstützt. Sehen Sie sich
TruncatedSVDfür eine Alternative mit sparsen Daten an.Ein Anwendungsbeispiel finden Sie unter Hauptkomponentenanalyse (PCA) am Iris-Datensatz
Lesen Sie mehr im Benutzerhandbuch.
- Parameter:
- n_componentsint, float oder ‘mle’, Standard=None
Anzahl der zu behaltenden Komponenten. Wenn n_components nicht gesetzt ist, werden alle Komponenten beibehalten
n_components == min(n_samples, n_features)
Wenn
n_components == 'mle'undsvd_solver == 'full', wird Minka's MLE verwendet, um die Dimension zu erraten. Die Verwendung vonn_components == 'mle'interpretiertsvd_solver == 'auto'alssvd_solver == 'full'.Wenn
0 < n_components < 1undsvd_solver == 'full', wird die Anzahl der Komponenten so gewählt, dass die zu erklärende Varianz den durch n_components angegebenen Prozentsatz überschreitet.Wenn
svd_solver == 'arpack', muss die Anzahl der Komponenten strikt kleiner als das Minimum von n_features und n_samples sein.Daher ergibt der Fall None
n_components == min(n_samples, n_features) - 1
- copybool, Standard=True
Wenn False, werden die an fit übergebenen Daten überschrieben und das Ausführen von fit(X).transform(X) liefert nicht die erwarteten Ergebnisse; verwenden Sie stattdessen fit_transform(X).
- whitenbool, Standard=False
Wenn True (Standard ist False), werden die
components_Vektoren mit der Quadratwurzel von n_samples multipliziert und dann durch die Singulärwerte geteilt, um unkorrelierte Ausgaben mit Einheitsvarianzen pro Komponente zu gewährleisten.Whitening entfernt einige Informationen aus dem transformierten Signal (die relativen Varianzskalen der Komponenten), kann aber manchmal die Vorhersagegenauigkeit der nachgeschalteten Schätzer verbessern, indem es ihre Daten einige hartkodierte Annahmen erfüllen lässt.
- svd_solver{‘auto’, ‘full’, ‘covariance_eigh’, ‘arpack’, ‘randomized’}, Standard=’auto’
- „auto“
Der Solver wird nach einer Standardrichtlinie „auto“ ausgewählt, die auf
X.shapeundn_componentsbasiert: Wenn die Eingabedaten weniger als 1000 Merkmale und mehr als das 10-fache der Stichproben haben, wird der Solver „covariance_eigh“ verwendet. Andernfalls, wenn die Eingabedaten größer als 500x500 sind und die Anzahl der zu extrahierenden Komponenten weniger als 80% der kleinsten Dimension der Daten beträgt, wird die effizientere Methode „randomized“ ausgewählt. Andernfalls wird die exakte „full“ SVD berechnet und optional danach abgeschnitten.- „full“
Führt die exakte vollständige SVD durch Aufruf des Standard-LAPACK-Solvers über
scipy.linalg.svdaus und wählt die Komponenten durch Nachbearbeitung aus.- „covariance_eigh“
Berechnet die Kovarianzmatrix vorab (auf zentrierten Daten), führt eine klassische Eigenwertzerlegung der Kovarianzmatrix typischerweise mit LAPACK durch und wählt die Komponenten durch Nachbearbeitung aus. Dieser Solver ist sehr effizient für n_samples >> n_features und kleine n_features. Er ist jedoch für große n_features anderweitig nicht handhabbar (großer Speicherbedarf zur Materialisierung der Kovarianzmatrix). Beachten Sie auch, dass dieser Solver im Vergleich zum „full“-Solver die Konditionszahl effektiv verdoppelt und daher numerisch weniger stabil ist (z.B. bei Eingabedaten mit einem großen Bereich von Singulärwerten).
- „arpack“
Führt eine SVD, abgeschnitten auf
n_components, aus und ruft den ARPACK-Solver überscipy.sparse.linalg.svdsauf. Er erfordert strikt0 < n_components < min(X.shape)- „randomized“
Führt eine randomisierte SVD nach der Methode von Halko et al. aus.
Hinzugefügt in Version 0.18.0.
Geändert in Version 1.5: Der Solver „covariance_eigh“ wurde hinzugefügt.
- tolfloat, default=0.0
Toleranz für Singulärwerte, berechnet von svd_solver == ‘arpack’. Muss im Bereich [0.0, infinity) liegen.
Hinzugefügt in Version 0.18.0.
- iterated_powerint oder ‘auto’, Standard=’auto’
Anzahl der Iterationen für die Potenzmethode, berechnet von svd_solver == ‘randomized’. Muss im Bereich [0, infinity) liegen.
Hinzugefügt in Version 0.18.0.
- n_oversamplesint, Standard=10
Dieser Parameter ist nur relevant, wenn
svd_solver="randomized". Er entspricht der zusätzlichen Anzahl von Zufallsvektoren zur Abtastung des Bereichs vonX, um eine ordnungsgemäße Konditionierung zu gewährleisten. Weitere Details finden Sie unterrandomized_svd.Hinzugefügt in Version 1.1.
- power_iteration_normalizer{‘auto’, ‘QR’, ‘LU’, ‘none’}, Standard=’auto’
Normalisierung der Potenziteration für den Solver der randomisierten SVD. Wird von ARPACK nicht verwendet. Weitere Details finden Sie unter
randomized_svd.Hinzugefügt in Version 1.1.
- random_stateint, RandomState-Instanz oder None, default=None
Wird verwendet, wenn die Solver ‘arpack’ oder ‘randomized’ benutzt werden. Geben Sie eine Ganzzahl für reproduzierbare Ergebnisse über mehrere Funktionsaufrufe hinweg an. Siehe Glossar.
Hinzugefügt in Version 0.18.0.
- Attribute:
- components_ndarray der Form (n_components, n_features)
Hauptachsen im Merkmalsraum, die die Richtungen maximaler Varianz in den Daten darstellen. Äquivalent dazu sind die rechten Singulärvektoren der zentrierten Eingabedaten, parallel zu ihren Eigenvektoren. Die Komponenten sind nach abnehmender
explained_variance_sortiert.- explained_variance_ndarray der Form (n_components,)
Der von jeder der ausgewählten Komponenten erklärte Varianzbetrag. Die Varianzschätzung verwendet
n_samples - 1Freiheitsgrade.Gleich den n_components größten Eigenwerten der Kovarianzmatrix von X.
Hinzugefügt in Version 0.18.
- explained_variance_ratio_ndarray der Form (n_components,)
Prozentsatz der Varianz, der von jeder der ausgewählten Komponenten erklärt wird.
Wenn
n_componentsnicht gesetzt ist, werden alle Komponenten gespeichert und die Summe der Verhältnisse ist gleich 1.0.- singular_values_ndarray der Form (n_components,)
Die Singulärwerte, die jeder der ausgewählten Komponenten entsprechen. Die Singulärwerte sind gleich den 2-Normen der
n_componentsVariablen im niederdimensionalen Raum.Hinzugefügt in Version 0.19.
- mean_ndarray der Form (n_features,)
Empirischer Mittelwert pro Merkmal, geschätzt aus dem Trainingsdatensatz.
Gleich
X.mean(axis=0).- n_components_int
Die geschätzte Anzahl der Komponenten. Wenn n_components auf ‘mle’ oder eine Zahl zwischen 0 und 1 gesetzt ist (mit svd_solver == ‘full’), wird diese Anzahl aus den Eingabedaten geschätzt. Andernfalls entspricht sie dem Parameter n_components oder dem kleineren Wert von n_features und n_samples, wenn n_components None ist.
- n_samples_int
Anzahl der Stichproben in den Trainingsdaten.
- noise_variance_float
Die geschätzte Rauschkovarianz gemäß dem probabilistischen PCA-Modell von Tipping und Bishop 1999. Siehe „Pattern Recognition and Machine Learning“ von C. Bishop, 12.2.1 S. 574 oder http://www.miketipping.com/papers/met-mppca.pdf. Sie wird zur Berechnung der geschätzten Datenkovarianz und zur Bewertung von Stichproben benötigt.
Gleich dem Durchschnitt der (min(n_features, n_samples) - n_components) kleinsten Eigenwerte der Kovarianzmatrix von X.
- n_features_in_int
Anzahl der während des fits gesehenen Merkmale.
Hinzugefügt in Version 0.24.
- feature_names_in_ndarray mit Form (
n_features_in_,) Namen der während fit gesehenen Merkmale. Nur definiert, wenn
XMerkmalnamen hat, die alle Zeichenketten sind.Hinzugefügt in Version 1.0.
Siehe auch
KernelPCAKernel Principal Component Analysis.
SparsePCASparse Principal Component Analysis.
TruncatedSVDDimensionsreduktion mittels abgeschnittener SVD.
IncrementalPCAInkrementelle Hauptkomponentenanalyse.
Referenzen
Für n_components == ‘mle’ verwendet diese Klasse die Methode von: Minka, T. P.. „Automatic choice of dimensionality for PCA“. In NIPS, S. 598-604
Implementiert das probabilistische PCA-Modell von: Tipping, M. E., und Bishop, C. M. (1999). „Probabilistic principal component analysis“. Journal of the Royal Statistical Society: Series B (Statistical Methodology), 61(3), 611-622. über die Methoden score und score_samples.
Für svd_solver == ‘arpack’ siehe
scipy.sparse.linalg.svds.Für svd_solver == ‘randomized’ siehe: Halko, N., Martinsson, P. G., und Tropp, J. A. (2011). „Finding structure with randomness: Probabilistic algorithms for constructing approximate matrix decompositions“. SIAM review, 53(2), 217-288. und auch Martinsson, P. G., Rokhlin, V., und Tygert, M. (2011). „A randomized algorithm for the decomposition of matrices“. Applied and Computational Harmonic Analysis, 30(1), 47-68.
Beispiele
>>> import numpy as np >>> from sklearn.decomposition import PCA >>> X = np.array([[-1, -1], [-2, -1], [-3, -2], [1, 1], [2, 1], [3, 2]]) >>> pca = PCA(n_components=2) >>> pca.fit(X) PCA(n_components=2) >>> print(pca.explained_variance_ratio_) [0.9924 0.0075] >>> print(pca.singular_values_) [6.30061 0.54980]
>>> pca = PCA(n_components=2, svd_solver='full') >>> pca.fit(X) PCA(n_components=2, svd_solver='full') >>> print(pca.explained_variance_ratio_) [0.9924 0.00755] >>> print(pca.singular_values_) [6.30061 0.54980]
>>> pca = PCA(n_components=1, svd_solver='arpack') >>> pca.fit(X) PCA(n_components=1, svd_solver='arpack') >>> print(pca.explained_variance_ratio_) [0.99244] >>> print(pca.singular_values_) [6.30061]
- fit(X, y=None)[Quelle]#
Passt das Modell an X an.
- Parameter:
- X{array-like, sparse matrix} der Form (n_samples, n_features)
Trainingsdaten, wobei
n_samplesdie Anzahl der Stichproben undn_featuresdie Anzahl der Merkmale ist.- yIgnoriert
Ignoriert.
- Gibt zurück:
- selfobject
Gibt die Instanz selbst zurück.
- fit_transform(X, y=None)[Quelle]#
Passt das Modell an X an und wendet die Dimensionsreduktion auf X an.
- Parameter:
- X{array-like, sparse matrix} der Form (n_samples, n_features)
Trainingsdaten, wobei
n_samplesdie Anzahl der Stichproben undn_featuresdie Anzahl der Merkmale ist.- yIgnoriert
Ignoriert.
- Gibt zurück:
- X_newndarray der Form (n_samples, n_components)
Transformierte Werte.
Anmerkungen
Diese Methode gibt ein Fortran-geordnetes Array zurück. Um es in ein C-geordnetes Array zu konvertieren, verwenden Sie 'np.ascontiguousarray'.
- get_covariance()[Quelle]#
Berechnet die Datenkovarianz mit dem generativen Modell.
cov = components_.T * S**2 * components_ + sigma2 * eye(n_features)wobei S**2 die erklärten Varianzen enthält und sigma2 die Rauschvarianzen.- Gibt zurück:
- covarray der Form (n_features, n_features)
Geschätzte Kovarianz der Daten.
- get_feature_names_out(input_features=None)[Quelle]#
Holt die Ausgabemerkmale für die Transformation.
Die Feature-Namen werden mit dem kleingeschriebenen Klassennamen präfixiert. Wenn der Transformer z.B. 3 Features ausgibt, dann sind die Feature-Namen:
["klassenname0", "klassenname1", "klassenname2"].- Parameter:
- input_featuresarray-like von str oder None, default=None
Wird nur verwendet, um die Feature-Namen mit den in
fitgesehenen Namen zu validieren.
- Gibt zurück:
- feature_names_outndarray von str-Objekten
Transformierte Merkmalnamen.
- get_metadata_routing()[Quelle]#
Holt das Metadaten-Routing dieses Objekts.
Bitte prüfen Sie im Benutzerhandbuch, wie der Routing-Mechanismus funktioniert.
- Gibt zurück:
- routingMetadataRequest
Ein
MetadataRequest, der Routing-Informationen kapselt.
- get_params(deep=True)[Quelle]#
Holt Parameter für diesen Schätzer.
- Parameter:
- deepbool, default=True
Wenn True, werden die Parameter für diesen Schätzer und die enthaltenen Unterobjekte, die Schätzer sind, zurückgegeben.
- Gibt zurück:
- paramsdict
Parameternamen, zugeordnet ihren Werten.
- get_precision()[Quelle]#
Berechnet die Datenpräzisionsmatrix mit dem generativen Modell.
Gleich der Inversen der Kovarianz, aber zur Effizienz mit dem Matrix-Inversions-Lemma berechnet.
- Gibt zurück:
- precisionarray der Form (n_features, n_features)
Geschätzte Präzision der Daten.
- inverse_transform(X)[Quelle]#
Daten zurück in ihren ursprünglichen Raum transformieren.
Mit anderen Worten, gibt eine Eingabe
X_originalzurück, deren Transformation X wäre.- Parameter:
- Xarray-ähnlich von der Form (n_samples, n_components)
Neue Daten, wobei
n_samplesdie Anzahl der Stichproben undn_componentsdie Anzahl der Komponenten ist.
- Gibt zurück:
- X_originalarray-ähnlich der Form (n_samples, n_features)
Ursprüngliche Daten, wobei
n_samplesdie Anzahl der Stichproben undn_featuresdie Anzahl der Merkmale ist.
Anmerkungen
Wenn Whitening aktiviert ist, berechnet inverse_transform die exakte inverse Operation, die die Umkehrung des Whitening einschließt.
- score(X, y=None)[Quelle]#
Gibt die durchschnittliche Log-Likelihood aller Stichproben zurück.
Siehe. „Pattern Recognition and Machine Learning“ von C. Bishop, 12.2.1 S. 574 oder http://www.miketipping.com/papers/met-mppca.pdf
- Parameter:
- Xarray-like der Form (n_samples, n_features)
Die Daten.
- yIgnoriert
Ignoriert.
- Gibt zurück:
- llfloat
Durchschnittliche Log-Likelihood der Stichproben unter dem aktuellen Modell.
- score_samples(X)[Quelle]#
Gibt die Log-Likelihood jeder Stichprobe zurück.
Siehe. „Pattern Recognition and Machine Learning“ von C. Bishop, 12.2.1 S. 574 oder http://www.miketipping.com/papers/met-mppca.pdf
- Parameter:
- Xarray-like der Form (n_samples, n_features)
Die Daten.
- Gibt zurück:
- llndarray der Form (n_samples,)
Log-Likelihood jeder Stichprobe unter dem aktuellen Modell.
- set_output(*, transform=None)[Quelle]#
Ausgabecontainer festlegen.
Siehe Einführung in die set_output API für ein Beispiel zur Verwendung der API.
- Parameter:
- transform{“default”, “pandas”, “polars”}, default=None
Konfiguriert die Ausgabe von
transformundfit_transform."default": Standardausgabeformat eines Transformers"pandas": DataFrame-Ausgabe"polars": Polars-AusgabeNone: Die Transformationskonfiguration bleibt unverändert
Hinzugefügt in Version 1.4: Die Option
"polars"wurde hinzugefügt.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
- set_params(**params)[Quelle]#
Setzt die Parameter dieses Schätzers.
Die Methode funktioniert sowohl bei einfachen Schätzern als auch bei verschachtelten Objekten (wie
Pipeline). Letztere haben Parameter der Form<component>__<parameter>, so dass es möglich ist, jede Komponente eines verschachtelten Objekts zu aktualisieren.- Parameter:
- **paramsdict
Schätzer-Parameter.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
- transform(X)[Quelle]#
Wendet Dimensionsreduktion auf X an.
X wird auf die ersten Hauptkomponenten projiziert, die zuvor aus einem Trainingsdatensatz extrahiert wurden.
- Parameter:
- X{array-like, sparse matrix} der Form (n_samples, n_features)
Neue Daten, wobei
n_samplesdie Anzahl der Stichproben undn_featuresdie Anzahl der Merkmale ist.
- Gibt zurück:
- X_newarray-ähnlich der Form (n_samples, n_components)
Projektion von X in die ersten Hauptkomponenten, wobei
n_samplesdie Anzahl der Stichproben undn_componentsdie Anzahl der Komponenten ist.
Galeriebeispiele#
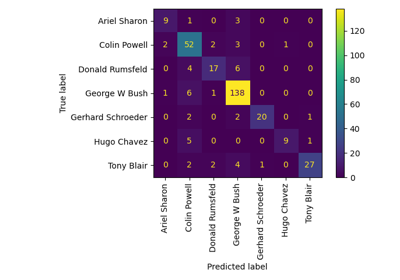
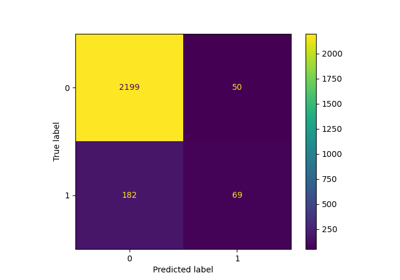
Gesichtserkennungsbeispiel mit Eigenfaces und SVMs
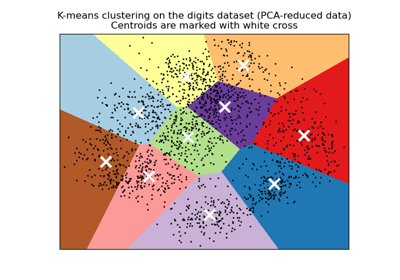
Eine Demo des K-Means Clusterings auf den handschriftlichen Zifferndaten
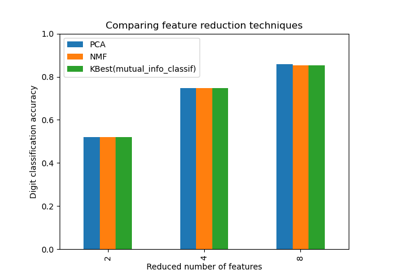
Dimensionsreduktion auswählen mit Pipeline und GridSearchCV
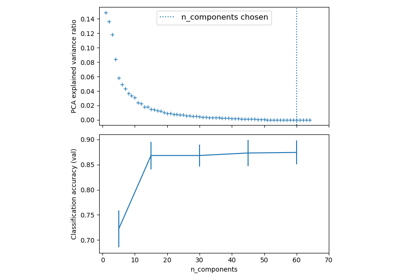
Pipelining: Verkettung einer PCA und einer logistischen Regression
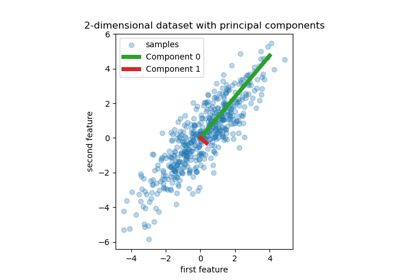
Principal Component Regression vs. Partial Least Squares Regression
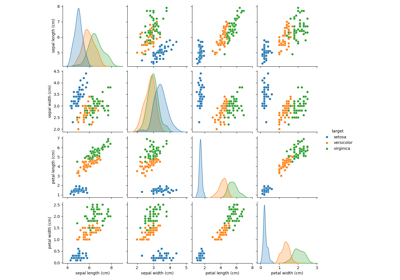
Principal Component Analysis (PCA) auf dem Iris-Datensatz
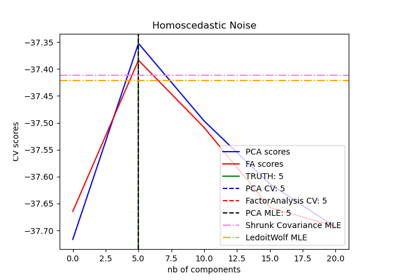
Modellauswahl mit Probabilistischem PCA und Faktorenanalyse (FA)
Vergleich von LDA und PCA 2D-Projektion des Iris-Datensatzes
Faktorenanalyse (mit Rotation) zur Visualisierung von Mustern
Modellkomplexität und kreuzvalidierter Score ausbalancieren
Dimensionsreduktion mit Neighborhood Components Analysis