KFold#
- class sklearn.model_selection.KFold(n_splits=5, *, shuffle=False, random_state=None)[Quelle]#
K-Fold Kreuzvalidierer.
Stellt Trainings-/Testindizes zum Aufteilen von Daten in Trainings-/Testsets bereit. Teilt den Datensatz in k aufeinanderfolgende Folds auf (standardmäßig ohne Mischen).
Jeder Fold wird einmal als Validierungsdatensatz verwendet, während die verbleibenden k - 1 Folds den Trainingsdatensatz bilden.
Lesen Sie mehr im Benutzerhandbuch.
Zur Visualisierung des Kreuzvalidierungsverhaltens und zum Vergleich zwischen gängigen scikit-learn-Split-Methoden siehe Visualisierung des Kreuzvalidierungsverhaltens in scikit-learn
- Parameter:
- n_splitsint, default=5
Anzahl der Folds. Muss mindestens 2 sein.
Geändert in Version 0.22:
n_splitsStandardwert von 3 auf 5 geändert.- shufflebool, default=False
Ob die Daten vor dem Aufteilen in Batches gemischt werden sollen. Beachten Sie, dass die Stichproben innerhalb jedes Splits nicht gemischt werden.
- random_stateint, RandomState-Instanz oder None, default=None
Wenn
shuffleTrue ist, beeinflusstrandom_statedie Reihenfolge der Indizes, die die Zufälligkeit jedes Folds steuert. Andernfalls hat dieser Parameter keine Auswirkung. Übergeben Sie eine Ganzzahl für reproduzierbare Ergebnisse über mehrere Funktionsaufrufe hinweg. Siehe Glossar.
Siehe auch
StratifiedKFoldBerücksichtigt Klasseninformationen, um Folds mit unausgewogenen Klassenverteilungen zu vermeiden (für binäre oder multiklassige Klassifizierungsaufgaben).
GroupKFoldK-Fold Iterator-Variante mit nicht überlappenden Gruppen.
RepeatedKFoldWiederholt K-Fold n Mal.
Anmerkungen
Die ersten
n_samples % n_splitsFolds haben die Größen_samples // n_splits + 1, andere Folds haben die Größen_samples // n_splits, wobein_samplesdie Anzahl der Stichproben ist.Randomisierte CV-Splitter können bei jedem Aufruf von split unterschiedliche Ergebnisse liefern. Sie können die Ergebnisse identisch machen, indem Sie
random_stateauf eine Ganzzahl setzen.Beispiele
>>> import numpy as np >>> from sklearn.model_selection import KFold >>> X = np.array([[1, 2], [3, 4], [1, 2], [3, 4]]) >>> y = np.array([1, 2, 3, 4]) >>> kf = KFold(n_splits=2) >>> kf.get_n_splits() 2 >>> print(kf) KFold(n_splits=2, random_state=None, shuffle=False) >>> for i, (train_index, test_index) in enumerate(kf.split(X)): ... print(f"Fold {i}:") ... print(f" Train: index={train_index}") ... print(f" Test: index={test_index}") Fold 0: Train: index=[2 3] Test: index=[0 1] Fold 1: Train: index=[0 1] Test: index=[2 3]
- get_metadata_routing()[Quelle]#
Holt das Metadaten-Routing dieses Objekts.
Bitte prüfen Sie im Benutzerhandbuch, wie der Routing-Mechanismus funktioniert.
- Gibt zurück:
- routingMetadataRequest
Ein
MetadataRequest, der Routing-Informationen kapselt.
- get_n_splits(X=None, y=None, groups=None)[Quelle]#
Gibt die Anzahl der Splitting-Iterationen zurück, die mit dem Parameter
n_splitsbei der Instanziierung des Kreuzvalidierers festgelegt wurden.- Parameter:
- Xarray-like von Shape (n_samples, n_features), default=None
Immer ignoriert, existiert aus API-Kompatibilitätsgründen.
- yarray-like mit Form (n_samples,), default=None
Immer ignoriert, existiert aus API-Kompatibilitätsgründen.
- groupsarray-like of shape (n_samples,), default=None
Immer ignoriert, existiert aus API-Kompatibilitätsgründen.
- Gibt zurück:
- n_splitsint
Gibt die Anzahl der Teilungsschritte im Kreuzvalidierer zurück.
- split(X, y=None, groups=None)[Quelle]#
Generiert Indizes zum Aufteilen von Daten in Trainings- und Testsets.
- Parameter:
- Xarray-like der Form (n_samples, n_features)
Trainingsdaten, wobei
n_samplesdie Anzahl der Stichproben undn_featuresdie Anzahl der Merkmale ist.- yarray-like mit Form (n_samples,), default=None
Die Zielvariable für überwachte Lernprobleme.
- groupsarray-like of shape (n_samples,), default=None
Immer ignoriert, existiert aus API-Kompatibilitätsgründen.
- Gibt:
- trainndarray
Die Trainingsset-Indizes für diese Teilung.
- testndarray
Die Testset-Indizes für diese Teilung.
Galeriebeispiele#

Vergleich von Random Forests und Histogram Gradient Boosting Modellen
Visualisierung des Kreuzvalidierungsverhaltens in scikit-learn
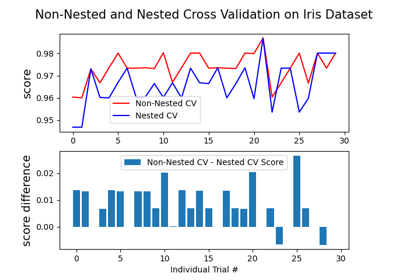
Verschachtelte vs. nicht verschachtelte Kreuzvalidierung