OPTICS#
- class sklearn.cluster.OPTICS(*, min_samples=5, max_eps=inf, metric='minkowski', p=2, metric_params=None, cluster_method='xi', eps=None, xi=0.05, predecessor_correction=True, min_cluster_size=None, algorithm='auto', leaf_size=30, memory=None, n_jobs=None)[Quelle]#
Schätzt die Clustering-Struktur aus einer Vektor-Array.
OPTICS (Ordering Points To Identify the Clustering Structure), eng verwandt mit DBSCAN, findet Kernpunkte hoher Dichte und erweitert Cluster von ihnen aus [1]. Im Gegensatz zu DBSCAN behält es die Clusterhierarchie für einen variablen Nachbarschaftsradius bei. Besser geeignet für die Verwendung auf großen Datensätzen als die aktuelle scikit-learn-Implementierung von DBSCAN.
Cluster werden dann aus der Clusterreihenfolge mittels einer DBSCAN-ähnlichen Methode (cluster_method = ‘dbscan’) oder einer automatischen Technik, die in [1] vorgeschlagen wurde (cluster_method = ‘xi’), extrahiert.
Diese Implementierung weicht vom ursprünglichen OPTICS ab, indem sie zuerst k-nächste-Nachbarschaftssuchen an allen Punkten durchführt, um die Kernpunktgrößen aller Punkte zu identifizieren (anstatt Nachbarn während der Schleife durch Punkte zu berechnen). Dann werden Erreichbarkeitsabstände zu nur unverarbeiteten Punkten berechnet, um die Clusterreihenfolge zu konstruieren, ähnlich dem ursprünglichen OPTICS. Beachten Sie, dass wir keinen Heap zur Verwaltung der Erweiterungskandidaten verwenden, sodass die Zeitkomplexität O(n^2) beträgt.
Lesen Sie mehr im Benutzerhandbuch.
- Parameter:
- min_samplesint > 1 oder float zwischen 0 und 1, default=5
Die Anzahl der Stichproben in einer Nachbarschaft, damit ein Punkt als Kernpunkt betrachtet wird. Außerdem dürfen auf- und absteigende steile Regionen nicht mehr als
min_samplesaufeinanderfolgende nicht-steile Punkte enthalten. Ausgedrückt als absolute Zahl oder als Bruchteil der Anzahl der Stichproben (auf mindestens 2 gerundet).- max_epsfloat, default=np.inf
Der maximale Abstand zwischen zwei Stichproben, damit eine als Nachbar der anderen betrachtet wird. Der Standardwert von
np.infidentifiziert Cluster über alle Skalen hinweg; eine Reduzierung vonmax_epsführt zu kürzeren Laufzeiten.- metricstr oder aufrufbar, Standard=’minkowski’
Metrik, die zur Distanzberechnung verwendet werden soll. Jede Metrik von scikit-learn oder
scipy.spatial.distancekann verwendet werden.Wenn
metriceine aufrufbare Funktion ist, wird sie für jedes Paar von Instanzen (Zeilen) aufgerufen und der resultierende Wert aufgezeichnet. Die aufrufbare Funktion sollte zwei Arrays als Eingabe nehmen und einen Wert zurückgeben, der den Abstand zwischen ihnen angibt. Dies funktioniert für Scipy-Metriken, ist aber weniger effizient als die Angabe der Metrik als Zeichenkette. Wenn metric = „precomputed“ ist, wird angenommen, dassXeine Distanzmatrix ist und quadratisch sein muss.Gültige Werte für metric sind
aus scikit-learn: [‘cityblock’, ‘cosine’, ‘euclidean’, ‘l1’, ‘l2’, ‘manhattan’]
aus scipy.spatial.distance: [‘braycurtis’, ‘canberra’, ‘chebyshev’, ‘correlation’, ‘dice’, ‘hamming’, ‘jaccard’, ‘kulsinski’, ‘mahalanobis’, ‘minkowski’, ‘rogerstanimoto’, ‘russellrao’, ‘seuclidean’, ‘sokalmichener’, ‘sokalsneath’, ‘sqeuclidean’, ‘yule’]
Sparse Matrizen werden nur von scikit-learn-Metriken unterstützt. Einzelheiten zu diesen Metriken finden Sie unter
scipy.spatial.distance.Hinweis
'kulsinski'ist seit SciPy 1.9 veraltet und wird in SciPy 1.11 entfernt.- pfloat, Standard=2
Parameter für die Minkowski-Metrik aus
pairwise_distances. Wenn p = 1, ist dies äquivalent zur Verwendung von manhattan_distance (l1) und euclidean_distance (l2) für p = 2. Für beliebiges p wird minkowski_distance (l_p) verwendet.- metric_paramsdict, Standard=None
Zusätzliche Schlüsselwortargumente für die Metrikfunktion.
- cluster_method{‘xi’, ‘dbscan’}, default=’xi’
Die Extraktionsmethode, die zur Extraktion von Clustern unter Verwendung der berechneten Erreichbarkeit und Ordnung verwendet wird.
- epsfloat, default=None
Der maximale Abstand zwischen zwei Stichproben, damit eine als Nachbar der anderen betrachtet wird. Standardmäßig wird derselbe Wert wie
max_epsangenommen. Wird nur verwendet, wenncluster_method='dbscan'.- xifloat zwischen 0 und 1, default=0.05
Bestimmt die minimale Steilheit im Erreichbarkeitsdiagramm, die eine Clustergrenze darstellt. Zum Beispiel wird ein aufsteigender Punkt im Erreichbarkeitsdiagramm durch das Verhältnis von einem Punkt zu seinem Nachfolger definiert, das höchstens 1-xi beträgt. Wird nur verwendet, wenn
cluster_method='xi'.- predecessor_correctionbool, default=True
Korrigiert Cluster gemäß den von OPTICS berechneten Vorgängern [2]. Dieser Parameter hat nur geringe Auswirkungen auf die meisten Datensätze. Wird nur verwendet, wenn
cluster_method='xi'.- min_cluster_sizeint > 1 oder float zwischen 0 und 1, default=None
Minimale Anzahl von Stichproben in einem OPTICS-Cluster, ausgedrückt als absolute Zahl oder als Bruchteil der Anzahl der Stichproben (auf mindestens 2 gerundet). Wenn
None, wird stattdessen der Wert vonmin_samplesverwendet. Wird nur verwendet, wenncluster_method='xi'.- algorithm{‘auto’, ‘ball_tree’, ‘kd_tree’, ‘brute’}, Standard=’auto’
Algorithmus zur Berechnung der nächsten Nachbarn
„ball_tree“ verwendet
BallTree.„kd_tree“ verwendet
KDTree.‘brute’ verwendet eine brute-force Suche.
„auto“ (Standard) versucht, den am besten geeigneten Algorithmus basierend auf den an die
fit-Methode übergebenen Werten zu bestimmen.
Hinweis: Das Anpassen an dünnbesetzte Eingaben überschreibt die Einstellung dieses Parameters und verwendet brute force.
- leaf_sizeint, Standard=30
Leaf-Größe, die an
BallTreeoderKDTreeübergeben wird. Dies kann die Geschwindigkeit des Aufbaus und der Abfrage sowie den Speicherbedarf für den Baum beeinflussen. Der optimale Wert hängt von der Art des Problems ab.- memoryString oder Objekt mit dem joblib.Memory-Interface, Standard=None
Wird verwendet, um die Ausgabe der Berechnung des Baumes zu cachen. Standardmäßig erfolgt kein Caching. Wenn ein String angegeben wird, ist dies der Pfad zum Cache-Verzeichnis.
- n_jobsint, default=None
Die Anzahl der parallelen Jobs für die Nachbarsuche.
Nonebedeutet 1, es sei denn, es befindet sich in einemjoblib.parallel_backendKontext.-1bedeutet, alle Prozessoren zu verwenden. Siehe Glossar für weitere Details.
- Attribute:
- labels_ndarray der Form (n_samples,)
Cluster-Labels für jeden Punkt im Datensatz, der an fit() übergeben wurde. Rauschhafte Stichproben und Punkte, die nicht in einem Blattcluster von
cluster_hierarchy_enthalten sind, werden als -1 markiert.- reachability_ndarray der Form (n_samples,)
Erreichbarkeitsabstände pro Stichprobe, indiziert nach Objektordnung. Verwenden Sie
clust.reachability_[clust.ordering_], um im Cluster-Auftrag zuzugreifen.- ordering_ndarray der Form (n_samples,)
Die nach Clustern geordnete Liste der Stichprobenindizes.
- core_distances_ndarray der Form (n_samples,)
Abstand, bei dem jede Stichprobe zu einem Kernpunkt wird, indiziert nach Objektordnung. Punkte, die niemals Kernpunkte werden, haben einen Abstand von inf. Verwenden Sie
clust.core_distances_[clust.ordering_], um im Cluster-Auftrag zuzugreifen.- predecessor_ndarray der Form (n_samples,)
Punkt, von dem eine Stichprobe erreicht wurde, indiziert nach Objektordnung. Startpunkte haben einen Vorgänger von -1.
- cluster_hierarchy_ndarray der Form (n_clusters, 2)
Die Liste der Cluster in der Form
[start, end]in jeder Zeile, wobei alle Indizes inklusive sind. Die Cluster sind nach(end, -start)(aufsteigend) geordnet, sodass größere Cluster, die kleinere umfassen, nach diesen kleineren kommen. Dalabels_die Hierarchie nicht widerspiegelt, ist normalerweiselen(cluster_hierarchy_) > np.unique(optics.labels_). Bitte beachten Sie auch, dass diese Indizes sich aufordering_beziehen, d.h.X[ordering_][start:end + 1]bilden einen Cluster. Nur verfügbar, wenncluster_method='xi'.- n_features_in_int
Anzahl der während des fits gesehenen Merkmale.
Hinzugefügt in Version 0.24.
- feature_names_in_ndarray mit Form (
n_features_in_,) Namen der während fit gesehenen Merkmale. Nur definiert, wenn
XMerkmalnamen hat, die alle Zeichenketten sind.Hinzugefügt in Version 1.0.
Siehe auch
DBSCANEine ähnliche Clusterbildung für einen bestimmten Nachbarschaftsradius (eps). Unsere Implementierung ist auf Laufzeit optimiert.
Referenzen
[1] (1,2)Ankerst, Mihael, Markus M. Breunig, Hans-Peter Kriegel und Jörg Sander. „OPTICS: ordering points to identify the clustering structure.“ ACM SIGMOD Record 28, Nr. 2 (1999): 49-60.
[2]Schubert, Erich, Michael Gertz. „Improving the Cluster Structure Extracted from OPTICS Plots.“ Proc. of the Conference “Lernen, Wissen, Daten, Analysen” (LWDA) (2018): 318-329.
Beispiele
>>> from sklearn.cluster import OPTICS >>> import numpy as np >>> X = np.array([[1, 2], [2, 5], [3, 6], ... [8, 7], [8, 8], [7, 3]]) >>> clustering = OPTICS(min_samples=2).fit(X) >>> clustering.labels_ array([0, 0, 0, 1, 1, 1])
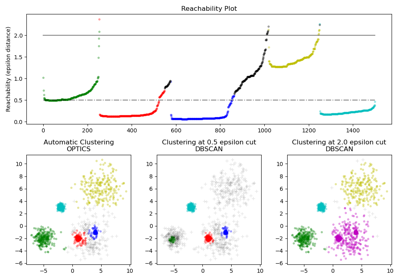
Ein detaillierteres Beispiel finden Sie in Demo des OPTICS-Clustering-Algorithmus.
Ein Vergleich von OPTICS mit anderen Clustering-Algorithmen finden Sie in Vergleich verschiedener Clustering-Algorithmen auf Beispiel-Datensätzen
- fit(X, y=None)[Quelle]#
Führt OPTICS-Clustering durch.
Extrahiert eine geordnete Liste von Punkten und Erreichbarkeitsabstände und führt eine anfängliche Clusterbildung mit einem Abstand von
max_epsdurch, der bei der Instanziierung des OPTICS-Objekts angegeben wurde.- Parameter:
- X{ndarray, sparse matrix} der Form (n_samples, n_features), oder (n_samples, n_samples), wenn metric=’precomputed’
Ein Merkmalsarray oder ein Array von Abständen zwischen Stichproben, wenn metric=’precomputed’ ist. Wenn eine Sparse-Matrix angegeben wird, wird sie in das CSR-Format konvertiert.
- yIgnoriert
Wird nicht verwendet, ist aber aus Gründen der API-Konsistenz per Konvention vorhanden.
- Gibt zurück:
- selfobject
Gibt eine angepasste Instanz von self zurück.
- fit_predict(X, y=None, **kwargs)[Quelle]#
Führt Clustering auf
Xdurch und gibt Cluster-Labels zurück.- Parameter:
- Xarray-like der Form (n_samples, n_features)
Eingabedaten.
- yIgnoriert
Wird nicht verwendet, ist aber aus Gründen der API-Konsistenz per Konvention vorhanden.
- **kwargsdict
Argumente, die an
fitübergeben werden sollen.Hinzugefügt in Version 1.4.
- Gibt zurück:
- labelsndarray der Form (n_samples,), dtype=np.int64
Clusterbeschriftungen.
- get_metadata_routing()[Quelle]#
Holt das Metadaten-Routing dieses Objekts.
Bitte prüfen Sie im Benutzerhandbuch, wie der Routing-Mechanismus funktioniert.
- Gibt zurück:
- routingMetadataRequest
Ein
MetadataRequest, der Routing-Informationen kapselt.
- get_params(deep=True)[Quelle]#
Holt Parameter für diesen Schätzer.
- Parameter:
- deepbool, default=True
Wenn True, werden die Parameter für diesen Schätzer und die enthaltenen Unterobjekte, die Schätzer sind, zurückgegeben.
- Gibt zurück:
- paramsdict
Parameternamen, zugeordnet ihren Werten.
- set_params(**params)[Quelle]#
Setzt die Parameter dieses Schätzers.
Die Methode funktioniert sowohl bei einfachen Schätzern als auch bei verschachtelten Objekten (wie
Pipeline). Letztere haben Parameter der Form<component>__<parameter>, so dass es möglich ist, jede Komponente eines verschachtelten Objekts zu aktualisieren.- Parameter:
- **paramsdict
Schätzer-Parameter.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
Galeriebeispiele#
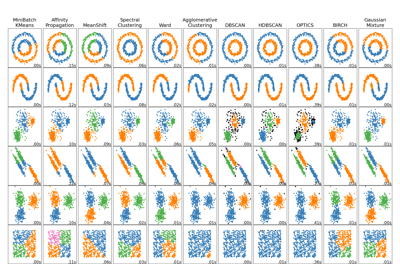
Vergleich verschiedener Clustering-Algorithmen auf Toy-Datensätzen