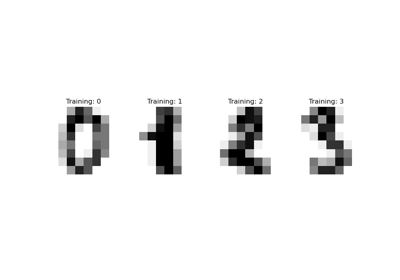train_test_split#
- sklearn.model_selection.train_test_split(*arrays, test_size=None, train_size=None, random_state=None, shuffle=True, stratify=None)[Quelle]#
Teilt Arrays oder Matrizen in zufällige Trainings- und Testteilmengen auf.
Schnelle Hilfsfunktion, die die Eingabevalidierung,
next(ShuffleSplit().split(X, y))und die Anwendung auf Eingabedaten in einem einzigen Aufruf für das Aufteilen (und optional Subsampling) von Daten in einer Einzeiligen kombiniert.Lesen Sie mehr im Benutzerhandbuch.
- Parameter:
- *arraysSequenz von indexierbaren Objekten mit gleicher Länge / Form[0]
Zulässige Eingaben sind Listen, Numpy-Arrays, SciPy-Sparse-Matrizen oder Pandas-DataFrames.
- test_sizefloat oder int, Standardwert=None
Wenn float, sollte zwischen 0,0 und 1,0 liegen und den Anteil des Datensatzes darstellen, der in den Test-Split einbezogen werden soll. Wenn int, stellt die absolute Anzahl der Teststichproben dar. Wenn None, wird der Wert auf das Komplement der Trainingsgröße gesetzt. Wenn
train_sizeebenfalls None ist, wird er auf 0,25 gesetzt.- train_sizefloat oder int, Standardwert=None
Wenn float, sollte zwischen 0.0 und 1.0 liegen und den Anteil des Datensatzes darstellen, der in die Trainingsaufteilung aufgenommen werden soll. Wenn int, repräsentiert die absolute Anzahl der Trainingsstichproben. Wenn None, wird der Wert automatisch auf das Komplement der Testgröße gesetzt.
- random_stateint, RandomState-Instanz oder None, default=None
Steuert das Mischen, das vor der Anwendung des Splits auf die Daten angewendet wird. Geben Sie einen int für reproduzierbare Ausgaben über mehrere Funktionsaufrufe hinweg an. Siehe Glossar.
- shufflebool, Standard=True
Ob die Daten vor dem Aufteilen gemischt werden sollen oder nicht. Wenn shuffle=False, muss stratify None sein.
- stratifyarray-artig, Standard=None
Wenn nicht None, werden die Daten stratifiziert aufgeteilt, wobei dies als Klassenbezeichnungen verwendet wird. Lesen Sie mehr im Benutzerhandbuch.
- Gibt zurück:
- splittingListe, Länge=2 * len(arrays)
Liste, die den Trainings-Test-Split der Eingaben enthält.
Hinzugefügt in Version 0.16: Wenn die Eingabe spärlich ist, ist die Ausgabe eine
scipy.sparse.csr_matrix. Andernfalls ist der Ausgabetyp derselbe wie der Eingabetyp.
Beispiele
>>> import numpy as np >>> from sklearn.model_selection import train_test_split >>> X, y = np.arange(10).reshape((5, 2)), range(5) >>> X array([[0, 1], [2, 3], [4, 5], [6, 7], [8, 9]]) >>> list(y) [0, 1, 2, 3, 4]
>>> X_train, X_test, y_train, y_test = train_test_split( ... X, y, test_size=0.33, random_state=42) ... >>> X_train array([[4, 5], [0, 1], [6, 7]]) >>> y_train [2, 0, 3] >>> X_test array([[2, 3], [8, 9]]) >>> y_test [1, 4]
>>> train_test_split(y, shuffle=False) [[0, 1, 2], [3, 4]]
>>> from sklearn import datasets >>> iris = datasets.load_iris(as_frame=True) >>> X, y = iris['data'], iris['target'] >>> X.head() sepal length (cm) sepal width (cm) petal length (cm) petal width (cm) 0 5.1 3.5 1.4 0.2 1 4.9 3.0 1.4 0.2 2 4.7 3.2 1.3 0.2 3 4.6 3.1 1.5 0.2 4 5.0 3.6 1.4 0.2 >>> y.head() 0 0 1 0 2 0 3 0 4 0 ...
>>> X_train, X_test, y_train, y_test = train_test_split( ... X, y, test_size=0.33, random_state=42) ... >>> X_train.head() sepal length (cm) sepal width (cm) petal length (cm) petal width (cm) 96 5.7 2.9 4.2 1.3 105 7.6 3.0 6.6 2.1 66 5.6 3.0 4.5 1.5 0 5.1 3.5 1.4 0.2 122 7.7 2.8 6.7 2.0 >>> y_train.head() 96 1 105 2 66 1 0 0 122 2 ... >>> X_test.head() sepal length (cm) sepal width (cm) petal length (cm) petal width (cm) 73 6.1 2.8 4.7 1.2 18 5.7 3.8 1.7 0.3 118 7.7 2.6 6.9 2.3 78 6.0 2.9 4.5 1.5 76 6.8 2.8 4.8 1.4 >>> y_test.head() 73 1 18 0 118 2 78 1 76 1 ...
Galeriebeispiele#
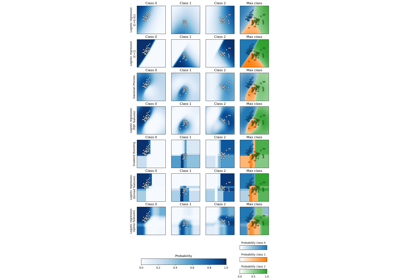
Gesichtserkennungsbeispiel mit Eigenfaces und SVMs
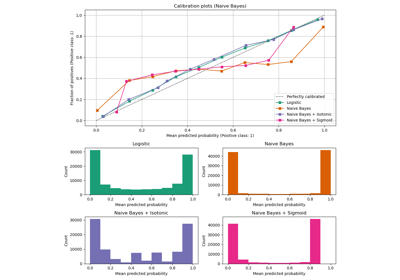
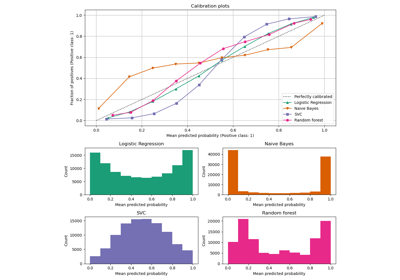
Wahrscheinlichkeitskalibrierung von Klassifikatoren
Auswirkung der Transformation der Ziele in einem Regressionsmodell
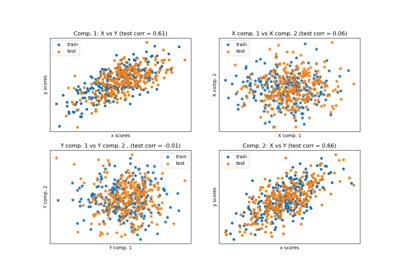
Principal Component Regression vs. Partial Least Squares Regression
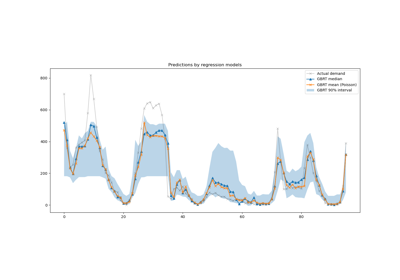
Vorhersageintervalle für Gradient Boosting Regression
Vergleich von Random Forests und dem Multi-Output Meta-Estimator
Versagen des maschinellen Lernens bei der Inferenz kausaler Effekte
Häufige Fallstricke bei der Interpretation von Koeffizienten linearer Modelle

Permutations-Wichtigkeit vs. Random Forest Merkmals-Wichtigkeit (MDI)
Permutations-Wichtigkeit bei multikollinearen oder korrelierten Merkmalen
Skalierbares Lernen mit Polynom-Kernel-Approximation
Gewöhnliche kleinste Quadrate und Ridge Regression

Poisson-Regression und nicht-normale Verlustfunktion
Multiklassen-Sparse-Logistische-Regression auf 20newgroups
MNIST-Klassifikation mittels multinomialer Logistik + L1
Leistung eines Klassifikators mit Konfusionsmatrix bewerten
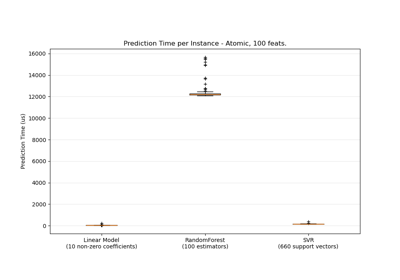
Post-Hoc-Anpassung des Entscheidungsschwellenwerts für kostenempfindliches Lernen

Benutzerdefinierte Refit-Strategie einer Gitter-Suche mit Kreuzvalidierung
Klassen-Likelihood-Verhältnisse zur Messung der Klassifikationsleistung
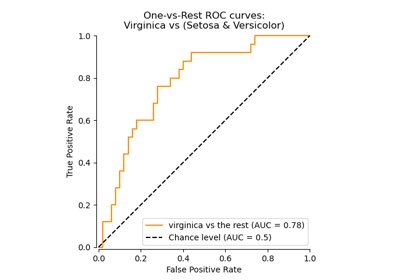
Multiklassen-Receiver Operating Characteristic (ROC)
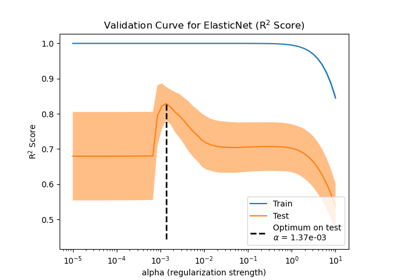
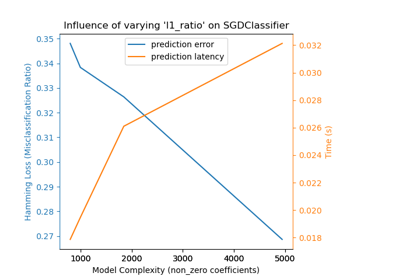
Auswirkung der Modellregularisierung auf Trainings- und Testfehler
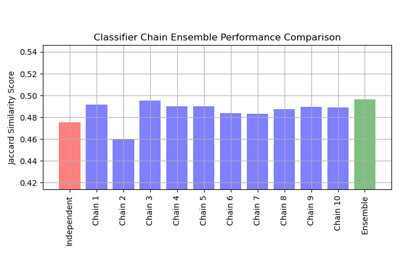
Multilabel-Klassifikation mit einem Klassifikator-Ketten
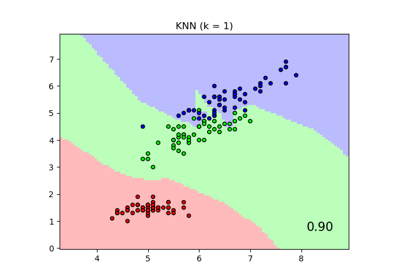
Vergleich von Nächsten Nachbarn mit und ohne Neighborhood Components Analysis
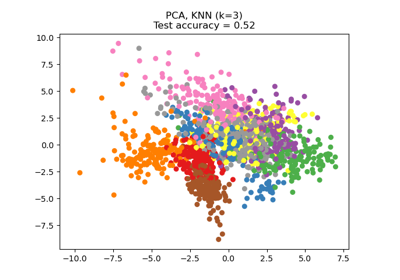
Dimensionsreduktion mit Neighborhood Components Analysis
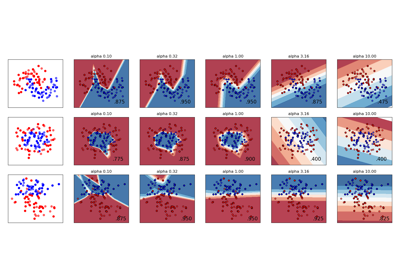
Variierende Regularisierung im Multi-Layer Perceptron

Restricted Boltzmann Machine Merkmale für Ziffernklassifikation
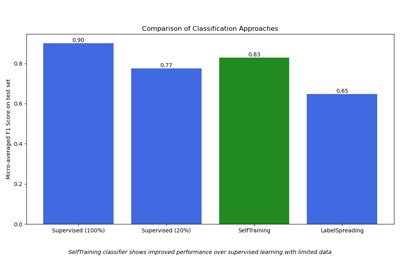
Semi-überwachte Klassifikation auf einem Textdatensatz
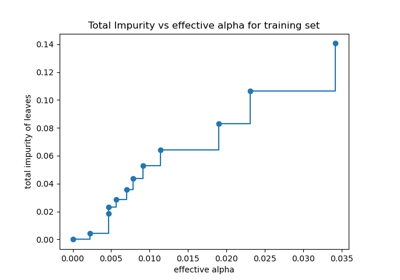
Post-Pruning Entscheidungsbäume mit Kostenkomplexität