Hinweis
Zum Ende springen, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Merkmale-Transformationen mit Baum-Ensembles#
Transformieren Sie Ihre Merkmale in einen höherdimensionalen, spärlichen Raum. Trainieren Sie dann ein lineares Modell auf diesen Merkmalen.
Zuerst wird ein Ensemble von Bäumen (vollständig zufällige Bäume, ein Random Forest oder Gradient Boosting Bäume) auf dem Trainingsdatensatz angepasst. Dann wird jedem Blatt jedes Baumes im Ensemble ein fester, willkürlicher Merkmalsindex in einem neuen Merkmalsraum zugewiesen. Diese Blattindizes werden dann im One-Hot-Verfahren kodiert.
Jede Stichprobe durchläuft die Entscheidungen jedes Baumes des Ensembles und landet in einem Blatt pro Baum. Die Stichprobe wird kodiert, indem die Merkmalswerte für diese Blätter auf 1 und die anderen Merkmalswerte auf 0 gesetzt werden.
Der resultierende Transformer hat dann eine überwachte, spärliche, hochdimensionale kategoriale Einbettung der Daten gelernt.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Zuerst erstellen wir einen großen Datensatz und teilen ihn in drei Sätze auf
ein Satz zum Trainieren der Ensemble-Methoden, die später als Merkmal-Engineering-Transformer verwendet werden;
ein Satz zum Trainieren des linearen Modells;
ein Satz zum Testen des linearen Modells.
Es ist wichtig, die Daten so aufzuteilen, um Überanpassung durch Datenlecks zu vermeiden.
from sklearn.datasets import make_classification
from sklearn.model_selection import train_test_split
X, y = make_classification(n_samples=80_000, random_state=10)
X_full_train, X_test, y_full_train, y_test = train_test_split(
X, y, test_size=0.5, random_state=10
)
X_train_ensemble, X_train_linear, y_train_ensemble, y_train_linear = train_test_split(
X_full_train, y_full_train, test_size=0.5, random_state=10
)
Für jede der Ensemble-Methoden verwenden wir 10 Estimators und eine maximale Tiefe von 3 Ebenen.
n_estimators = 10
max_depth = 3
Zuerst beginnen wir mit dem Trainieren des Random Forest und des Gradient Boosting auf dem getrennten Trainingsdatensatz
from sklearn.ensemble import GradientBoostingClassifier, RandomForestClassifier
random_forest = RandomForestClassifier(
n_estimators=n_estimators, max_depth=max_depth, random_state=10
)
random_forest.fit(X_train_ensemble, y_train_ensemble)
gradient_boosting = GradientBoostingClassifier(
n_estimators=n_estimators, max_depth=max_depth, random_state=10
)
_ = gradient_boosting.fit(X_train_ensemble, y_train_ensemble)
Beachten Sie, dass HistGradientBoostingClassifier viel schneller ist als GradientBoostingClassifier ab mittleren Datensätzen (n_samples >= 10_000), was bei diesem Beispiel nicht der Fall ist.
Die RandomTreesEmbedding ist eine unbeaufsichtigte Methode und muss daher nicht unabhängig trainiert werden.
from sklearn.ensemble import RandomTreesEmbedding
random_tree_embedding = RandomTreesEmbedding(
n_estimators=n_estimators, max_depth=max_depth, random_state=0
)
Nun erstellen wir drei Pipelines, die die obige Einbettung als Vorverarbeitungsstufe verwenden.
Die Random Trees Einbettung kann direkt mit der logistischen Regression in eine Pipeline integriert werden, da es sich um einen Standard-Scikit-learn-Transformer handelt.
from sklearn.linear_model import LogisticRegression
from sklearn.pipeline import make_pipeline
rt_model = make_pipeline(random_tree_embedding, LogisticRegression(max_iter=1000))
rt_model.fit(X_train_linear, y_train_linear)
Pipeline(steps=[('randomtreesembedding',
RandomTreesEmbedding(max_depth=3, n_estimators=10,
random_state=0)),
('logisticregression', LogisticRegression(max_iter=1000))])In einer Jupyter-Umgebung führen Sie diese Zelle bitte erneut aus, um die HTML-Darstellung anzuzeigen, oder vertrauen Sie dem Notebook.Auf GitHub kann die HTML-Darstellung nicht gerendert werden. Versuchen Sie bitte, diese Seite mit nbviewer.org zu laden.
Parameter
Parameter
Parameter
Dann können wir einen Random Forest oder Gradient Boosting mit einer logistischen Regression pipelinen. Die Merkmals-Transformation findet jedoch durch Aufruf der Methode apply statt. Die Pipeline in Scikit-learn erwartet einen Aufruf von transform. Daher haben wir den Aufruf von apply in einen FunctionTransformer eingepackt.
from sklearn.preprocessing import FunctionTransformer, OneHotEncoder
def rf_apply(X, model):
return model.apply(X)
rf_leaves_yielder = FunctionTransformer(rf_apply, kw_args={"model": random_forest})
rf_model = make_pipeline(
rf_leaves_yielder,
OneHotEncoder(handle_unknown="ignore"),
LogisticRegression(max_iter=1000),
)
rf_model.fit(X_train_linear, y_train_linear)
Pipeline(steps=[('functiontransformer',
FunctionTransformer(func=<function rf_apply at 0x7fb4864dfce0>,
kw_args={'model': RandomForestClassifier(max_depth=3,
n_estimators=10,
random_state=10)})),
('onehotencoder', OneHotEncoder(handle_unknown='ignore')),
('logisticregression', LogisticRegression(max_iter=1000))])In einer Jupyter-Umgebung führen Sie diese Zelle bitte erneut aus, um die HTML-Darstellung anzuzeigen, oder vertrauen Sie dem Notebook.Auf GitHub kann die HTML-Darstellung nicht gerendert werden. Versuchen Sie bitte, diese Seite mit nbviewer.org zu laden.
Parameter
Parameter
Parameter
Parameter
def gbdt_apply(X, model):
return model.apply(X)[:, :, 0]
gbdt_leaves_yielder = FunctionTransformer(
gbdt_apply, kw_args={"model": gradient_boosting}
)
gbdt_model = make_pipeline(
gbdt_leaves_yielder,
OneHotEncoder(handle_unknown="ignore"),
LogisticRegression(max_iter=1000),
)
gbdt_model.fit(X_train_linear, y_train_linear)
Pipeline(steps=[('functiontransformer',
FunctionTransformer(func=<function gbdt_apply at 0x7fb4864dd800>,
kw_args={'model': GradientBoostingClassifier(n_estimators=10,
random_state=10)})),
('onehotencoder', OneHotEncoder(handle_unknown='ignore')),
('logisticregression', LogisticRegression(max_iter=1000))])In einer Jupyter-Umgebung führen Sie diese Zelle bitte erneut aus, um die HTML-Darstellung anzuzeigen, oder vertrauen Sie dem Notebook.Auf GitHub kann die HTML-Darstellung nicht gerendert werden. Versuchen Sie bitte, diese Seite mit nbviewer.org zu laden.
Parameter
Parameter
Parameter
Parameter
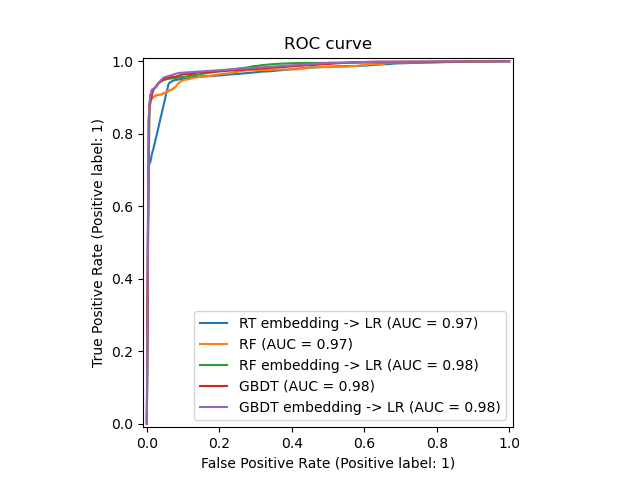
Wir können abschließend die verschiedenen ROC-Kurven für alle Modelle anzeigen.
import matplotlib.pyplot as plt
from sklearn.metrics import RocCurveDisplay
_, ax = plt.subplots()
models = [
("RT embedding -> LR", rt_model),
("RF", random_forest),
("RF embedding -> LR", rf_model),
("GBDT", gradient_boosting),
("GBDT embedding -> LR", gbdt_model),
]
model_displays = {}
for name, pipeline in models:
model_displays[name] = RocCurveDisplay.from_estimator(
pipeline, X_test, y_test, ax=ax, name=name
)
_ = ax.set_title("ROC curve")
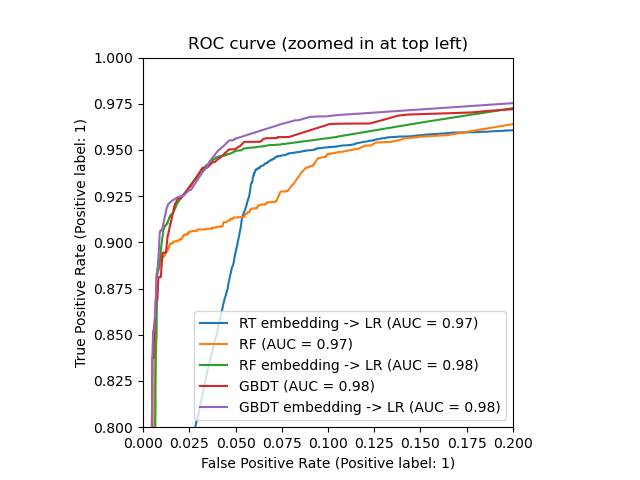
_, ax = plt.subplots()
for name, pipeline in models:
model_displays[name].plot(ax=ax)
ax.set_xlim(0, 0.2)
ax.set_ylim(0.8, 1)
_ = ax.set_title("ROC curve (zoomed in at top left)")
Gesamtlaufzeit des Skripts: (0 Minuten 2,026 Sekunden)
Verwandte Beispiele

Manifold Learning auf handschriftlichen Ziffern: Locally Linear Embedding, Isomap…

Vergleich von Random Forests und Histogram Gradient Boosting Modellen