Hinweis
Gehen Sie zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Lasso-Modellauswahl mittels Informationskriterien#
Dieses Beispiel reproduziert das Beispiel von Abb. 2 in [ZHT2007]. Ein LassoLarsIC-Schätzer wird auf einem Diabetes-Datensatz angepasst, und die Kriterien AIC und BIC werden zur Auswahl des besten Modells verwendet.
Hinweis
Es ist wichtig zu beachten, dass die Optimierung zur Ermittlung von alpha mit LassoLarsIC auf den im Trainingsdatensatz direkt berechneten AIC- oder BIC-Kriterien basiert. Dieser Ansatz unterscheidet sich vom Kreuzvalidierungsverfahren. Ein Vergleich der beiden Ansätze ist im folgenden Beispiel zu finden: Lasso-Modellauswahl: AIC-BIC / Kreuzvalidierung.
Referenzen
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Wir verwenden den Diabetes-Datensatz.
from sklearn.datasets import load_diabetes
X, y = load_diabetes(return_X_y=True, as_frame=True)
n_samples = X.shape[0]
X.head()
Scikit-learn bietet einen Schätzer namens LassoLarsIC, der entweder das Akaike-Informationskriterium (AIC) oder das Bayes'sche Informationskriterium (BIC) zur Auswahl des besten Modells verwendet. Bevor wir dieses Modell anpassen, skalieren wir den Datensatz.
Im Folgenden passen wir zwei Modelle an, um die von AIC und BIC gemeldeten Werte zu vergleichen.
from sklearn.linear_model import LassoLarsIC
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import StandardScaler
lasso_lars_ic = make_pipeline(StandardScaler(), LassoLarsIC(criterion="aic")).fit(X, y)
Um der Definition in [ZHT2007] zu entsprechen, müssen wir AIC und BIC neu skalieren. Zou et al. ignorieren nämlich einige konstante Terme im Vergleich zur ursprünglichen Definition von AIC, die sich aus der maximalen Log-Likelihood eines linearen Modells ergibt. Sie können auf den Abschnitt mathematische Details im Benutzerhandbuch verweisen.
import numpy as np
aic_criterion = zou_et_al_criterion_rescaling(
lasso_lars_ic[-1].criterion_,
n_samples,
lasso_lars_ic[-1].noise_variance_,
)
index_alpha_path_aic = np.flatnonzero(
lasso_lars_ic[-1].alphas_ == lasso_lars_ic[-1].alpha_
)[0]
lasso_lars_ic.set_params(lassolarsic__criterion="bic").fit(X, y)
bic_criterion = zou_et_al_criterion_rescaling(
lasso_lars_ic[-1].criterion_,
n_samples,
lasso_lars_ic[-1].noise_variance_,
)
index_alpha_path_bic = np.flatnonzero(
lasso_lars_ic[-1].alphas_ == lasso_lars_ic[-1].alpha_
)[0]
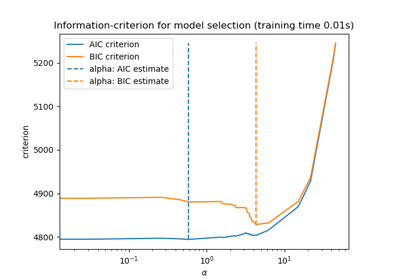
Nachdem wir die AIC- und BIC-Werte gesammelt haben, können wir auch überprüfen, ob die Minima beider Kriterien bei demselben Alpha auftreten. Dann können wir die folgende Abbildung vereinfachen.
index_alpha_path_aic == index_alpha_path_bic
np.True_
Schließlich können wir das AIC- und BIC-Kriterium sowie den darauf basierenden ausgewählten Regularisierungsparameter darstellen.
import matplotlib.pyplot as plt
plt.plot(aic_criterion, color="tab:blue", marker="o", label="AIC criterion")
plt.plot(bic_criterion, color="tab:orange", marker="o", label="BIC criterion")
plt.vlines(
index_alpha_path_bic,
aic_criterion.min(),
aic_criterion.max(),
color="black",
linestyle="--",
label="Selected alpha",
)
plt.legend()
plt.ylabel("Information criterion")
plt.xlabel("Lasso model sequence")
_ = plt.title("Lasso model selection via AIC and BIC")

Gesamtlaufzeit des Skripts: (0 Minuten 0,080 Sekunden)
Verwandte Beispiele