Hinweis
Zum Ende springen, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Illustration von Gaußschem Prozess vor und nach der Inferenz für verschiedene Kerne#
Dieses Beispiel illustriert die a priori und a posteriori Verteilung eines GaussianProcessRegressor mit verschiedenen Kernen. Mittelwert, Standardabweichung und 5 Stichproben werden für die a priori und a posteriori Verteilungen gezeigt.
Hier geben wir nur einige Illustrationen. Um mehr über die Formulierung von Kernen zu erfahren, siehe das Benutzerhandbuch.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Hilfsfunktion#
Bevor wir jeden einzelnen für Gaußsche Prozesse verfügbaren Kern vorstellen, definieren wir eine Hilfsfunktion, die es uns ermöglicht, aus dem Gaußschen Prozess gezogene Stichproben zu plotten.
Diese Funktion nimmt ein GaussianProcessRegressor-Modell und zieht Stichproben aus dem Gaußschen Prozess. Wenn das Modell nicht trainiert wurde, werden die Stichproben aus der a priori Verteilung gezogen, während nach dem Trainieren des Modells die Stichproben aus der a posteriori Verteilung gezogen werden.
import matplotlib.pyplot as plt
import numpy as np
def plot_gpr_samples(gpr_model, n_samples, ax):
"""Plot samples drawn from the Gaussian process model.
If the Gaussian process model is not trained then the drawn samples are
drawn from the prior distribution. Otherwise, the samples are drawn from
the posterior distribution. Be aware that a sample here corresponds to a
function.
Parameters
----------
gpr_model : `GaussianProcessRegressor`
A :class:`~sklearn.gaussian_process.GaussianProcessRegressor` model.
n_samples : int
The number of samples to draw from the Gaussian process distribution.
ax : matplotlib axis
The matplotlib axis where to plot the samples.
"""
x = np.linspace(0, 5, 100)
X = x.reshape(-1, 1)
y_mean, y_std = gpr_model.predict(X, return_std=True)
y_samples = gpr_model.sample_y(X, n_samples)
for idx, single_prior in enumerate(y_samples.T):
ax.plot(
x,
single_prior,
linestyle="--",
alpha=0.7,
label=f"Sampled function #{idx + 1}",
)
ax.plot(x, y_mean, color="black", label="Mean")
ax.fill_between(
x,
y_mean - y_std,
y_mean + y_std,
alpha=0.1,
color="black",
label=r"$\pm$ 1 std. dev.",
)
ax.set_xlabel("x")
ax.set_ylabel("y")
ax.set_ylim([-3, 3])
Datensatz und Erzeugung des Gaußschen Prozesses#
Wir erstellen einen Trainingsdatensatz, den wir in den verschiedenen Abschnitten verwenden werden.
rng = np.random.RandomState(4)
X_train = rng.uniform(0, 5, 10).reshape(-1, 1)
y_train = np.sin((X_train[:, 0] - 2.5) ** 2)
n_samples = 5
Kernel-Kochbuch#
In diesem Abschnitt illustrieren wir einige Stichproben, die aus den a priori und a posteriori Verteilungen des Gaußschen Prozesses mit verschiedenen Kernen gezogen wurden.
Radial Basis Function Kernel#
from sklearn.gaussian_process import GaussianProcessRegressor
from sklearn.gaussian_process.kernels import RBF
kernel = 1.0 * RBF(length_scale=1.0, length_scale_bounds=(1e-1, 10.0))
gpr = GaussianProcessRegressor(kernel=kernel, random_state=0)
fig, axs = plt.subplots(nrows=2, sharex=True, sharey=True, figsize=(10, 8))
# plot prior
plot_gpr_samples(gpr, n_samples=n_samples, ax=axs[0])
axs[0].set_title("Samples from prior distribution")
# plot posterior
gpr.fit(X_train, y_train)
plot_gpr_samples(gpr, n_samples=n_samples, ax=axs[1])
axs[1].scatter(X_train[:, 0], y_train, color="red", zorder=10, label="Observations")
axs[1].legend(bbox_to_anchor=(1.05, 1.5), loc="upper left")
axs[1].set_title("Samples from posterior distribution")
fig.suptitle("Radial Basis Function kernel", fontsize=18)
plt.tight_layout()
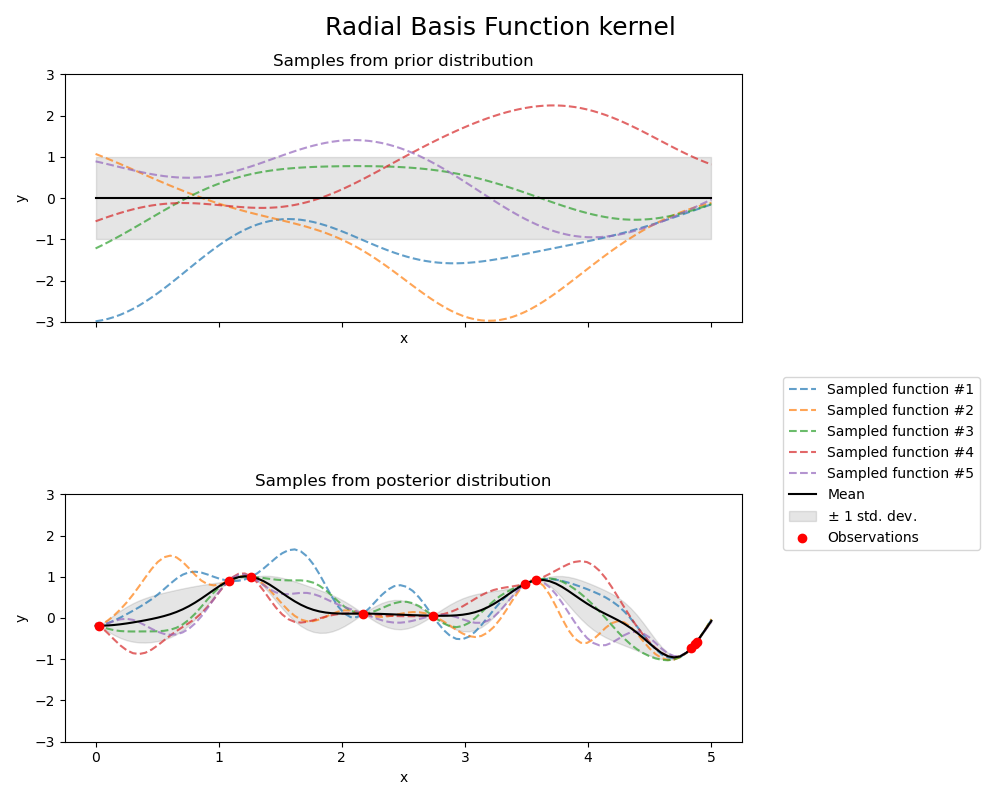
print(f"Kernel parameters before fit:\n{kernel})")
print(
f"Kernel parameters after fit: \n{gpr.kernel_} \n"
f"Log-likelihood: {gpr.log_marginal_likelihood(gpr.kernel_.theta):.3f}"
)
Kernel parameters before fit:
1**2 * RBF(length_scale=1))
Kernel parameters after fit:
0.594**2 * RBF(length_scale=0.279)
Log-likelihood: -0.067
Rational Quadratic Kernel#
from sklearn.gaussian_process.kernels import RationalQuadratic
kernel = 1.0 * RationalQuadratic(length_scale=1.0, alpha=0.1, alpha_bounds=(1e-5, 1e15))
gpr = GaussianProcessRegressor(kernel=kernel, random_state=0)
fig, axs = plt.subplots(nrows=2, sharex=True, sharey=True, figsize=(10, 8))
# plot prior
plot_gpr_samples(gpr, n_samples=n_samples, ax=axs[0])
axs[0].set_title("Samples from prior distribution")
# plot posterior
gpr.fit(X_train, y_train)
plot_gpr_samples(gpr, n_samples=n_samples, ax=axs[1])
axs[1].scatter(X_train[:, 0], y_train, color="red", zorder=10, label="Observations")
axs[1].legend(bbox_to_anchor=(1.05, 1.5), loc="upper left")
axs[1].set_title("Samples from posterior distribution")
fig.suptitle("Rational Quadratic kernel", fontsize=18)
plt.tight_layout()
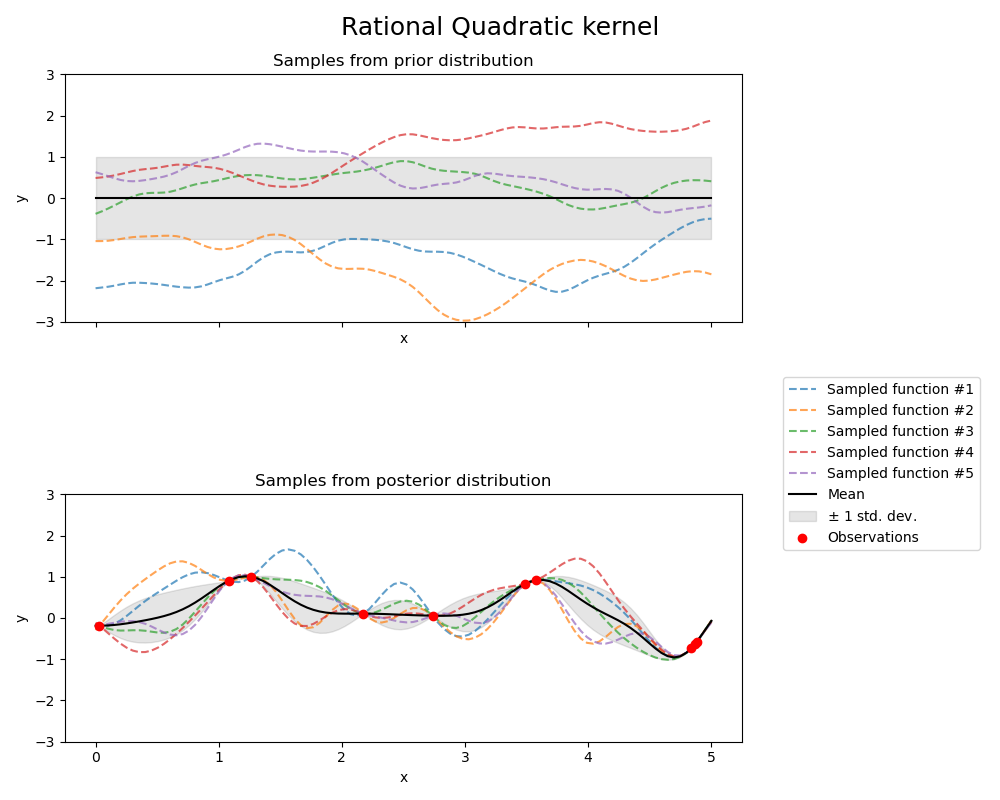
print(f"Kernel parameters before fit:\n{kernel})")
print(
f"Kernel parameters after fit: \n{gpr.kernel_} \n"
f"Log-likelihood: {gpr.log_marginal_likelihood(gpr.kernel_.theta):.3f}"
)
Kernel parameters before fit:
1**2 * RationalQuadratic(alpha=0.1, length_scale=1))
Kernel parameters after fit:
0.594**2 * RationalQuadratic(alpha=1.73e+06, length_scale=0.279)
Log-likelihood: -0.067
Exp-Sine-Squared Kernel#
from sklearn.gaussian_process.kernels import ExpSineSquared
kernel = 1.0 * ExpSineSquared(
length_scale=1.0,
periodicity=3.0,
length_scale_bounds=(0.1, 10.0),
periodicity_bounds=(1.0, 10.0),
)
gpr = GaussianProcessRegressor(kernel=kernel, random_state=0)
fig, axs = plt.subplots(nrows=2, sharex=True, sharey=True, figsize=(10, 8))
# plot prior
plot_gpr_samples(gpr, n_samples=n_samples, ax=axs[0])
axs[0].set_title("Samples from prior distribution")
# plot posterior
gpr.fit(X_train, y_train)
plot_gpr_samples(gpr, n_samples=n_samples, ax=axs[1])
axs[1].scatter(X_train[:, 0], y_train, color="red", zorder=10, label="Observations")
axs[1].legend(bbox_to_anchor=(1.05, 1.5), loc="upper left")
axs[1].set_title("Samples from posterior distribution")
fig.suptitle("Exp-Sine-Squared kernel", fontsize=18)
plt.tight_layout()
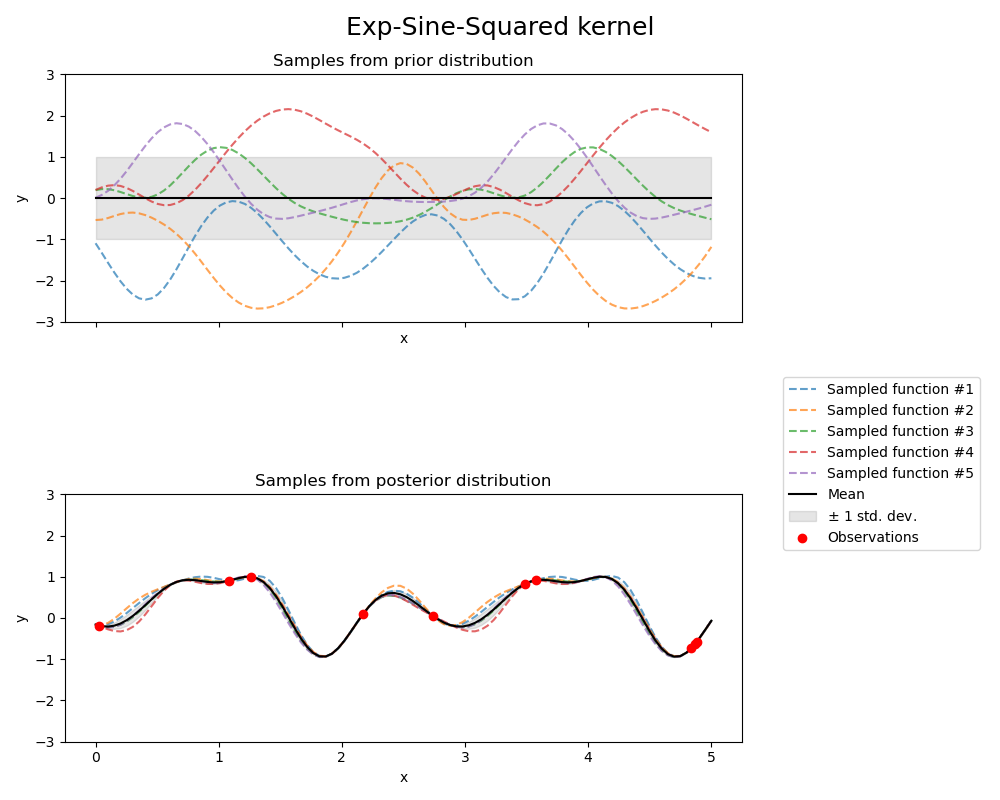
print(f"Kernel parameters before fit:\n{kernel})")
print(
f"Kernel parameters after fit: \n{gpr.kernel_} \n"
f"Log-likelihood: {gpr.log_marginal_likelihood(gpr.kernel_.theta):.3f}"
)
Kernel parameters before fit:
1**2 * ExpSineSquared(length_scale=1, periodicity=3))
Kernel parameters after fit:
0.799**2 * ExpSineSquared(length_scale=0.791, periodicity=2.87)
Log-likelihood: 3.394
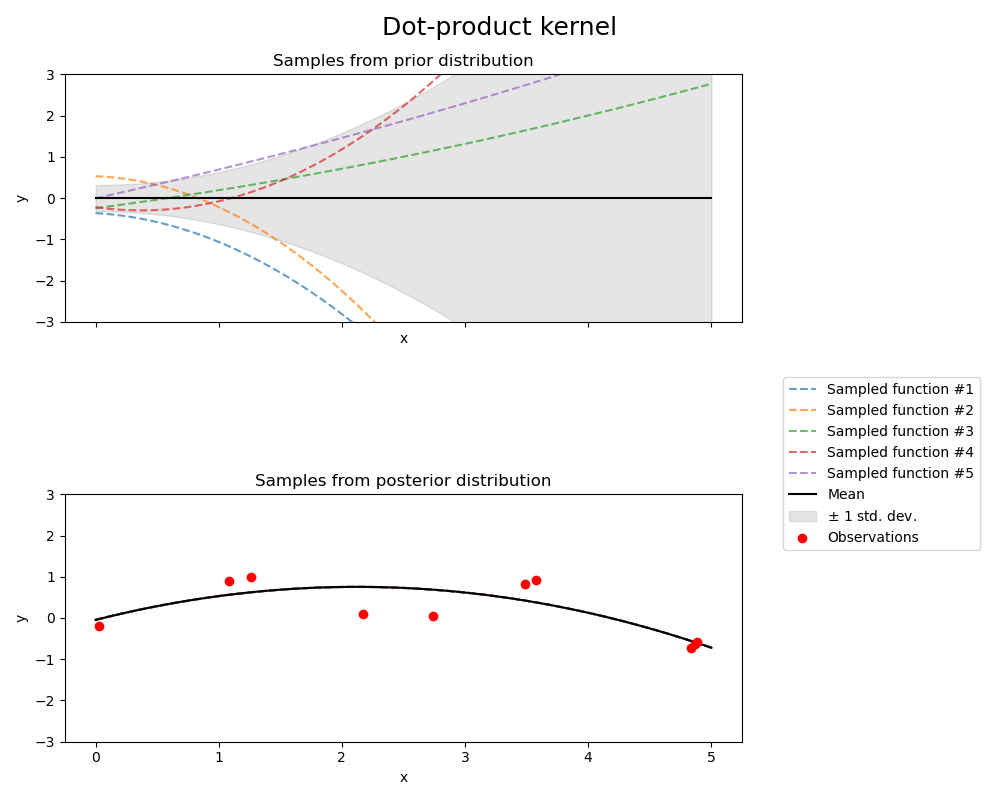
Dot-product Kernel#
from sklearn.gaussian_process.kernels import ConstantKernel, DotProduct
kernel = ConstantKernel(0.1, (0.01, 10.0)) * (
DotProduct(sigma_0=1.0, sigma_0_bounds=(0.1, 10.0)) ** 2
)
gpr = GaussianProcessRegressor(kernel=kernel, random_state=0, normalize_y=True)
fig, axs = plt.subplots(nrows=2, sharex=True, sharey=True, figsize=(10, 8))
# plot prior
plot_gpr_samples(gpr, n_samples=n_samples, ax=axs[0])
axs[0].set_title("Samples from prior distribution")
# plot posterior
gpr.fit(X_train, y_train)
plot_gpr_samples(gpr, n_samples=n_samples, ax=axs[1])
axs[1].scatter(X_train[:, 0], y_train, color="red", zorder=10, label="Observations")
axs[1].legend(bbox_to_anchor=(1.05, 1.5), loc="upper left")
axs[1].set_title("Samples from posterior distribution")
fig.suptitle("Dot-product kernel", fontsize=18)
plt.tight_layout()
print(f"Kernel parameters before fit:\n{kernel})")
print(
f"Kernel parameters after fit: \n{gpr.kernel_} \n"
f"Log-likelihood: {gpr.log_marginal_likelihood(gpr.kernel_.theta):.3f}"
)
Kernel parameters before fit:
0.316**2 * DotProduct(sigma_0=1) ** 2)
Kernel parameters after fit:
0.697**2 * DotProduct(sigma_0=0.454) ** 2
Log-likelihood: -18108182014.707
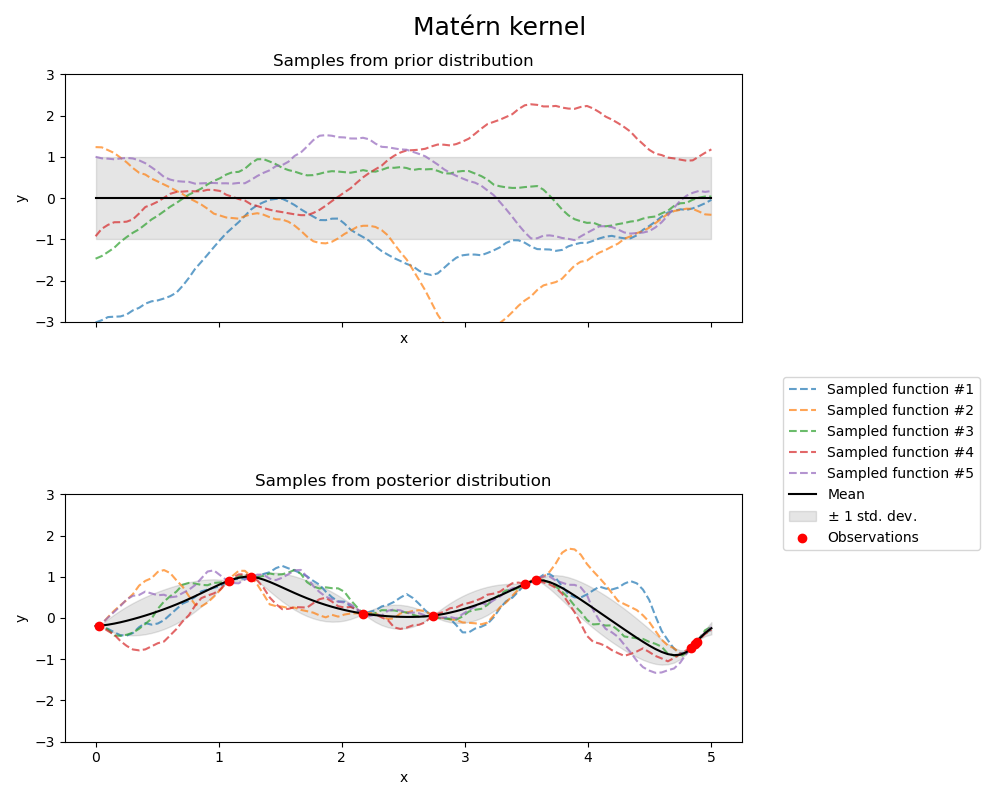
Matérn Kernel#
from sklearn.gaussian_process.kernels import Matern
kernel = 1.0 * Matern(length_scale=1.0, length_scale_bounds=(1e-1, 10.0), nu=1.5)
gpr = GaussianProcessRegressor(kernel=kernel, random_state=0)
fig, axs = plt.subplots(nrows=2, sharex=True, sharey=True, figsize=(10, 8))
# plot prior
plot_gpr_samples(gpr, n_samples=n_samples, ax=axs[0])
axs[0].set_title("Samples from prior distribution")
# plot posterior
gpr.fit(X_train, y_train)
plot_gpr_samples(gpr, n_samples=n_samples, ax=axs[1])
axs[1].scatter(X_train[:, 0], y_train, color="red", zorder=10, label="Observations")
axs[1].legend(bbox_to_anchor=(1.05, 1.5), loc="upper left")
axs[1].set_title("Samples from posterior distribution")
fig.suptitle("Matérn kernel", fontsize=18)
plt.tight_layout()
print(f"Kernel parameters before fit:\n{kernel})")
print(
f"Kernel parameters after fit: \n{gpr.kernel_} \n"
f"Log-likelihood: {gpr.log_marginal_likelihood(gpr.kernel_.theta):.3f}"
)
Kernel parameters before fit:
1**2 * Matern(length_scale=1, nu=1.5))
Kernel parameters after fit:
0.609**2 * Matern(length_scale=0.484, nu=1.5)
Log-likelihood: -1.185
Gesamtlaufzeit des Skripts: (0 Minuten 1,205 Sekunden)
Verwandte Beispiele
Fähigkeit der Gauß-Prozess-Regression (GPR) zur Schätzung des Datenrauschpegels
Vergleich der Leistung von Bisecting K-Means und Regular K-Means
Gauß-Prozesse Regression: grundlegendes Einführungsexempel