Hinweis
Gehe zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in deinem Browser auszuführen.
Lernkurven plotten und Skalierbarkeit von Modellen prüfen#
In diesem Beispiel zeigen wir, wie die Klasse LearningCurveDisplay verwendet werden kann, um einfach Lernkurven zu plotten. Darüber hinaus geben wir eine Interpretation der Lernkurven, die für einen Naive Bayes und SVM-Klassifikator erhalten wurden.
Anschließend untersuchen wir die Skalierbarkeit dieser prädiktiven Modelle und ziehen einige Schlussfolgerungen, indem wir ihre rechnerischen Kosten und nicht nur ihre statistische Genauigkeit betrachten.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Lernkurve#
Lernkurven zeigen den Effekt des Hinzufügens weiterer Stichproben während des Trainingsprozesses. Der Effekt wird durch die Überprüfung der statistischen Leistung des Modells in Bezug auf den Trainings- und Test-Score dargestellt.
Hier berechnen wir die Lernkurve eines Naive Bayes-Klassifikators und eines SVM-Klassifikators mit einem RBF-Kernel unter Verwendung des Digits-Datensatzes.
from sklearn.datasets import load_digits
from sklearn.naive_bayes import GaussianNB
from sklearn.svm import SVC
X, y = load_digits(return_X_y=True)
naive_bayes = GaussianNB()
svc = SVC(kernel="rbf", gamma=0.001)
Die Methode from_estimator zeigt die Lernkurve, gegeben den Datensatz und das zu analysierende prädiktive Modell. Um eine Schätzung der Unsicherheit der Scores zu erhalten, verwendet diese Methode ein Kreuzvalidierungsverfahren.
import matplotlib.pyplot as plt
import numpy as np
from sklearn.model_selection import LearningCurveDisplay, ShuffleSplit
fig, ax = plt.subplots(nrows=1, ncols=2, figsize=(10, 6), sharey=True)
common_params = {
"X": X,
"y": y,
"train_sizes": np.linspace(0.1, 1.0, 5),
"cv": ShuffleSplit(n_splits=50, test_size=0.2, random_state=0),
"score_type": "both",
"n_jobs": 4,
"line_kw": {"marker": "o"},
"std_display_style": "fill_between",
"score_name": "Accuracy",
}
for ax_idx, estimator in enumerate([naive_bayes, svc]):
LearningCurveDisplay.from_estimator(estimator, **common_params, ax=ax[ax_idx])
handles, label = ax[ax_idx].get_legend_handles_labels()
ax[ax_idx].legend(handles[:2], ["Training Score", "Test Score"])
ax[ax_idx].set_title(f"Learning Curve for {estimator.__class__.__name__}")

Wir analysieren zunächst die Lernkurve des Naive Bayes-Klassifikators. Ihre Form ist bei komplexeren Datensätzen sehr oft anzutreffen: Der Trainings-Score ist sehr hoch, wenn wenige Stichproben zum Trainieren verwendet werden, und sinkt, wenn die Anzahl der Stichproben zunimmt, während der Test-Score anfangs sehr niedrig ist und dann steigt, wenn Stichproben hinzugefügt werden. Die Trainings- und Test-Scores werden realistischer, wenn alle Stichproben zum Trainieren verwendet werden.
Wir sehen eine weitere typische Lernkurve für den SVM-Klassifikator mit RBF-Kernel. Der Trainings-Score bleibt unabhängig von der Größe des Trainingsdatensatzes hoch. Auf der anderen Seite steigt der Test-Score mit der Größe des Trainingsdatensatzes. Tatsächlich steigt er bis zu einem Punkt, an dem er ein Plateau erreicht. Die Beobachtung eines solchen Plateaus ist ein Hinweis darauf, dass es möglicherweise nicht nützlich ist, neue Daten zu erwerben, um das Modell zu trainieren, da die Generalisierungsleistung des Modells nicht mehr zunehmen wird.
Komplexitätsanalyse#
Zusätzlich zu diesen Lernkurven ist es auch möglich, die Skalierbarkeit der prädiktiven Modelle in Bezug auf Trainings- und Scoring-Zeiten zu betrachten.
Die Klasse LearningCurveDisplay bietet solche Informationen nicht. Stattdessen müssen wir auf die Funktion learning_curve zurückgreifen und den Plot manuell erstellen.
from sklearn.model_selection import learning_curve
common_params = {
"X": X,
"y": y,
"train_sizes": np.linspace(0.1, 1.0, 5),
"cv": ShuffleSplit(n_splits=50, test_size=0.2, random_state=0),
"n_jobs": 4,
"return_times": True,
}
train_sizes, _, test_scores_nb, fit_times_nb, score_times_nb = learning_curve(
naive_bayes, **common_params
)
train_sizes, _, test_scores_svm, fit_times_svm, score_times_svm = learning_curve(
svc, **common_params
)
fig, ax = plt.subplots(nrows=2, ncols=2, figsize=(16, 12), sharex=True)
for ax_idx, (fit_times, score_times, estimator) in enumerate(
zip(
[fit_times_nb, fit_times_svm],
[score_times_nb, score_times_svm],
[naive_bayes, svc],
)
):
# scalability regarding the fit time
ax[0, ax_idx].plot(train_sizes, fit_times.mean(axis=1), "o-")
ax[0, ax_idx].fill_between(
train_sizes,
fit_times.mean(axis=1) - fit_times.std(axis=1),
fit_times.mean(axis=1) + fit_times.std(axis=1),
alpha=0.3,
)
ax[0, ax_idx].set_ylabel("Fit time (s)")
ax[0, ax_idx].set_title(
f"Scalability of the {estimator.__class__.__name__} classifier"
)
# scalability regarding the score time
ax[1, ax_idx].plot(train_sizes, score_times.mean(axis=1), "o-")
ax[1, ax_idx].fill_between(
train_sizes,
score_times.mean(axis=1) - score_times.std(axis=1),
score_times.mean(axis=1) + score_times.std(axis=1),
alpha=0.3,
)
ax[1, ax_idx].set_ylabel("Score time (s)")
ax[1, ax_idx].set_xlabel("Number of training samples")

Wir sehen, dass die Skalierbarkeit der SVM- und Naive Bayes-Klassifikatoren sehr unterschiedlich ist. Die Komplexität des SVM-Klassifikators zur Fit- und Score-Zeit steigt mit der Anzahl der Stichproben schnell an. Tatsächlich ist bekannt, dass die Fit-Zeit-Komplexität dieses Klassifikators mehr als quadratisch mit der Anzahl der Stichproben ist, was die Skalierung auf Datensätze mit mehr als einigen 10.000 Stichproben erschwert. Im Gegensatz dazu skaliert der Naive Bayes-Klassifikator mit einer geringeren Komplexität zur Fit- und Score-Zeit wesentlich besser.
Anschließend können wir den Kompromiss zwischen erhöhter Trainingszeit und dem Kreuzvalidierungs-Score überprüfen.
fig, ax = plt.subplots(nrows=1, ncols=2, figsize=(16, 6))
for ax_idx, (fit_times, test_scores, estimator) in enumerate(
zip(
[fit_times_nb, fit_times_svm],
[test_scores_nb, test_scores_svm],
[naive_bayes, svc],
)
):
ax[ax_idx].plot(fit_times.mean(axis=1), test_scores.mean(axis=1), "o-")
ax[ax_idx].fill_between(
fit_times.mean(axis=1),
test_scores.mean(axis=1) - test_scores.std(axis=1),
test_scores.mean(axis=1) + test_scores.std(axis=1),
alpha=0.3,
)
ax[ax_idx].set_ylabel("Accuracy")
ax[ax_idx].set_xlabel("Fit time (s)")
ax[ax_idx].set_title(
f"Performance of the {estimator.__class__.__name__} classifier"
)
plt.show()

In diesen Plots können wir nach dem Wendepunkt suchen, an dem der Kreuzvalidierungs-Score nicht mehr steigt und nur noch die Trainingszeit zunimmt.
Gesamtlaufzeit des Skripts: (0 Minuten 29,969 Sekunden)
Verwandte Beispiele
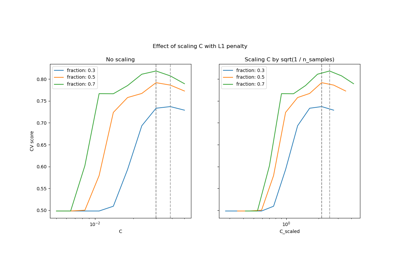
Skalierung des Regularisierungsparameters für SVCs
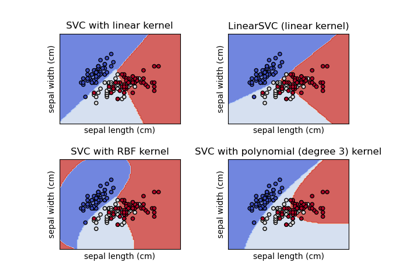
Verschiedene SVM-Klassifikatoren im Iris-Datensatz plotten