Hinweis
Gehen Sie zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Feature-Importanzen mit einem Wald von Bäumen#
Dieses Beispiel zeigt die Verwendung eines Waldes von Bäumen zur Bewertung der Bedeutung von Merkmalen bei einer künstlichen Klassifizierungsaufgabe. Die blauen Balken stellen die Feature-Importanzen des Waldes dar, zusammen mit ihrer Variabilität zwischen den Bäumen, die durch die Fehlerbalken repräsentiert wird.
Wie erwartet legt die Darstellung nahe, dass 3 Merkmale informativ sind, während die übrigen es nicht sind.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
import matplotlib.pyplot as plt
Datengenerierung und Modellfitting#
Wir generieren einen synthetischen Datensatz mit nur 3 informativen Merkmalen. Wir werden den Datensatz explizit nicht mischen, um sicherzustellen, dass die informativen Merkmale den drei ersten Spalten von X entsprechen. Darüber hinaus werden wir unseren Datensatz in Trainings- und Testuntergruppen aufteilen.
from sklearn.datasets import make_classification
from sklearn.model_selection import train_test_split
X, y = make_classification(
n_samples=1000,
n_features=10,
n_informative=3,
n_redundant=0,
n_repeated=0,
n_classes=2,
random_state=0,
shuffle=False,
)
X_train, X_test, y_train, y_test = train_test_split(X, y, stratify=y, random_state=42)
Ein Random Forest-Klassifikator wird gefittet, um die Feature-Importanzen zu berechnen.
from sklearn.ensemble import RandomForestClassifier
feature_names = [f"feature {i}" for i in range(X.shape[1])]
forest = RandomForestClassifier(random_state=0)
forest.fit(X_train, y_train)
Feature-Importanz basierend auf der mittleren Verringerung der Unreinheit#
Feature-Importanzen werden durch das gefittete Attribut feature_importances_ bereitgestellt und als Mittelwert und Standardabweichung der Akkumulation der Unreinheitsverringerung innerhalb jedes Baumes berechnet.
Warnung
Unreinheitsbasierte Feature-Importanzen können bei **Merkmalen mit hoher Kardinalität** (viele eindeutige Werte) irreführend sein. Siehe Permutation Feature Importance als Alternative unten.
Elapsed time to compute the importances: 0.014 seconds
Lassen Sie uns die unreinheitsbasierte Bedeutung plotten.
import pandas as pd
forest_importances = pd.Series(importances, index=feature_names)
fig, ax = plt.subplots()
forest_importances.plot.bar(yerr=std, ax=ax)
ax.set_title("Feature importances using MDI")
ax.set_ylabel("Mean decrease in impurity")
fig.tight_layout()

Wir stellen fest, dass, wie erwartet, die ersten drei Merkmale als wichtig erachtet werden.
Feature-Importanz basierend auf Permutation von Merkmalen#
Die Permutation Feature Importance überwindet Einschränkungen der unreinheitsbasierten Feature Importance: Sie hat keine Voreingenommenheit gegenüber Merkmalen mit hoher Kardinalität und kann auf einem links liegenden Testdatensatz berechnet werden.
from sklearn.inspection import permutation_importance
start_time = time.time()
result = permutation_importance(
forest, X_test, y_test, n_repeats=10, random_state=42, n_jobs=2
)
elapsed_time = time.time() - start_time
print(f"Elapsed time to compute the importances: {elapsed_time:.3f} seconds")
forest_importances = pd.Series(result.importances_mean, index=feature_names)
Elapsed time to compute the importances: 0.906 seconds
Die Berechnung für die vollständige Permutations-Wichtigkeit ist aufwendiger. Jedes Merkmal wird n Mal gemischt und das Modell wird verwendet, um Vorhersagen auf den permutierten Daten zu treffen, um den Leistungsabfall zu sehen. Weitere Einzelheiten finden Sie unter Permutation Feature Importance. Wir können nun das Ranking der Wichtigkeit plotten.
fig, ax = plt.subplots()
forest_importances.plot.bar(yerr=result.importances_std, ax=ax)
ax.set_title("Feature importances using permutation on full model")
ax.set_ylabel("Mean accuracy decrease")
fig.tight_layout()
plt.show()

Dieselbe Merkmale werden mit beiden Methoden als am wichtigsten erkannt. Obwohl sich die relativen Bedeutungen unterscheiden. Wie in den Plots zu sehen ist, ist MDI weniger wahrscheinlich als die Permutations-Wichtigkeit, ein Merkmal vollständig auszulassen.
Gesamtlaufzeit des Skripts: (0 Minuten 1,351 Sekunden)
Verwandte Beispiele

Permutations-Wichtigkeit vs. Random Forest Merkmals-Wichtigkeit (MDI)
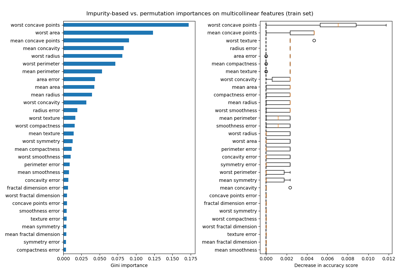
Permutations-Wichtigkeit bei multikollinearen oder korrelierten Merkmalen