Hinweis
Zum Ende gehen, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Release Highlights für scikit-learn 0.22#
Wir freuen uns, die Veröffentlichung von scikit-learn 0.22 bekannt zu geben, die viele Fehlerbehebungen und neue Funktionen enthält! Nachfolgend erläutern wir einige der wichtigsten Neuerungen dieser Version. Eine vollständige Liste aller Änderungen finden Sie in den Release Notes.
Um die neueste Version zu installieren (mit pip)
pip install --upgrade scikit-learn
oder mit conda
conda install -c conda-forge scikit-learn
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Neue Plotting-API#
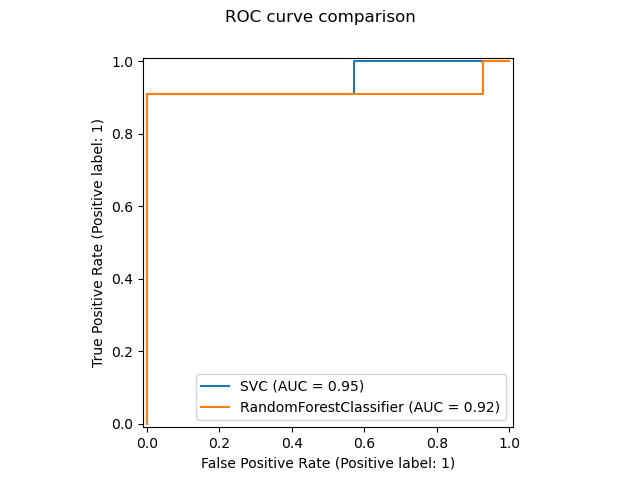
Eine neue Plotting-API steht zur Erstellung von Visualisierungen zur Verfügung. Diese neue API ermöglicht die schnelle Anpassung der visuellen Darstellung eines Plots, ohne dass eine Neuberechnung erforderlich ist. Es ist auch möglich, verschiedene Plots zur selben Abbildung hinzuzufügen. Das folgende Beispiel illustriert plot_roc_curve, aber es werden auch andere Plot-Utilities unterstützt, wie z. B. plot_partial_dependence, plot_precision_recall_curve und plot_confusion_matrix. Lesen Sie mehr über diese neue API im Benutzerhandbuch.
import matplotlib
import matplotlib.pyplot as plt
from sklearn.datasets import make_classification
from sklearn.ensemble import RandomForestClassifier
# from sklearn.metrics import plot_roc_curve
from sklearn.metrics import RocCurveDisplay
from sklearn.model_selection import train_test_split
from sklearn.svm import SVC
from sklearn.utils.fixes import parse_version
X, y = make_classification(random_state=0)
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42)
svc = SVC(random_state=42)
svc.fit(X_train, y_train)
rfc = RandomForestClassifier(random_state=42)
rfc.fit(X_train, y_train)
# plot_roc_curve has been removed in version 1.2. From 1.2, use RocCurveDisplay instead.
# svc_disp = plot_roc_curve(svc, X_test, y_test)
# rfc_disp = plot_roc_curve(rfc, X_test, y_test, ax=svc_disp.ax_)
svc_disp = RocCurveDisplay.from_estimator(svc, X_test, y_test)
rfc_disp = RocCurveDisplay.from_estimator(rfc, X_test, y_test, ax=svc_disp.ax_)
rfc_disp.figure_.suptitle("ROC curve comparison")
plt.show()
Stacking Classifier und Regressor#
StackingClassifier und StackingRegressor ermöglichen Ihnen, einen Stapel von Schätzern mit einem finalen Klassifikator oder Regressor zu haben. Gestapelte Generalisierung besteht darin, die Ausgaben einzelner Schätzer zu stapeln und einen Klassifikator zur Berechnung der finalen Vorhersage zu verwenden. Stacking ermöglicht es, die Stärken jedes einzelnen Schätzers zu nutzen, indem deren Ausgaben als Eingaben für einen finalen Schätzer verwendet werden. Basisschätzer werden auf dem gesamten X trainiert, während der finale Schätzer mithilfe von kreuzvalidierten Vorhersagen der Basisschätzer unter Verwendung von cross_val_predict trainiert wird.
Lesen Sie mehr im Benutzerhandbuch.
from sklearn.datasets import load_iris
from sklearn.ensemble import StackingClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.model_selection import train_test_split
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.svm import LinearSVC
X, y = load_iris(return_X_y=True)
estimators = [
("rf", RandomForestClassifier(n_estimators=10, random_state=42)),
("svr", make_pipeline(StandardScaler(), LinearSVC(dual="auto", random_state=42))),
]
clf = StackingClassifier(estimators=estimators, final_estimator=LogisticRegression())
X_train, X_test, y_train, y_test = train_test_split(X, y, stratify=y, random_state=42)
clf.fit(X_train, y_train).score(X_test, y_test)
0.9473684210526315
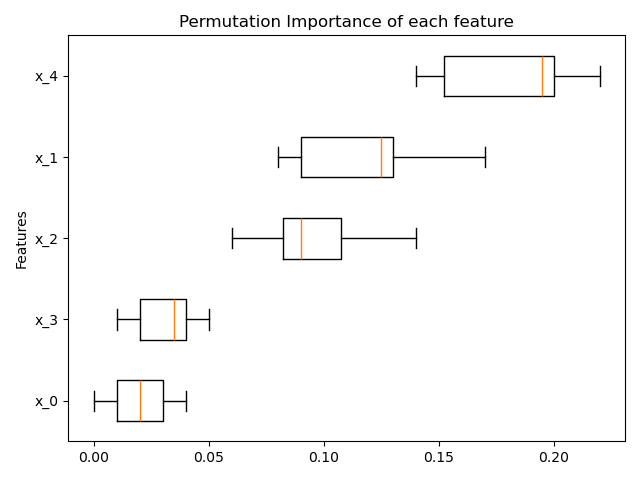
Permutationsbasierte Feature-Wichtigkeit#
Die Funktion inspection.permutation_importance kann verwendet werden, um eine Schätzung der Wichtigkeit jedes Merkmals für jeden trainierten Schätzer zu erhalten.
import matplotlib.pyplot as plt
import numpy as np
from sklearn.datasets import make_classification
from sklearn.ensemble import RandomForestClassifier
from sklearn.inspection import permutation_importance
X, y = make_classification(random_state=0, n_features=5, n_informative=3)
feature_names = np.array([f"x_{i}" for i in range(X.shape[1])])
rf = RandomForestClassifier(random_state=0).fit(X, y)
result = permutation_importance(rf, X, y, n_repeats=10, random_state=0, n_jobs=2)
fig, ax = plt.subplots()
sorted_idx = result.importances_mean.argsort()
# `labels` argument in boxplot is deprecated in matplotlib 3.9 and has been
# renamed to `tick_labels`. The following code handles this, but as a
# scikit-learn user you probably can write simpler code by using `labels=...`
# (matplotlib < 3.9) or `tick_labels=...` (matplotlib >= 3.9).
tick_labels_parameter_name = (
"tick_labels"
if parse_version(matplotlib.__version__) >= parse_version("3.9")
else "labels"
)
tick_labels_dict = {tick_labels_parameter_name: feature_names[sorted_idx]}
ax.boxplot(result.importances[sorted_idx].T, vert=False, **tick_labels_dict)
ax.set_title("Permutation Importance of each feature")
ax.set_ylabel("Features")
fig.tight_layout()
plt.show()
Native Unterstützung für fehlende Werte für Gradient Boosting#
Die Klassen ensemble.HistGradientBoostingClassifier und ensemble.HistGradientBoostingRegressor haben jetzt native Unterstützung für fehlende Werte (NaNs). Das bedeutet, dass keine Daten imputiert werden müssen, wenn trainiert oder vorhergesagt wird.
from sklearn.ensemble import HistGradientBoostingClassifier
X = np.array([0, 1, 2, np.nan]).reshape(-1, 1)
y = [0, 0, 1, 1]
gbdt = HistGradientBoostingClassifier(min_samples_leaf=1).fit(X, y)
print(gbdt.predict(X))
[0 0 1 1]
Vorberechneter spärlicher Nachbar-Graph#
Die meisten auf Nachbar-Graphen basierenden Schätzer akzeptieren jetzt vorberechnete spärliche Graphen als Eingabe, um denselben Graphen für mehrere Schätzeranpassungen wiederzuverwenden. Um diese Funktion in einer Pipeline zu nutzen, kann der Parameter memory zusammen mit einem der beiden neuen Transformer, neighbors.KNeighborsTransformer und neighbors.RadiusNeighborsTransformer, verwendet werden. Die Vorberechnung kann auch von benutzerdefinierten Schätzern durchgeführt werden, um alternative Implementierungen zu verwenden, wie z. B. approximative Nachbar-Methoden. Weitere Details finden Sie im Benutzerhandbuch.
from tempfile import TemporaryDirectory
from sklearn.manifold import Isomap
from sklearn.neighbors import KNeighborsTransformer
from sklearn.pipeline import make_pipeline
X, y = make_classification(random_state=0)
with TemporaryDirectory(prefix="sklearn_cache_") as tmpdir:
estimator = make_pipeline(
KNeighborsTransformer(n_neighbors=10, mode="distance"),
Isomap(n_neighbors=10, metric="precomputed"),
memory=tmpdir,
)
estimator.fit(X)
# We can decrease the number of neighbors and the graph will not be
# recomputed.
estimator.set_params(isomap__n_neighbors=5)
estimator.fit(X)
KNN-basierte Imputation#
Wir unterstützen jetzt die Imputation zum Auffüllen fehlender Werte mithilfe von k-Nearest Neighbors.
Die fehlenden Werte jedes Samples werden mithilfe des Durchschnittswerts der n_neighbors nächsten Nachbarn, die im Trainingsdatensatz gefunden wurden, imputiert. Zwei Samples sind nahe beieinander, wenn die Merkmale, die bei keinem von beiden fehlen, nahe beieinander liegen. Standardmäßig wird eine euklidische Distanzmetrik, die fehlende Werte unterstützt, nan_euclidean_distances, verwendet, um die nächsten Nachbarn zu finden.
Lesen Sie mehr im Benutzerhandbuch.
from sklearn.impute import KNNImputer
X = [[1, 2, np.nan], [3, 4, 3], [np.nan, 6, 5], [8, 8, 7]]
imputer = KNNImputer(n_neighbors=2)
print(imputer.fit_transform(X))
[[1. 2. 4. ]
[3. 4. 3. ]
[5.5 6. 5. ]
[8. 8. 7. ]]
Baumschnitt (Pruning)#
Es ist jetzt möglich, die meisten baumbasierten Schätzer zu beschneiden, nachdem die Bäume erstellt wurden. Der Schnitt basiert auf minimaler Kosten-Komplexität. Lesen Sie mehr im Benutzerhandbuch für Details.
X, y = make_classification(random_state=0)
rf = RandomForestClassifier(random_state=0, ccp_alpha=0).fit(X, y)
print(
"Average number of nodes without pruning {:.1f}".format(
np.mean([e.tree_.node_count for e in rf.estimators_])
)
)
rf = RandomForestClassifier(random_state=0, ccp_alpha=0.05).fit(X, y)
print(
"Average number of nodes with pruning {:.1f}".format(
np.mean([e.tree_.node_count for e in rf.estimators_])
)
)
Average number of nodes without pruning 22.3
Average number of nodes with pruning 6.4
Abrufen von DataFrames von OpenML#
datasets.fetch_openml kann jetzt Pandas DataFrames zurückgeben und somit Datensätze mit heterogenen Daten korrekt verarbeiten.
from sklearn.datasets import fetch_openml
titanic = fetch_openml("titanic", version=1, as_frame=True, parser="pandas")
print(titanic.data.head()[["pclass", "embarked"]])
pclass embarked
0 1 S
1 1 S
2 1 S
3 1 S
4 1 S
Überprüfung der scikit-learn-Kompatibilität eines Schätzers#
Entwickler können die Kompatibilität ihrer scikit-learn-kompatiblen Schätzer mit check_estimator überprüfen. Zum Beispiel besteht check_estimator(LinearSVC()).
Wir bieten jetzt einen pytest-spezifischen Dekorator an, der es pytest ermöglicht, alle Überprüfungen unabhängig auszuführen und die fehlgeschlagenen Überprüfungen zu melden.
- ..note:
Dieser Eintrag wurde in Version 0.24 leicht aktualisiert, bei der die Übergabe von Klassen nicht mehr unterstützt wird: übergeben Sie stattdessen Instanzen.
from sklearn.linear_model import LogisticRegression
from sklearn.tree import DecisionTreeRegressor
from sklearn.utils.estimator_checks import parametrize_with_checks
@parametrize_with_checks([LogisticRegression(), DecisionTreeRegressor()])
def test_sklearn_compatible_estimator(estimator, check):
check(estimator)
ROC AUC unterstützt jetzt Multiklassen-Klassifizierung#
Die Funktion roc_auc_score kann auch in der Multiklassen-Klassifizierung verwendet werden. Zwei Durchschnittsstrategien werden derzeit unterstützt: der One-vs-One-Algorithmus berechnet den Durchschnitt der paarweisen ROC-AUC-Scores, und der One-vs-Rest-Algorithmus berechnet den Durchschnitt der ROC-AUC-Scores für jede Klasse gegen alle anderen Klassen. In beiden Fällen werden die Multiklassen-ROC-AUC-Scores aus den Wahrscheinlichkeitsschätzungen berechnet, dass ein Sample gemäß dem Modell zu einer bestimmten Klasse gehört. Die OvO- und OvR-Algorithmen unterstützen die gleichmäßige Gewichtung (average='macro') und die Gewichtung nach der Prävalenz (average='weighted').
Lesen Sie mehr im Benutzerhandbuch.
from sklearn.datasets import make_classification
from sklearn.metrics import roc_auc_score
from sklearn.svm import SVC
X, y = make_classification(n_classes=4, n_informative=16)
clf = SVC(decision_function_shape="ovo", probability=True).fit(X, y)
print(roc_auc_score(y, clf.predict_proba(X), multi_class="ovo"))
0.9892
Gesamtlaufzeit des Skripts: (0 Minuten 1,661 Sekunden)
Verwandte Beispiele