Hinweis
Zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Modellkomplexität und kreuzvalidierte Punktzahl ausbalancieren#
Dieses Beispiel demonstriert, wie die Modellkomplexität und die kreuzvalidierte Punktzahl ausbalanciert werden, indem eine angemessene Genauigkeit innerhalb von 1 Standardabweichung der besten Genauigkeit erzielt wird, während die Anzahl der PCA-Komponenten minimiert wird [1]. Es verwendet GridSearchCV mit einem benutzerdefinierten Refit-Callable, um das optimale Modell auszuwählen.
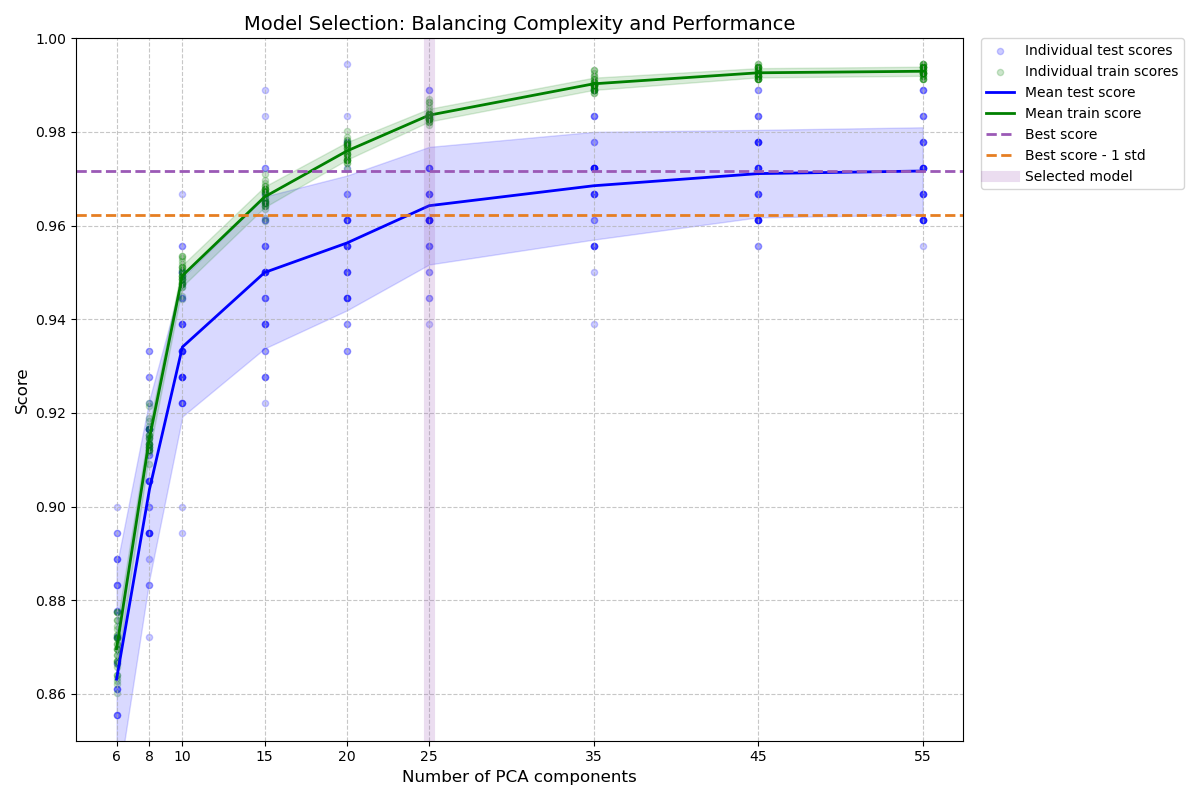
Die Abbildung zeigt den Kompromiss zwischen der kreuzvalidierten Punktzahl und der Anzahl der PCA-Komponenten. Der ausgewogene Fall ist, wenn n_components=10 und accuracy=0.88 ist, was in den Bereich innerhalb von 1 Standardabweichung der besten Genauigkeitspunktzahl fällt.
Referenzen#
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
import matplotlib.pyplot as plt
import numpy as np
import polars as pl
from sklearn.datasets import load_digits
from sklearn.decomposition import PCA
from sklearn.linear_model import LogisticRegression
from sklearn.model_selection import GridSearchCV, ShuffleSplit
from sklearn.pipeline import Pipeline
Einleitung#
Beim Abstimmen von Hyperparametern wollen wir oft Modellkomplexität und Leistung ausbalancieren. Die „Ein-Standardfehler“-Regel ist ein gängiger Ansatz: Wählen Sie das einfachste Modell, dessen Leistung innerhalb eines Standardfehlers der Leistung des besten Modells liegt. Dies hilft, Überanpassung zu vermeiden, indem einfachere Modelle bevorzugt werden, wenn ihre Leistung statistisch vergleichbar mit komplexeren Modellen ist.
Hilfsfunktionen#
Wir definieren zwei Hilfsfunktionen
lower_bound: Berechnet die Schwelle für akzeptable Leistung (beste Punktzahl - 1 Std.)best_low_complexity: Wählt das Modell mit den wenigsten PCA-Komponenten aus, das diesen Schwellenwert überschreitet
def lower_bound(cv_results):
"""
Calculate the lower bound within 1 standard deviation
of the best `mean_test_scores`.
Parameters
----------
cv_results : dict of numpy(masked) ndarrays
See attribute cv_results_ of `GridSearchCV`
Returns
-------
float
Lower bound within 1 standard deviation of the
best `mean_test_score`.
"""
best_score_idx = np.argmax(cv_results["mean_test_score"])
return (
cv_results["mean_test_score"][best_score_idx]
- cv_results["std_test_score"][best_score_idx]
)
def best_low_complexity(cv_results):
"""
Balance model complexity with cross-validated score.
Parameters
----------
cv_results : dict of numpy(masked) ndarrays
See attribute cv_results_ of `GridSearchCV`.
Return
------
int
Index of a model that has the fewest PCA components
while has its test score within 1 standard deviation of the best
`mean_test_score`.
"""
threshold = lower_bound(cv_results)
candidate_idx = np.flatnonzero(cv_results["mean_test_score"] >= threshold)
best_idx = candidate_idx[
cv_results["param_reduce_dim__n_components"][candidate_idx].argmin()
]
return best_idx
Pipeline und Parametergitter einrichten#
Wir erstellen eine Pipeline mit zwei Schritten
Dimensionsreduktion mittels PCA
Klassifizierung mittels LogisticRegression
Wir suchen nach verschiedenen Anzahlen von PCA-Komponenten, um die optimale Komplexität zu finden.
pipe = Pipeline(
[
("reduce_dim", PCA(random_state=42)),
("classify", LogisticRegression(random_state=42, C=0.01, max_iter=1000)),
]
)
param_grid = {"reduce_dim__n_components": [6, 8, 10, 15, 20, 25, 35, 45, 55]}
Suche mit GridSearchCV durchführen#
Wir verwenden GridSearchCV mit unserer benutzerdefinierten Funktion best_low_complexity als Refit-Parameter. Diese Funktion wählt das Modell mit den wenigsten PCA-Komponenten aus, das immer noch innerhalb einer Standardabweichung des besten Modells leistet.
grid = GridSearchCV(
pipe,
# Use a non-stratified CV strategy to make sure that the inter-fold
# standard deviation of the test scores is informative.
cv=ShuffleSplit(n_splits=30, random_state=0),
n_jobs=1, # increase this on your machine to use more physical cores
param_grid=param_grid,
scoring="accuracy",
refit=best_low_complexity,
return_train_score=True,
)
Laden des Digits-Datensatzes und Anpassen des Modells#
X, y = load_digits(return_X_y=True)
grid.fit(X, y)
Ergebnisse visualisieren#
Wir erstellen ein Balkendiagramm, das die Testpunktzahlen für verschiedene Anzahlen von PCA-Komponenten zeigt, zusammen mit horizontalen Linien, die die beste Punktzahl und den Schwellenwert einer Standardabweichung angeben.
n_components = grid.cv_results_["param_reduce_dim__n_components"]
test_scores = grid.cv_results_["mean_test_score"]
# Create a polars DataFrame for better data manipulation and visualization
results_df = pl.DataFrame(
{
"n_components": n_components,
"mean_test_score": test_scores,
"std_test_score": grid.cv_results_["std_test_score"],
"mean_train_score": grid.cv_results_["mean_train_score"],
"std_train_score": grid.cv_results_["std_train_score"],
"mean_fit_time": grid.cv_results_["mean_fit_time"],
"rank_test_score": grid.cv_results_["rank_test_score"],
}
)
# Sort by number of components
results_df = results_df.sort("n_components")
# Calculate the lower bound threshold
lower = lower_bound(grid.cv_results_)
# Get the best model information
best_index_ = grid.best_index_
best_components = n_components[best_index_]
best_score = grid.cv_results_["mean_test_score"][best_index_]
# Add a column to mark the selected model
results_df = results_df.with_columns(
pl.when(pl.col("n_components") == best_components)
.then(pl.lit("Selected"))
.otherwise(pl.lit("Regular"))
.alias("model_type")
)
# Get the number of CV splits from the results
n_splits = sum(
1
for key in grid.cv_results_.keys()
if key.startswith("split") and key.endswith("test_score")
)
# Extract individual scores for each split
test_scores = np.array(
[
[grid.cv_results_[f"split{i}_test_score"][j] for i in range(n_splits)]
for j in range(len(n_components))
]
)
train_scores = np.array(
[
[grid.cv_results_[f"split{i}_train_score"][j] for i in range(n_splits)]
for j in range(len(n_components))
]
)
# Calculate mean and std of test scores
mean_test_scores = np.mean(test_scores, axis=1)
std_test_scores = np.std(test_scores, axis=1)
# Find best score and threshold
best_mean_score = np.max(mean_test_scores)
threshold = best_mean_score - std_test_scores[np.argmax(mean_test_scores)]
# Create a single figure for visualization
fig, ax = plt.subplots(figsize=(12, 8))
# Plot individual points
for i, comp in enumerate(n_components):
# Plot individual test points
plt.scatter(
[comp] * n_splits,
test_scores[i],
alpha=0.2,
color="blue",
s=20,
label="Individual test scores" if i == 0 else "",
)
# Plot individual train points
plt.scatter(
[comp] * n_splits,
train_scores[i],
alpha=0.2,
color="green",
s=20,
label="Individual train scores" if i == 0 else "",
)
# Plot mean lines with error bands
plt.plot(
n_components,
np.mean(test_scores, axis=1),
"-",
color="blue",
linewidth=2,
label="Mean test score",
)
plt.fill_between(
n_components,
np.mean(test_scores, axis=1) - np.std(test_scores, axis=1),
np.mean(test_scores, axis=1) + np.std(test_scores, axis=1),
alpha=0.15,
color="blue",
)
plt.plot(
n_components,
np.mean(train_scores, axis=1),
"-",
color="green",
linewidth=2,
label="Mean train score",
)
plt.fill_between(
n_components,
np.mean(train_scores, axis=1) - np.std(train_scores, axis=1),
np.mean(train_scores, axis=1) + np.std(train_scores, axis=1),
alpha=0.15,
color="green",
)
# Add threshold lines
plt.axhline(
best_mean_score,
color="#9b59b6", # Purple
linestyle="--",
label="Best score",
linewidth=2,
)
plt.axhline(
threshold,
color="#e67e22", # Orange
linestyle="--",
label="Best score - 1 std",
linewidth=2,
)
# Highlight selected model
plt.axvline(
best_components,
color="#9b59b6", # Purple
alpha=0.2,
linewidth=8,
label="Selected model",
)
# Set titles and labels
plt.xlabel("Number of PCA components", fontsize=12)
plt.ylabel("Score", fontsize=12)
plt.title("Model Selection: Balancing Complexity and Performance", fontsize=14)
plt.grid(True, linestyle="--", alpha=0.7)
plt.legend(
bbox_to_anchor=(1.02, 1),
loc="upper left",
borderaxespad=0,
)
# Set axis properties
plt.xticks(n_components)
plt.ylim((0.85, 1.0))
# # Adjust layout
plt.tight_layout()
Ergebnisse ausgeben#
Wir geben Informationen über das ausgewählte Modell aus, einschließlich seiner Komplexität und Leistung. Wir zeigen auch eine zusammenfassende Tabelle aller Modelle mit polaren Daten.
print("Best model selected by the one-standard-error rule:")
print(f"Number of PCA components: {best_components}")
print(f"Accuracy score: {best_score:.4f}")
print(f"Best possible accuracy: {np.max(test_scores):.4f}")
print(f"Accuracy threshold (best - 1 std): {lower:.4f}")
# Create a summary table with polars
summary_df = results_df.select(
pl.col("n_components"),
pl.col("mean_test_score").round(4).alias("test_score"),
pl.col("std_test_score").round(4).alias("test_std"),
pl.col("mean_train_score").round(4).alias("train_score"),
pl.col("std_train_score").round(4).alias("train_std"),
pl.col("mean_fit_time").round(3).alias("fit_time"),
pl.col("rank_test_score").alias("rank"),
)
# Add a column to mark the selected model
summary_df = summary_df.with_columns(
pl.when(pl.col("n_components") == best_components)
.then(pl.lit("*"))
.otherwise(pl.lit(""))
.alias("selected")
)
print("\nModel comparison table:")
print(summary_df)
Best model selected by the one-standard-error rule:
Number of PCA components: 25
Accuracy score: 0.9643
Best possible accuracy: 0.9944
Accuracy threshold (best - 1 std): 0.9623
Model comparison table:
shape: (9, 8)
┌──────────────┬────────────┬──────────┬─────────────┬───────────┬──────────┬──────┬──────────┐
│ n_components ┆ test_score ┆ test_std ┆ train_score ┆ train_std ┆ fit_time ┆ rank ┆ selected │
│ --- ┆ --- ┆ --- ┆ --- ┆ --- ┆ --- ┆ --- ┆ --- │
│ i64 ┆ f64 ┆ f64 ┆ f64 ┆ f64 ┆ f64 ┆ i32 ┆ str │
╞══════════════╪════════════╪══════════╪═════════════╪═══════════╪══════════╪══════╪══════════╡
│ 6 ┆ 0.8631 ┆ 0.0241 ┆ 0.8697 ┆ 0.0048 ┆ 0.092 ┆ 9 ┆ │
│ 8 ┆ 0.9037 ┆ 0.0192 ┆ 0.9146 ┆ 0.0028 ┆ 0.084 ┆ 8 ┆ │
│ 10 ┆ 0.9341 ┆ 0.0148 ┆ 0.9493 ┆ 0.0023 ┆ 0.058 ┆ 7 ┆ │
│ 15 ┆ 0.95 ┆ 0.0162 ┆ 0.9662 ┆ 0.0022 ┆ 0.055 ┆ 6 ┆ │
│ 20 ┆ 0.9563 ┆ 0.0144 ┆ 0.9759 ┆ 0.0019 ┆ 0.055 ┆ 5 ┆ │
│ 25 ┆ 0.9643 ┆ 0.0126 ┆ 0.9836 ┆ 0.0014 ┆ 0.052 ┆ 4 ┆ * │
│ 35 ┆ 0.9685 ┆ 0.0115 ┆ 0.9903 ┆ 0.0013 ┆ 0.055 ┆ 3 ┆ │
│ 45 ┆ 0.9711 ┆ 0.0093 ┆ 0.9926 ┆ 0.001 ┆ 0.058 ┆ 2 ┆ │
│ 55 ┆ 0.9717 ┆ 0.0093 ┆ 0.993 ┆ 0.001 ┆ 0.061 ┆ 1 ┆ │
└──────────────┴────────────┴──────────┴─────────────┴───────────┴──────────┴──────┴──────────┘
Schlussfolgerung#
Die Ein-Standardfehler-Regel hilft uns, ein einfacheres Modell (weniger PCA-Komponenten) auszuwählen und gleichzeitig eine Leistung beizubehalten, die statistisch mit dem besten Modell vergleichbar ist. Dieser Ansatz kann helfen, Überanpassung zu verhindern und die Modellinterpretierbarkeit und Effizienz zu verbessern.
In diesem Beispiel haben wir gesehen, wie diese Regel mit einem benutzerdefinierten Refit-Callable mit GridSearchCV implementiert wird.
Wichtige Erkenntnisse
Die Ein-Standardfehler-Regel liefert eine gute Faustregel zur Auswahl einfacherer Modelle
Benutzerdefinierte Refit-Callables in
GridSearchCVermöglichen flexible Modell-AuswahlstrategienDie Visualisierung von Trainings- und Testpunktzahlen hilft bei der Identifizierung potenzieller Überanpassung
Dieser Ansatz kann auf andere Modellauswahl-Szenarien angewendet werden, bei denen die Ausbalancierung von Komplexität und Leistung wichtig ist, oder in Fällen, in denen eine anwendungsfallspezifische Auswahl des "besten" Modells gewünscht wird.
# Display the figure
plt.show()
Gesamtlaufzeit des Skripts: (0 Minuten 18,332 Sekunden)
Verwandte Beispiele
Pipelining: Verkettung einer PCA und einer logistischen Regression

Benutzerdefinierte Refit-Strategie einer Gitter-Suche mit Kreuzvalidierung
Rekursive Merkmalseliminierung mit Kreuzvalidierung