Hinweis
Gehen Sie zum Ende, um den vollständigen Beispielcode herunterzuladen oder um dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Imputieren fehlender Werte mit Varianten von IterativeImputer#
Die Klasse IterativeImputer ist sehr flexibel – sie kann mit einer Vielzahl von Schätzern verwendet werden, um Round-Robin-Regression durchzuführen, wobei jede Variable abwechselnd als Ausgabe behandelt wird.
In diesem Beispiel vergleichen wir einige Schätzer für die fehlende Merkmalsimputation mit IterativeImputer
BayesianRidge: regularisierte lineare RegressionRandomForestRegressor: Wälder von zufälligen Baumregressionenmake_pipeline(Nystroem,Ridge): eine Pipeline mit der Erweiterung eines Polynomkerns vom Grad 2 und regularisierter linearer RegressionKNeighborsRegressor: vergleichbar mit anderen KNN-Imputationsansätzen
Von besonderem Interesse ist die Fähigkeit von IterativeImputer, das Verhalten von missForest, einem beliebten Imputationspaket für R, nachzuahmen.
Beachten Sie, dass KNeighborsRegressor sich von der KNN-Imputation unterscheidet, die aus Stichproben mit fehlenden Werten lernt, indem eine Distanzmetrik verwendet wird, die fehlende Werte berücksichtigt, anstatt sie zu imputieren.
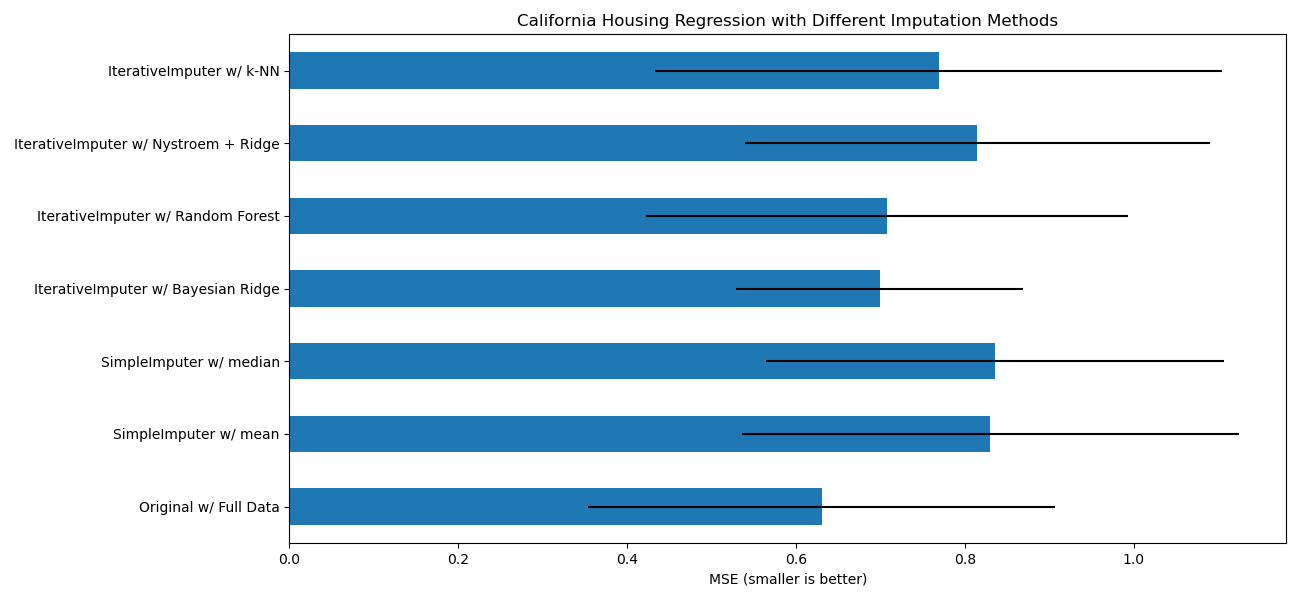
Ziel ist es, verschiedene Schätzer zu vergleichen, um festzustellen, welcher für den IterativeImputer am besten geeignet ist, wenn ein BayesianRidge-Schätzer auf dem kalifornischen Housing-Datensatz mit einem einzelnen zufällig aus jeder Zeile entfernten Wert verwendet wird.
Für dieses spezielle Muster fehlender Werte sehen wir, dass BayesianRidge und RandomForestRegressor die besten Ergebnisse liefern.
Es sollte beachtet werden, dass einige Schätzer wie HistGradientBoostingRegressor nativ mit fehlenden Merkmalen umgehen können und oft gegenüber der Erstellung von Pipelines mit komplexen und kostspieligen Strategien zur Imputation fehlender Werte empfohlen werden.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
from sklearn.datasets import fetch_california_housing
from sklearn.ensemble import RandomForestRegressor
# To use this experimental feature, we need to explicitly ask for it:
from sklearn.experimental import enable_iterative_imputer # noqa: F401
from sklearn.impute import IterativeImputer, SimpleImputer
from sklearn.kernel_approximation import Nystroem
from sklearn.linear_model import BayesianRidge, Ridge
from sklearn.model_selection import cross_val_score
from sklearn.neighbors import KNeighborsRegressor
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import RobustScaler
N_SPLITS = 5
X_full, y_full = fetch_california_housing(return_X_y=True)
# ~2k samples is enough for the purpose of the example.
# Remove the following two lines for a slower run with different error bars.
X_full = X_full[::10]
y_full = y_full[::10]
n_samples, n_features = X_full.shape
def compute_score_for(X, y, imputer=None):
# We scale data before imputation and training a target estimator,
# because our target estimator and some of the imputers assume
# that the features have similar scales.
if imputer is None:
estimator = make_pipeline(RobustScaler(), BayesianRidge())
else:
estimator = make_pipeline(RobustScaler(), imputer, BayesianRidge())
return cross_val_score(
estimator, X, y, scoring="neg_mean_squared_error", cv=N_SPLITS
)
# Estimate the score on the entire dataset, with no missing values
score_full_data = pd.DataFrame(
compute_score_for(X_full, y_full),
columns=["Full Data"],
)
# Add a single missing value to each row
rng = np.random.RandomState(0)
X_missing = X_full.copy()
y_missing = y_full
missing_samples = np.arange(n_samples)
missing_features = rng.choice(n_features, n_samples, replace=True)
X_missing[missing_samples, missing_features] = np.nan
# Estimate the score after imputation (mean and median strategies)
score_simple_imputer = pd.DataFrame()
for strategy in ("mean", "median"):
score_simple_imputer[strategy] = compute_score_for(
X_missing, y_missing, SimpleImputer(strategy=strategy)
)
# Estimate the score after iterative imputation of the missing values
# with different estimators
named_estimators = [
("Bayesian Ridge", BayesianRidge()),
(
"Random Forest",
RandomForestRegressor(
# We tuned the hyperparameters of the RandomForestRegressor to get a good
# enough predictive performance for a restricted execution time.
n_estimators=5,
max_depth=10,
bootstrap=True,
max_samples=0.5,
n_jobs=2,
random_state=0,
),
),
(
"Nystroem + Ridge",
make_pipeline(
Nystroem(kernel="polynomial", degree=2, random_state=0), Ridge(alpha=1e4)
),
),
(
"k-NN",
KNeighborsRegressor(n_neighbors=10),
),
]
score_iterative_imputer = pd.DataFrame()
# Iterative imputer is sensitive to the tolerance and
# dependent on the estimator used internally.
# We tuned the tolerance to keep this example run with limited computational
# resources while not changing the results too much compared to keeping the
# stricter default value for the tolerance parameter.
tolerances = (1e-3, 1e-1, 1e-1, 1e-2)
for (name, impute_estimator), tol in zip(named_estimators, tolerances):
score_iterative_imputer[name] = compute_score_for(
X_missing,
y_missing,
IterativeImputer(
random_state=0, estimator=impute_estimator, max_iter=40, tol=tol
),
)
scores = pd.concat(
[score_full_data, score_simple_imputer, score_iterative_imputer],
keys=["Original", "SimpleImputer", "IterativeImputer"],
axis=1,
)
# plot california housing results
fig, ax = plt.subplots(figsize=(13, 6))
means = -scores.mean()
errors = scores.std()
means.plot.barh(xerr=errors, ax=ax)
ax.set_title("California Housing Regression with Different Imputation Methods")
ax.set_xlabel("MSE (smaller is better)")
ax.set_yticks(np.arange(means.shape[0]))
ax.set_yticklabels([" w/ ".join(label) for label in means.index.tolist()])
plt.tight_layout(pad=1)
plt.show()
Gesamtlaufzeit des Skripts: (0 Minuten 6,116 Sekunden)
Verwandte Beispiele

Fehlende Werte imputieren, bevor ein Schätzer erstellt wird