Hinweis
Gehe zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Faktorenanalyse (mit Rotation) zur Visualisierung von Mustern#
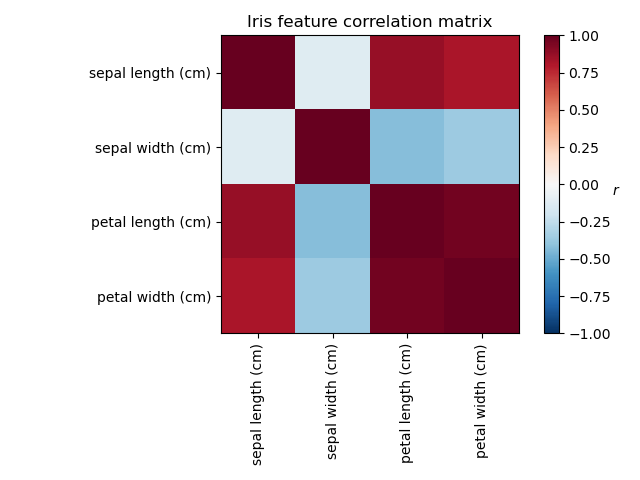
Bei der Untersuchung des Iris-Datensatzes stellen wir fest, dass Sepal Länge, Petal Länge und Petal Breite stark korreliert sind. Sepal Breite ist weniger redundant. Matrixdekompositionstechniken können diese latenten Muster aufdecken. Das Anwenden von Rotationen auf die resultierenden Komponenten verbessert den prädiktiven Wert des abgeleiteten latenten Raums nicht grundsätzlich, kann aber helfen, deren Struktur zu visualisieren. Hier findet beispielsweise die Varimax-Rotation, die durch Maximierung der quadrierten Varianzen der Gewichte ermittelt wird, eine Struktur, bei der die zweite Komponente nur positiv auf die Sepalbreite lädt.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
import matplotlib.pyplot as plt
import numpy as np
from sklearn.datasets import load_iris
from sklearn.decomposition import PCA, FactorAnalysis
from sklearn.preprocessing import StandardScaler
Iris-Daten laden
data = load_iris()
X = StandardScaler().fit_transform(data["data"])
feature_names = data["feature_names"]
Kovarianz von Iris-Merkmalen plotten
ax = plt.axes()
im = ax.imshow(np.corrcoef(X.T), cmap="RdBu_r", vmin=-1, vmax=1)
ax.set_xticks([0, 1, 2, 3])
ax.set_xticklabels(list(feature_names), rotation=90)
ax.set_yticks([0, 1, 2, 3])
ax.set_yticklabels(list(feature_names))
plt.colorbar(im).ax.set_ylabel("$r$", rotation=0)
ax.set_title("Iris feature correlation matrix")
plt.tight_layout()
Faktorenanalyse mit Varimax-Rotation durchführen
n_comps = 2
methods = [
("PCA", PCA()),
("Unrotated FA", FactorAnalysis()),
("Varimax FA", FactorAnalysis(rotation="varimax")),
]
fig, axes = plt.subplots(ncols=len(methods), figsize=(10, 8), sharey=True)
for ax, (method, fa) in zip(axes, methods):
fa.set_params(n_components=n_comps)
fa.fit(X)
components = fa.components_.T
print("\n\n %s :\n" % method)
print(components)
vmax = np.abs(components).max()
ax.imshow(components, cmap="RdBu_r", vmax=vmax, vmin=-vmax)
ax.set_yticks(np.arange(len(feature_names)))
ax.set_yticklabels(feature_names)
ax.set_title(str(method))
ax.set_xticks([0, 1])
ax.set_xticklabels(["Comp. 1", "Comp. 2"])
fig.suptitle("Factors")
plt.tight_layout()
plt.show()
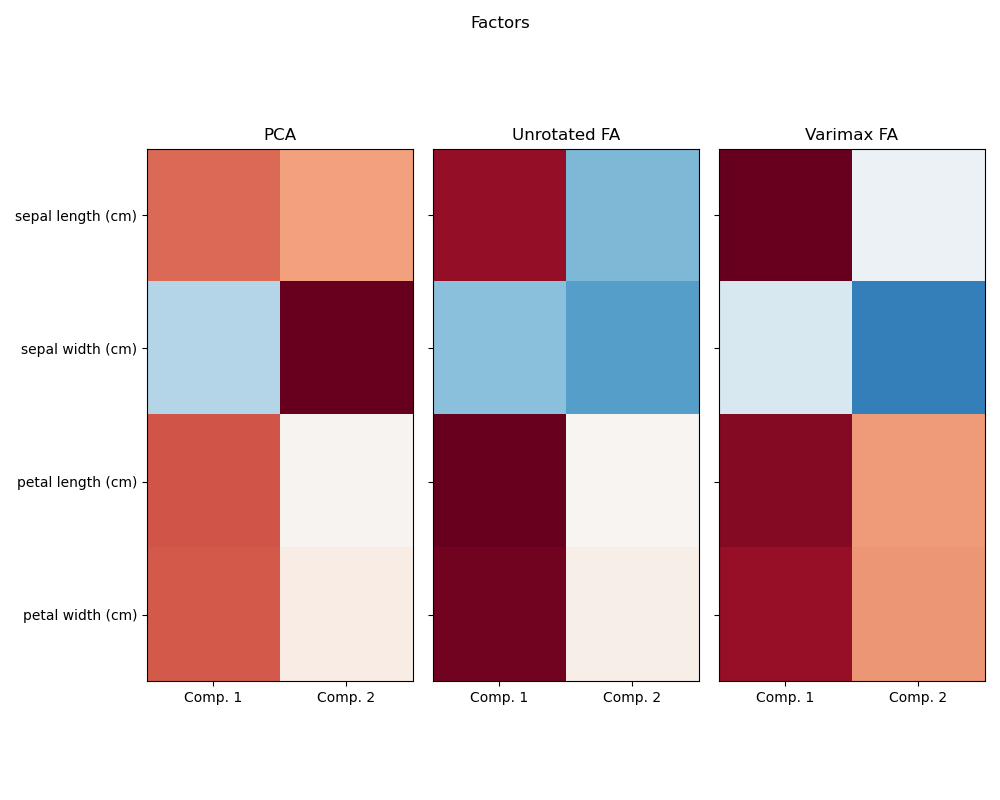
PCA :
[[ 0.52106591 0.37741762]
[-0.26934744 0.92329566]
[ 0.5804131 0.02449161]
[ 0.56485654 0.06694199]]
Unrotated FA :
[[ 0.88096009 -0.4472869 ]
[-0.41691605 -0.55390036]
[ 0.99918858 0.01915283]
[ 0.96228895 0.05840206]]
Varimax FA :
[[ 0.98633022 -0.05752333]
[-0.16052385 -0.67443065]
[ 0.90809432 0.41726413]
[ 0.85857475 0.43847489]]
Gesamtlaufzeit des Skripts: (0 Minuten 0,389 Sekunden)
Verwandte Beispiele
Principal Component Analysis (PCA) auf dem Iris-Datensatz
Vergleich von LDA und PCA 2D-Projektion des Iris-Datensatzes
Modellauswahl mit Probabilistischem PCA und Faktorenanalyse (FA)
