Hinweis
Gehen Sie zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
FastICA auf 2D-Punktwolken#
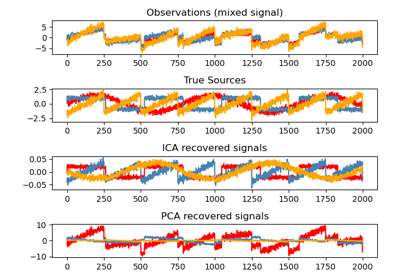
Dieses Beispiel veranschaulicht visuell im Merkmalsraum einen Vergleich der Ergebnisse zweier verschiedener Komponententechniken.
Independent Component Analysis (ICA) vs Principal Component Analysis (PCA).
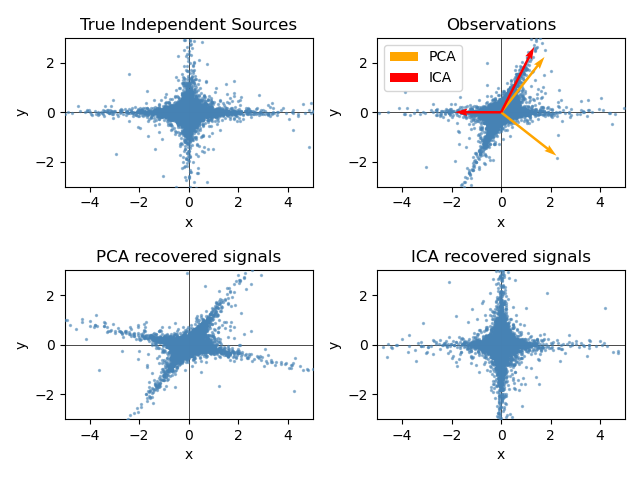
Die Darstellung von ICA im Merkmalsraum vermittelt die Sichtweise von „geometrischer ICA“: ICA ist ein Algorithmus, der Richtungen im Merkmalsraum findet, die Projektionen mit hoher Nicht-Gaußsität entsprechen. Diese Richtungen müssen im ursprünglichen Merkmalsraum nicht orthogonal sein, aber sie sind im geweißten Merkmalsraum orthogonal, in dem alle Richtungen der gleichen Varianz entsprechen.
PCA hingegen findet orthogonale Richtungen im Rohmerkmalsraum, die Richtungen entsprechen, die die maximale Varianz erklären.
Hier simulieren wir unabhängige Quellen mithilfe eines stark nicht-gaußschen Prozesses, einer Student-T-Verteilung mit wenigen Freiheitsgraden (obere linke Abbildung). Wir mischen sie, um Beobachtungen zu erzeugen (obere rechte Abbildung). Im Rohbeobachtungsraum werden die von PCA identifizierten Richtungen durch orangefarbene Vektoren dargestellt. Wir stellen das Signal im PCA-Raum dar, nach dem Weißen um die Varianz, die den PCA-Vektoren entspricht (unten links). Das Ausführen von ICA entspricht dem Finden einer Drehung in diesem Raum, um die Richtungen größter Nicht-Gaußsität zu identifizieren (unten rechts).
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Beispieldaten generieren#
import numpy as np
from sklearn.decomposition import PCA, FastICA
rng = np.random.RandomState(42)
S = rng.standard_t(1.5, size=(20000, 2))
S[:, 0] *= 2.0
# Mix data
A = np.array([[1, 1], [0, 2]]) # Mixing matrix
X = np.dot(S, A.T) # Generate observations
pca = PCA()
S_pca_ = pca.fit(X).transform(X)
ica = FastICA(random_state=rng, whiten="arbitrary-variance")
S_ica_ = ica.fit(X).transform(X) # Estimate the sources
Ergebnisse plotten#
import matplotlib.pyplot as plt
def plot_samples(S, axis_list=None):
plt.scatter(
S[:, 0], S[:, 1], s=2, marker="o", zorder=10, color="steelblue", alpha=0.5
)
if axis_list is not None:
for axis, color, label in axis_list:
x_axis, y_axis = axis / axis.std()
plt.quiver(
(0, 0),
(0, 0),
x_axis,
y_axis,
zorder=11,
width=0.01,
scale=6,
color=color,
label=label,
)
plt.hlines(0, -5, 5, color="black", linewidth=0.5)
plt.vlines(0, -3, 3, color="black", linewidth=0.5)
plt.xlim(-5, 5)
plt.ylim(-3, 3)
plt.gca().set_aspect("equal")
plt.xlabel("x")
plt.ylabel("y")
plt.figure()
plt.subplot(2, 2, 1)
plot_samples(S / S.std())
plt.title("True Independent Sources")
axis_list = [(pca.components_.T, "orange", "PCA"), (ica.mixing_, "red", "ICA")]
plt.subplot(2, 2, 2)
plot_samples(X / np.std(X), axis_list=axis_list)
legend = plt.legend(loc="upper left")
legend.set_zorder(100)
plt.title("Observations")
plt.subplot(2, 2, 3)
plot_samples(S_pca_ / np.std(S_pca_))
plt.title("PCA recovered signals")
plt.subplot(2, 2, 4)
plot_samples(S_ica_ / np.std(S_ica_))
plt.title("ICA recovered signals")
plt.subplots_adjust(0.09, 0.04, 0.94, 0.94, 0.26, 0.36)
plt.tight_layout()
plt.show()
Gesamtlaufzeit des Skripts: (0 Minuten 0,360 Sekunden)
Verwandte Beispiele
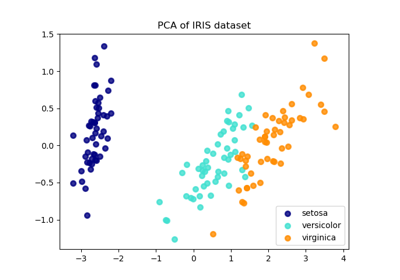
Vergleich von LDA und PCA 2D-Projektion des Iris-Datensatzes
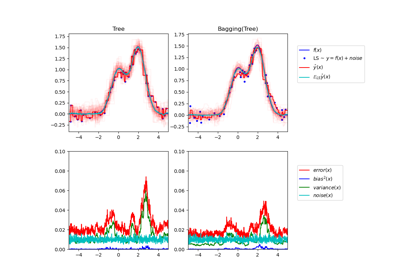
Einzelner Estimator versus Bagging: Bias-Varianz-Zerlegung
Principal Component Regression vs. Partial Least Squares Regression