Hinweis
Zum Ende springen, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Univariate Feature Selection#
Dieses Notebook ist ein Beispiel für die Verwendung der univariaten Merkmalsauswahl zur Verbesserung der Klassifikationsgenauigkeit auf einem verrauschten Datensatz.
In diesem Beispiel werden dem Iris-Datensatz einige verrauschte (nicht informative) Merkmale hinzugefügt. Eine Support Vector Machine (SVM) wird verwendet, um den Datensatz sowohl vor als auch nach der Anwendung der univariaten Merkmalsauswahl zu klassifizieren. Für jedes Merkmal plottieren wir die p-Werte für die univariate Merkmalsauswahl und die entsprechenden Gewichte der SVMs. Damit vergleichen wir die Modellgenauigkeit und untersuchen den Einfluss der univariaten Merkmalsauswahl auf die Modellgewichte.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Beispieldaten generieren#
import numpy as np
from sklearn.datasets import load_iris
from sklearn.model_selection import train_test_split
# The iris dataset
X, y = load_iris(return_X_y=True)
# Some noisy data not correlated
E = np.random.RandomState(42).uniform(0, 0.1, size=(X.shape[0], 20))
# Add the noisy data to the informative features
X = np.hstack((X, E))
# Split dataset to select feature and evaluate the classifier
X_train, X_test, y_train, y_test = train_test_split(X, y, stratify=y, random_state=0)
Univariate Merkmalsauswahl#
Univariate Merkmalsauswahl mit F-Test zur Merkmalsbewertung. Wir verwenden die Standard-Auswahlfunktion, um die vier signifikantesten Merkmale auszuwählen.
from sklearn.feature_selection import SelectKBest, f_classif
selector = SelectKBest(f_classif, k=4)
selector.fit(X_train, y_train)
scores = -np.log10(selector.pvalues_)
scores /= scores.max()
import matplotlib.pyplot as plt
X_indices = np.arange(X.shape[-1])
plt.figure(1)
plt.clf()
plt.bar(X_indices - 0.05, scores, width=0.2)
plt.title("Feature univariate score")
plt.xlabel("Feature number")
plt.ylabel(r"Univariate score ($-Log(p_{value})$)")
plt.show()
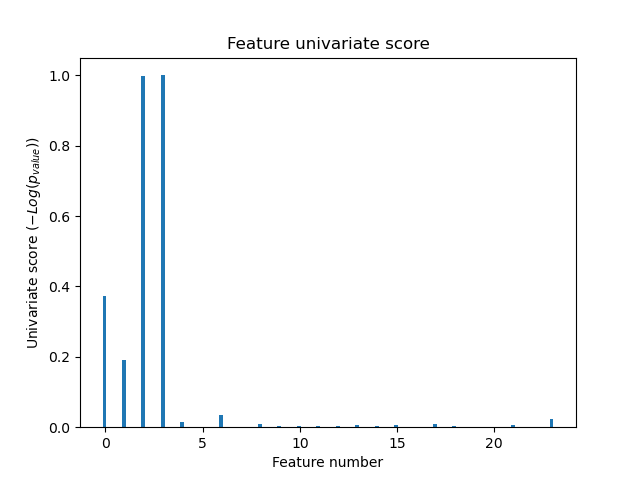
In der Gesamtheit der Merkmale sind nur 4 der ursprünglichen Merkmale signifikant. Wir können sehen, dass sie bei der univariaten Merkmalsauswahl die höchsten Werte haben.
Vergleich mit SVMs#
Ohne univariate Merkmalsauswahl
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import MinMaxScaler
from sklearn.svm import LinearSVC
clf = make_pipeline(MinMaxScaler(), LinearSVC())
clf.fit(X_train, y_train)
print(
"Classification accuracy without selecting features: {:.3f}".format(
clf.score(X_test, y_test)
)
)
svm_weights = np.abs(clf[-1].coef_).sum(axis=0)
svm_weights /= svm_weights.sum()
Classification accuracy without selecting features: 0.789
Nach univariater Merkmalsauswahl
clf_selected = make_pipeline(SelectKBest(f_classif, k=4), MinMaxScaler(), LinearSVC())
clf_selected.fit(X_train, y_train)
print(
"Classification accuracy after univariate feature selection: {:.3f}".format(
clf_selected.score(X_test, y_test)
)
)
svm_weights_selected = np.abs(clf_selected[-1].coef_).sum(axis=0)
svm_weights_selected /= svm_weights_selected.sum()
Classification accuracy after univariate feature selection: 0.868
plt.bar(
X_indices - 0.45, scores, width=0.2, label=r"Univariate score ($-Log(p_{value})$)"
)
plt.bar(X_indices - 0.25, svm_weights, width=0.2, label="SVM weight")
plt.bar(
X_indices[selector.get_support()] - 0.05,
svm_weights_selected,
width=0.2,
label="SVM weights after selection",
)
plt.title("Comparing feature selection")
plt.xlabel("Feature number")
plt.yticks(())
plt.axis("tight")
plt.legend(loc="upper right")
plt.show()
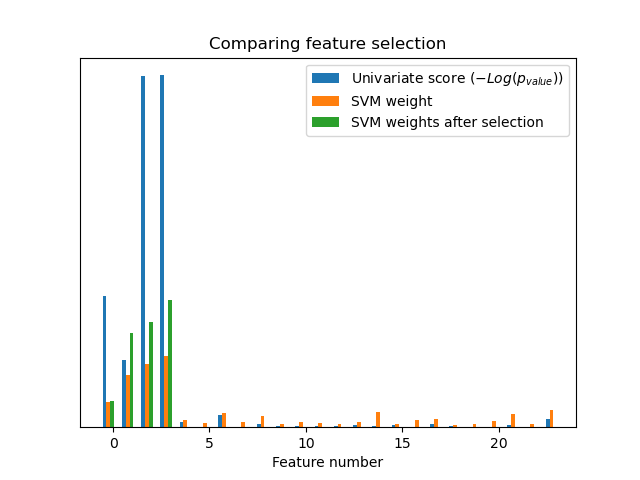
Ohne univariate Merkmalsauswahl weist die SVM ein großes Gewicht den ersten 4 ursprünglichen signifikanten Merkmalen zu, wählt aber auch viele der nicht-informativen Merkmale aus. Die Anwendung der univariaten Merkmalsauswahl vor der SVM erhöht das der signifikanten Merkmale zugewiesene SVM-Gewicht und verbessert somit die Klassifizierung.
Gesamtlaufzeit des Skripts: (0 Minuten 0,148 Sekunden)
Verwandte Beispiele