Hinweis
Gehen Sie zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Verschachtelte versus nicht verschachtelte Kreuzvalidierung#
Dieses Beispiel vergleicht nicht verschachtelte und verschachtelte Kreuzvalidierungsstrategien für einen Klassifikator auf dem Iris-Datensatz. Verschachtelte Kreuzvalidierung (CV) wird oft verwendet, um ein Modell zu trainieren, bei dem auch Hyperparameter optimiert werden müssen. Verschachtelte CV schätzt den Generalisierungsfehler des zugrundeliegenden Modells und dessen (Hyper-)Parameter-Suche. Die Auswahl von Parametern, die nicht verschachtelte CV maximieren, beeinflusst das Modell auf den Datensatz, was zu einem übermäßig optimistischen Ergebnis führt.
Modellauswahl ohne verschachtelte CV verwendet dieselben Daten, um Modellparameter abzustimmen und die Modellleistung zu bewerten. Informationen können somit in das Modell "lecken" und die Daten überanpassen. Das Ausmaß dieses Effekts hängt hauptsächlich von der Größe des Datensatzes und der Stabilität des Modells ab. Eine Analyse dieser Probleme finden Sie bei Cawley und Talbot [1].
Um dieses Problem zu vermeiden, verwendet verschachtelte CV effektiv eine Reihe von Trainings-/Validierungs-/Testdatensatz-Splits. In der inneren Schleife (hier ausgeführt von GridSearchCV) wird der Score durch Anpassen eines Modells an jeden Trainingsdatensatz approximativ maximiert, und dann durch Auswahl von (Hyper-)Parametern über den Validierungsdatensatz direkt maximiert. In der äußeren Schleife (hier in cross_val_score) wird der Generalisierungsfehler geschätzt, indem die Testdatensatz-Scores über mehrere Datensatz-Splits gemittelt werden.
Das folgende Beispiel verwendet einen Support-Vektor-Klassifikator mit einem nichtlinearen Kernel, um ein Modell mit optimierten Hyperparametern durch Grid Search zu erstellen. Wir vergleichen die Leistung von nicht verschachtelten und verschachtelten CV-Strategien, indem wir die Differenz zwischen ihren Scores berechnen.
Siehe auch
Referenzen
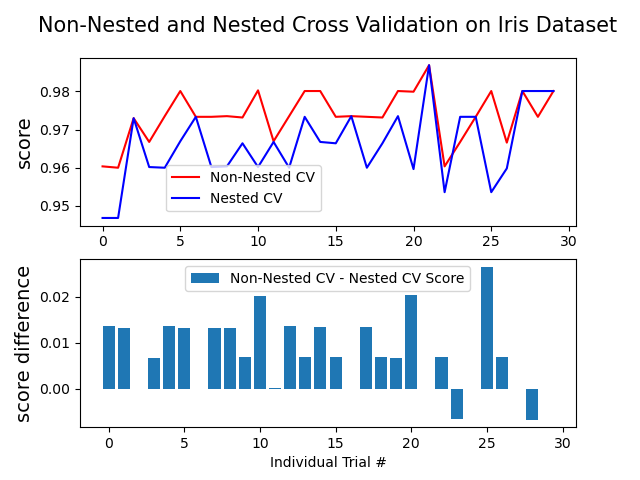
Average difference of 0.007581 with std. dev. of 0.007833.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
import numpy as np
from matplotlib import pyplot as plt
from sklearn.datasets import load_iris
from sklearn.model_selection import GridSearchCV, KFold, cross_val_score
from sklearn.svm import SVC
# Number of random trials
NUM_TRIALS = 30
# Load the dataset
iris = load_iris()
X_iris = iris.data
y_iris = iris.target
# Set up possible values of parameters to optimize over
p_grid = {"C": [1, 10, 100], "gamma": [0.01, 0.1]}
# We will use a Support Vector Classifier with "rbf" kernel
svm = SVC(kernel="rbf")
# Arrays to store scores
non_nested_scores = np.zeros(NUM_TRIALS)
nested_scores = np.zeros(NUM_TRIALS)
# Loop for each trial
for i in range(NUM_TRIALS):
# Choose cross-validation techniques for the inner and outer loops,
# independently of the dataset.
# E.g "GroupKFold", "LeaveOneOut", "LeaveOneGroupOut", etc.
inner_cv = KFold(n_splits=4, shuffle=True, random_state=i)
outer_cv = KFold(n_splits=4, shuffle=True, random_state=i)
# Non_nested parameter search and scoring
clf = GridSearchCV(estimator=svm, param_grid=p_grid, cv=outer_cv)
clf.fit(X_iris, y_iris)
non_nested_scores[i] = clf.best_score_
# Nested CV with parameter optimization
clf = GridSearchCV(estimator=svm, param_grid=p_grid, cv=inner_cv)
nested_score = cross_val_score(clf, X=X_iris, y=y_iris, cv=outer_cv)
nested_scores[i] = nested_score.mean()
score_difference = non_nested_scores - nested_scores
print(
"Average difference of {:6f} with std. dev. of {:6f}.".format(
score_difference.mean(), score_difference.std()
)
)
# Plot scores on each trial for nested and non-nested CV
plt.figure()
plt.subplot(211)
(non_nested_scores_line,) = plt.plot(non_nested_scores, color="r")
(nested_line,) = plt.plot(nested_scores, color="b")
plt.ylabel("score", fontsize="14")
plt.legend(
[non_nested_scores_line, nested_line],
["Non-Nested CV", "Nested CV"],
bbox_to_anchor=(0, 0.4, 0.5, 0),
)
plt.title(
"Non-Nested and Nested Cross Validation on Iris Dataset",
x=0.5,
y=1.1,
fontsize="15",
)
# Plot bar chart of the difference.
plt.subplot(212)
difference_plot = plt.bar(range(NUM_TRIALS), score_difference)
plt.xlabel("Individual Trial #")
plt.legend(
[difference_plot],
["Non-Nested CV - Nested CV Score"],
bbox_to_anchor=(0, 1, 0.8, 0),
)
plt.ylabel("score difference", fontsize="14")
plt.show()
Gesamtlaufzeit des Skripts: (0 Minuten 6,443 Sekunden)
Verwandte Beispiele
Übersicht über Multiklassen-Training Meta-Estimator
Visualisierung des Kreuzvalidierungsverhaltens in scikit-learn