Hinweis
Gehe zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Vergleich von linearen bayesianischen Regressoren#
Dieses Beispiel vergleicht zwei verschiedene bayesianische Regressoren
Im ersten Teil verwenden wir ein Ordinary Least Squares (OLS) Modell als Basis, um die Koeffizienten der Modelle mit den tatsächlichen Koeffizienten zu vergleichen. Danach zeigen wir, dass die Schätzung solcher Modelle durch iteratives Maximieren der marginalen Log-Likelihood der Beobachtungen erfolgt.
Im letzten Abschnitt plotten wir Vorhersagen und Unsicherheiten für die ARD- und die Bayesian Ridge-Regressionen unter Verwendung einer polynomialen Merkmalserweiterung, um eine nichtlineare Beziehung zwischen X und y anzupassen.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Robustheit der Modelle bei der Wiederherstellung der tatsächlichen Gewichte#
Synthetischen Datensatz generieren#
Wir generieren einen Datensatz, bei dem X und y linear verknüpft sind: 10 der Merkmale von X werden zur Erzeugung von y verwendet. Die anderen Merkmale sind nicht nützlich für die Vorhersage von y. Darüber hinaus generieren wir einen Datensatz, bei dem n_samples == n_features gilt. Eine solche Einstellung ist für ein OLS-Modell schwierig und führt potenziell zu beliebig großen Gewichten. Ein Priori auf die Gewichte und eine Strafe mildern das Problem. Schließlich wird Gaußsches Rauschen hinzugefügt.
from sklearn.datasets import make_regression
X, y, true_weights = make_regression(
n_samples=100,
n_features=100,
n_informative=10,
noise=8,
coef=True,
random_state=42,
)
Anpassen der Regressoren#
Wir passen nun beide bayesianischen Modelle und das OLS an, um später die Koeffizienten der Modelle zu vergleichen.
import pandas as pd
from sklearn.linear_model import ARDRegression, BayesianRidge, LinearRegression
olr = LinearRegression().fit(X, y)
brr = BayesianRidge(compute_score=True, max_iter=30).fit(X, y)
ard = ARDRegression(compute_score=True, max_iter=30).fit(X, y)
df = pd.DataFrame(
{
"Weights of true generative process": true_weights,
"ARDRegression": ard.coef_,
"BayesianRidge": brr.coef_,
"LinearRegression": olr.coef_,
}
)
Plotten der tatsächlichen und geschätzten Koeffizienten#
Nun vergleichen wir die Koeffizienten jedes Modells mit den Gewichten des wahren generativen Modells.
import matplotlib.pyplot as plt
import seaborn as sns
from matplotlib.colors import SymLogNorm
plt.figure(figsize=(10, 6))
ax = sns.heatmap(
df.T,
norm=SymLogNorm(linthresh=10e-4, vmin=-80, vmax=80),
cbar_kws={"label": "coefficients' values"},
cmap="seismic_r",
)
plt.ylabel("linear model")
plt.xlabel("coefficients")
plt.tight_layout(rect=(0, 0, 1, 0.95))
_ = plt.title("Models' coefficients")
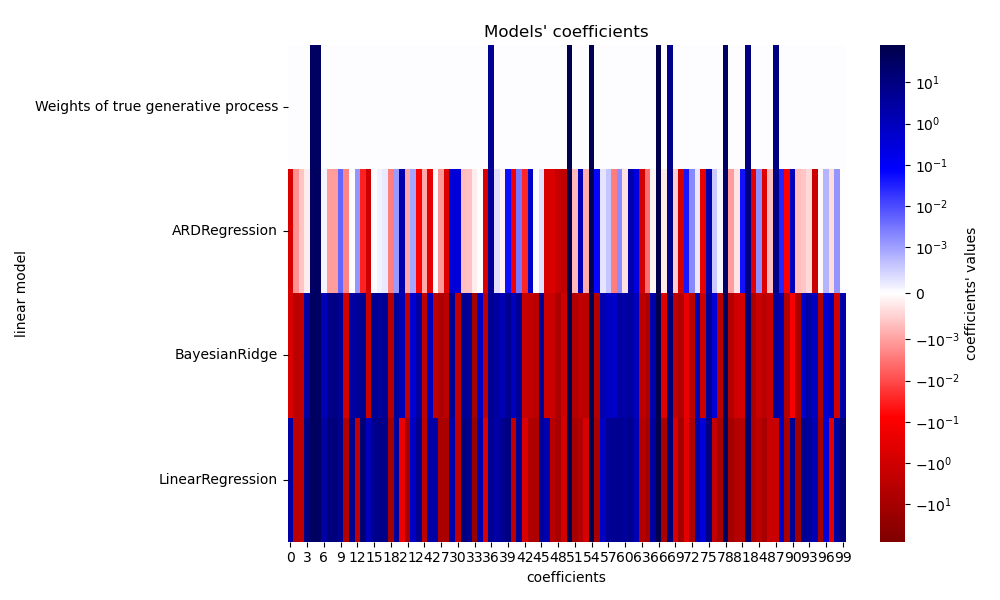
Aufgrund des hinzugefügten Rauschens rekonstruiert keines der Modelle die wahren Gewichte. Tatsächlich haben alle Modelle immer mehr als 10 Koeffizienten ungleich Null. Im Vergleich zum OLS-Schätzer sind die Koeffizienten der Bayesian Ridge-Regression leicht gegen Null verschoben, was sie stabilisiert. Die ARD-Regression liefert eine dünnere Lösung: Einige der nicht-informativen Koeffizienten werden exakt auf Null gesetzt, während andere näher an Null verschoben werden. Einige nicht-informative Koeffizienten sind immer noch vorhanden und behalten große Werte.
Plotten der marginalen Log-Likelihood#
import numpy as np
ard_scores = -np.array(ard.scores_)
brr_scores = -np.array(brr.scores_)
plt.plot(ard_scores, color="navy", label="ARD")
plt.plot(brr_scores, color="red", label="BayesianRidge")
plt.ylabel("Log-likelihood")
plt.xlabel("Iterations")
plt.xlim(1, 30)
plt.legend()
_ = plt.title("Models log-likelihood")
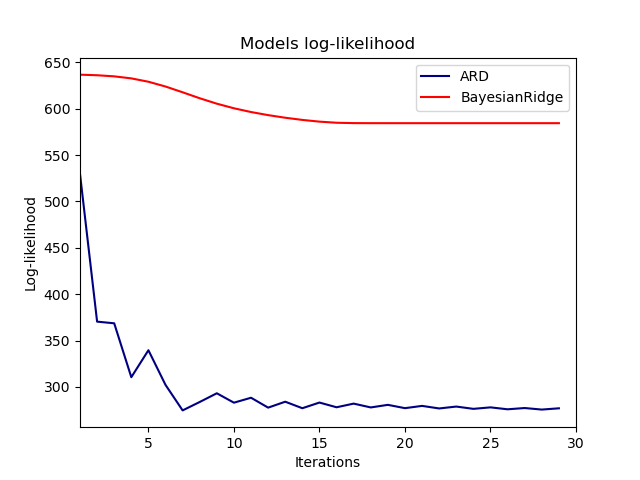
Tatsächlich minimieren beide Modelle die Log-Likelihood bis zu einem willkürlichen Grenzwert, der durch den Parameter max_iter definiert ist.
Bayesianische Regressionen mit polynomialer Merkmalserweiterung#
Generieren eines synthetischen Datensatzes#
Wir erstellen ein Ziel, das eine nichtlineare Funktion des Eingabemerkmals ist. Rauschen, das einer Standardgleichverteilung folgt, wird hinzugefügt.
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import PolynomialFeatures, StandardScaler
rng = np.random.RandomState(0)
n_samples = 110
# sort the data to make plotting easier later
X = np.sort(-10 * rng.rand(n_samples) + 10)
noise = rng.normal(0, 1, n_samples) * 1.35
y = np.sqrt(X) * np.sin(X) + noise
full_data = pd.DataFrame({"input_feature": X, "target": y})
X = X.reshape((-1, 1))
# extrapolation
X_plot = np.linspace(10, 10.4, 10)
y_plot = np.sqrt(X_plot) * np.sin(X_plot)
X_plot = np.concatenate((X, X_plot.reshape((-1, 1))))
y_plot = np.concatenate((y - noise, y_plot))
Anpassen der Regressoren#
Hier versuchen wir ein Polynom vom Grad 10, um potenziell zu überanpassen, obwohl die bayesianischen linearen Modelle die Größe der polynomialen Koeffizienten regularisieren. Da fit_intercept=True standardmäßig für ARDRegression und BayesianRidge gilt, sollte PolynomialFeatures kein zusätzliches Bias-Merkmal einführen. Durch Setzen von return_std=True geben die bayesianischen Regressoren die Standardabweichung der Posterior-Verteilung für die Modellparameter zurück.
ard_poly = make_pipeline(
PolynomialFeatures(degree=10, include_bias=False),
StandardScaler(),
ARDRegression(),
).fit(X, y)
brr_poly = make_pipeline(
PolynomialFeatures(degree=10, include_bias=False),
StandardScaler(),
BayesianRidge(),
).fit(X, y)
y_ard, y_ard_std = ard_poly.predict(X_plot, return_std=True)
y_brr, y_brr_std = brr_poly.predict(X_plot, return_std=True)
Plotten von polynomialen Regressionen mit Standardfehlern der Scores#
ax = sns.scatterplot(
data=full_data, x="input_feature", y="target", color="black", alpha=0.75
)
ax.plot(X_plot, y_plot, color="black", label="Ground Truth")
ax.plot(X_plot, y_brr, color="red", label="BayesianRidge with polynomial features")
ax.plot(X_plot, y_ard, color="navy", label="ARD with polynomial features")
ax.fill_between(
X_plot.ravel(),
y_ard - y_ard_std,
y_ard + y_ard_std,
color="navy",
alpha=0.3,
)
ax.fill_between(
X_plot.ravel(),
y_brr - y_brr_std,
y_brr + y_brr_std,
color="red",
alpha=0.3,
)
ax.legend()
_ = ax.set_title("Polynomial fit of a non-linear feature")
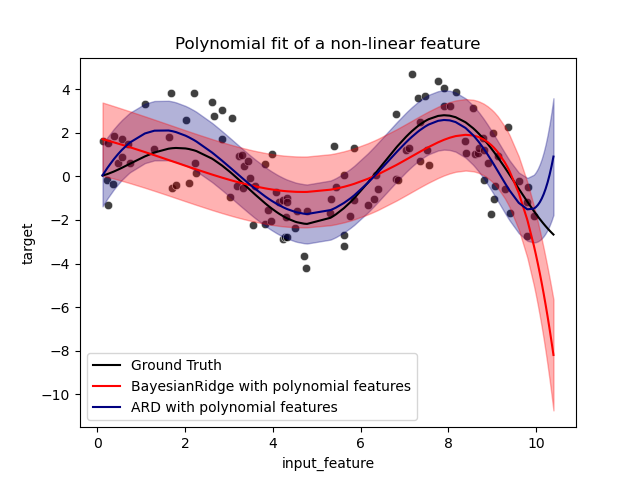
Die Fehlerbalken stellen eine Standardabweichung der vorhergesagten Gaußschen Verteilung der Abfragepunkte dar. Beachten Sie, dass die ARD-Regression die tatsächlichen Werte bei Verwendung der Standardparameter in beiden Modellen am besten erfasst, aber eine weitere Reduzierung des Hyperparameters lambda_init der Bayesian Ridge kann deren Bias reduzieren (siehe Beispiel Kurvenanpassung mit Bayesian Ridge Regression). Aufgrund der inhärenten Einschränkungen einer polynomialen Regression versagen beide Modelle schließlich bei der Extrapolation.
Gesamtlaufzeit des Skripts: (0 Minuten 0,530 Sekunden)
Verwandte Beispiele
Ridge-Koeffizienten als Funktion der L2-Regularisierung