Hinweis
Zum Ende springen, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Eine Demo der K-Means-Clusterbildung auf den handschriftlichen Zifferndaten#
In diesem Beispiel vergleichen wir die verschiedenen Initialisierungsstrategien für K-Means hinsichtlich Laufzeit und Qualität der Ergebnisse.
Da die Grundwahrheit hier bekannt ist, wenden wir auch verschiedene Cluster-Qualitätsmetriken an, um die Güte der Anpassung der Cluster-Labels an die Grundwahrheit zu beurteilen.
Ausgewertete Cluster-Qualitätsmetriken (Definitionen und Diskussionen der Metriken finden Sie unter Clustering-Leistungsbewertung)
Abkürzung |
Voller Name |
|---|---|
homo |
Homogenitätswert |
compl |
Vollständigkeitswert |
v-meas |
V-Maß |
ARI |
angepasster Randindex |
AMI |
angepasste gegenseitige Information |
silhouette |
Silhouettenkoeffizient |
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Laden des Datensatzes#
Wir beginnen mit dem Laden des digits-Datensatzes. Dieser Datensatz enthält handschriftliche Ziffern von 0 bis 9. Im Kontext der Clusterbildung möchte man Bilder gruppieren, so dass die handschriftlichen Ziffern auf dem Bild gleich sind.
import numpy as np
from sklearn.datasets import load_digits
data, labels = load_digits(return_X_y=True)
(n_samples, n_features), n_digits = data.shape, np.unique(labels).size
print(f"# digits: {n_digits}; # samples: {n_samples}; # features {n_features}")
# digits: 10; # samples: 1797; # features 64
Definieren unseres Bewertungs-Benchmarks#
Wir werden zunächst unseren Bewertungs-Benchmark erstellen. Während dieses Benchmarks beabsichtigen wir, verschiedene Initialisierungsmethoden für KMeans zu vergleichen. Unser Benchmark wird
eine Pipeline erstellen, die die Daten mit einem
StandardScalerskaliert;die Pipeline-Anpassung trainieren und zeitlich erfassen;
die Leistung der Clusterbildung anhand verschiedener Metriken messen.
from time import time
from sklearn import metrics
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import StandardScaler
def bench_k_means(kmeans, name, data, labels):
"""Benchmark to evaluate the KMeans initialization methods.
Parameters
----------
kmeans : KMeans instance
A :class:`~sklearn.cluster.KMeans` instance with the initialization
already set.
name : str
Name given to the strategy. It will be used to show the results in a
table.
data : ndarray of shape (n_samples, n_features)
The data to cluster.
labels : ndarray of shape (n_samples,)
The labels used to compute the clustering metrics which requires some
supervision.
"""
t0 = time()
estimator = make_pipeline(StandardScaler(), kmeans).fit(data)
fit_time = time() - t0
results = [name, fit_time, estimator[-1].inertia_]
# Define the metrics which require only the true labels and estimator
# labels
clustering_metrics = [
metrics.homogeneity_score,
metrics.completeness_score,
metrics.v_measure_score,
metrics.adjusted_rand_score,
metrics.adjusted_mutual_info_score,
]
results += [m(labels, estimator[-1].labels_) for m in clustering_metrics]
# The silhouette score requires the full dataset
results += [
metrics.silhouette_score(
data,
estimator[-1].labels_,
metric="euclidean",
sample_size=300,
)
]
# Show the results
formatter_result = (
"{:9s}\t{:.3f}s\t{:.0f}\t{:.3f}\t{:.3f}\t{:.3f}\t{:.3f}\t{:.3f}\t{:.3f}"
)
print(formatter_result.format(*results))
Ausführen des Benchmarks#
Wir werden drei Ansätze vergleichen:
eine Initialisierung mit
k-means++. Diese Methode ist stochastisch und wir werden die Initialisierung 4 Mal ausführen;eine zufällige Initialisierung. Diese Methode ist ebenfalls stochastisch und wir werden die Initialisierung 4 Mal ausführen;
eine Initialisierung basierend auf einer
PCA-Projektion. Tatsächlich werden wir die Komponenten derPCAzur Initialisierung von KMeans verwenden. Diese Methode ist deterministisch und eine einzige Initialisierung reicht aus.
from sklearn.cluster import KMeans
from sklearn.decomposition import PCA
print(82 * "_")
print("init\t\ttime\tinertia\thomo\tcompl\tv-meas\tARI\tAMI\tsilhouette")
kmeans = KMeans(init="k-means++", n_clusters=n_digits, n_init=4, random_state=0)
bench_k_means(kmeans=kmeans, name="k-means++", data=data, labels=labels)
kmeans = KMeans(init="random", n_clusters=n_digits, n_init=4, random_state=0)
bench_k_means(kmeans=kmeans, name="random", data=data, labels=labels)
pca = PCA(n_components=n_digits).fit(data)
kmeans = KMeans(init=pca.components_, n_clusters=n_digits, n_init=1)
bench_k_means(kmeans=kmeans, name="PCA-based", data=data, labels=labels)
print(82 * "_")
__________________________________________________________________________________
init time inertia homo compl v-meas ARI AMI silhouette
k-means++ 0.033s 69545 0.598 0.645 0.621 0.469 0.617 0.158
random 0.038s 69735 0.681 0.723 0.701 0.574 0.698 0.173
PCA-based 0.014s 69513 0.600 0.647 0.622 0.468 0.618 0.162
__________________________________________________________________________________
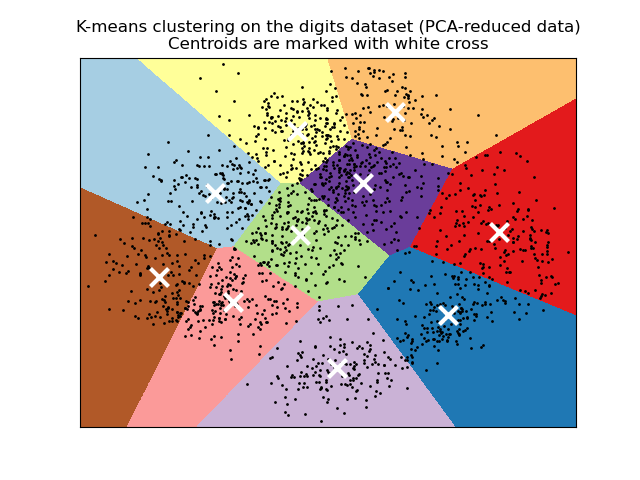
Visualisieren der Ergebnisse auf PCA-reduzierten Daten#
PCA ermöglicht die Projektion der Daten aus dem ursprünglichen 64-dimensionalen Raum in einen niedrigerdimensionalen Raum. Anschließend können wir PCA verwenden, um in einen 2-dimensionalen Raum zu projizieren und die Daten und Cluster in diesem neuen Raum zu plotten.
import matplotlib.pyplot as plt
reduced_data = PCA(n_components=2).fit_transform(data)
kmeans = KMeans(init="k-means++", n_clusters=n_digits, n_init=4)
kmeans.fit(reduced_data)
# Step size of the mesh. Decrease to increase the quality of the VQ.
h = 0.02 # point in the mesh [x_min, x_max]x[y_min, y_max].
# Plot the decision boundary. For that, we will assign a color to each
x_min, x_max = reduced_data[:, 0].min() - 1, reduced_data[:, 0].max() + 1
y_min, y_max = reduced_data[:, 1].min() - 1, reduced_data[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, h), np.arange(y_min, y_max, h))
# Obtain labels for each point in mesh. Use last trained model.
Z = kmeans.predict(np.c_[xx.ravel(), yy.ravel()])
# Put the result into a color plot
Z = Z.reshape(xx.shape)
plt.figure(1)
plt.clf()
plt.imshow(
Z,
interpolation="nearest",
extent=(xx.min(), xx.max(), yy.min(), yy.max()),
cmap=plt.cm.Paired,
aspect="auto",
origin="lower",
)
plt.plot(reduced_data[:, 0], reduced_data[:, 1], "k.", markersize=2)
# Plot the centroids as a white X
centroids = kmeans.cluster_centers_
plt.scatter(
centroids[:, 0],
centroids[:, 1],
marker="x",
s=169,
linewidths=3,
color="w",
zorder=10,
)
plt.title(
"K-means clustering on the digits dataset (PCA-reduced data)\n"
"Centroids are marked with white cross"
)
plt.xlim(x_min, x_max)
plt.ylim(y_min, y_max)
plt.xticks(())
plt.yticks(())
plt.show()
Gesamtlaufzeit des Skripts: (0 Minuten 4,309 Sekunden)
Verwandte Beispiele
Vergleich der K-Means und MiniBatchKMeans Clustering-Algorithmen
Empirische Auswertung des Einflusses der K-Means Initialisierung