Hinweis
Gehen Sie zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Hauptkomponentenanalyse (PCA) für den Iris-Datensatz#
Dieses Beispiel zeigt eine bekannte Zerfallstechnik, die als Hauptkomponentenanalyse (PCA) auf dem Iris-Datensatz bekannt ist.
Dieser Datensatz besteht aus 4 Merkmalen: Kelchblattlänge, Kelchblattbreite, Blütenblattlänge, Blütenblattbreite. Wir verwenden PCA, um diesen 4-dimensionalen Merkmalsraum in einen 3-dimensionalen Raum zu projizieren.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Laden des Iris-Datensatzes#
Der Iris-Datensatz ist direkt als Teil von scikit-learn verfügbar. Er kann mit der Funktion load_iris geladen werden. Mit den Standardparametern wird ein Bunch-Objekt zurückgegeben, das die Daten, die Zielwerte, die Merkmalsnamen und die Zielnamen enthält.
dict_keys(['data', 'target', 'frame', 'target_names', 'DESCR', 'feature_names', 'filename', 'data_module'])
Plot von Merkmalspaaren des Iris-Datensatzes#
Lassen Sie uns zuerst die Paare von Merkmalen des Iris-Datensatzes plotten.
import seaborn as sns
# Rename classes using the iris target names
iris.frame["target"] = iris.target_names[iris.target]
_ = sns.pairplot(iris.frame, hue="target")
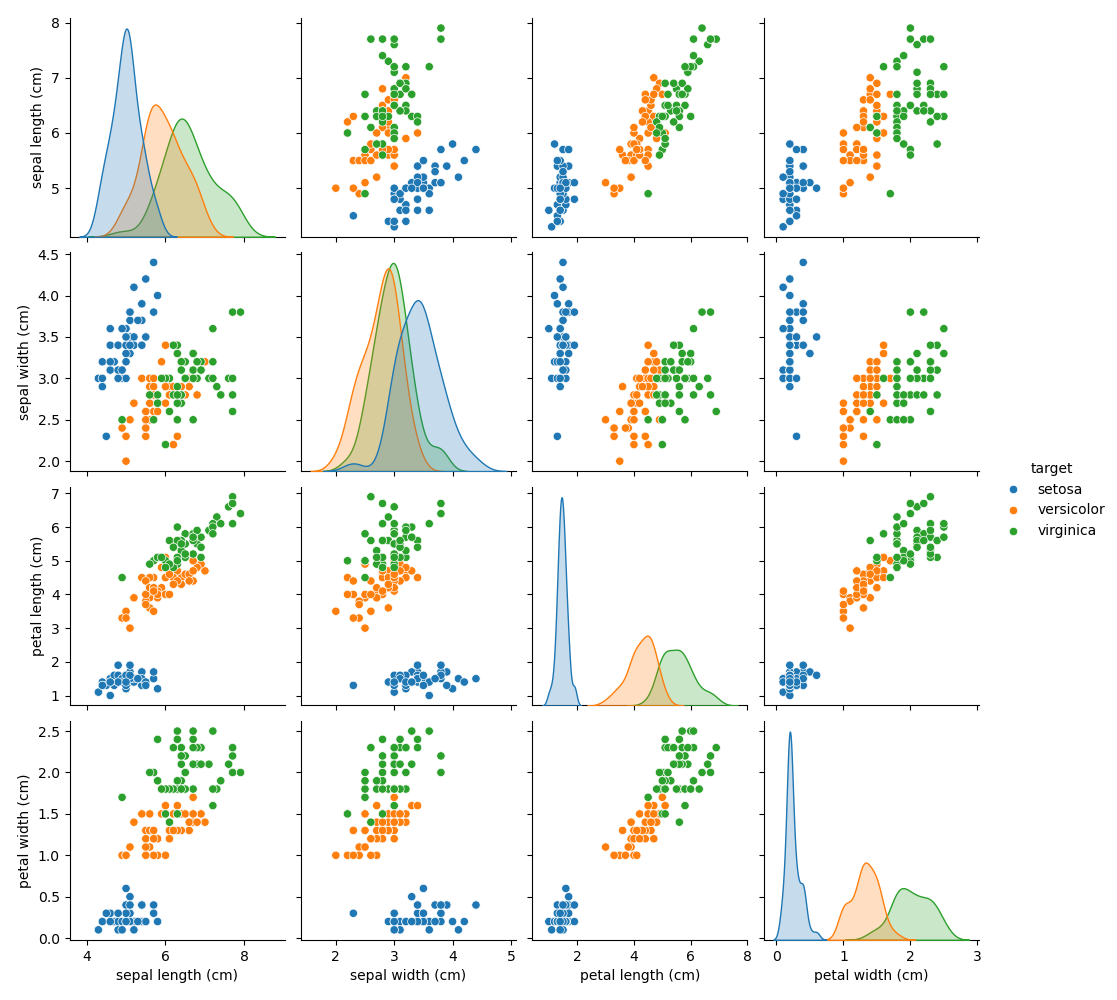
Jeder Datenpunkt in jedem Streudiagramm bezieht sich auf eine der 150 Iris-Blumen im Datensatz, wobei die Farbe ihren jeweiligen Typ angibt (Setosa, Versicolor und Virginica).
Sie können bereits ein Muster bezüglich des Typs Setosa erkennen, der anhand seiner kurzen und breiten Kelchblätter leicht identifizierbar ist. Allein unter Berücksichtigung dieser beiden Dimensionen, Kelchblattbreite und -länge, gibt es immer noch Überschneidungen zwischen den Typen Versicolor und Virginica.
Die Diagonale des Plots zeigt die Verteilung jedes Merkmals. Wir beobachten, dass die Blütenblattbreite und die Blütenblattlänge die diskriminantesten Merkmale für die drei Typen sind.
Plot einer PCA-Darstellung#
Lassen Sie uns eine Hauptkomponentenanalyse (PCA) auf den Iris-Datensatz anwenden und dann die Iris-Blumen anhand der ersten drei Hauptkomponenten plotten. Dies wird uns ermöglichen, die drei Typen besser zu unterscheiden!
import matplotlib.pyplot as plt
# unused but required import for doing 3d projections with matplotlib < 3.2
import mpl_toolkits.mplot3d # noqa: F401
from sklearn.decomposition import PCA
fig = plt.figure(1, figsize=(8, 6))
ax = fig.add_subplot(111, projection="3d", elev=-150, azim=110)
X_reduced = PCA(n_components=3).fit_transform(iris.data)
scatter = ax.scatter(
X_reduced[:, 0],
X_reduced[:, 1],
X_reduced[:, 2],
c=iris.target,
s=40,
)
ax.set(
title="First three principal components",
xlabel="1st Principal Component",
ylabel="2nd Principal Component",
zlabel="3rd Principal Component",
)
ax.xaxis.set_ticklabels([])
ax.yaxis.set_ticklabels([])
ax.zaxis.set_ticklabels([])
# Add a legend
legend1 = ax.legend(
scatter.legend_elements()[0],
iris.target_names.tolist(),
loc="upper right",
title="Classes",
)
ax.add_artist(legend1)
plt.show()
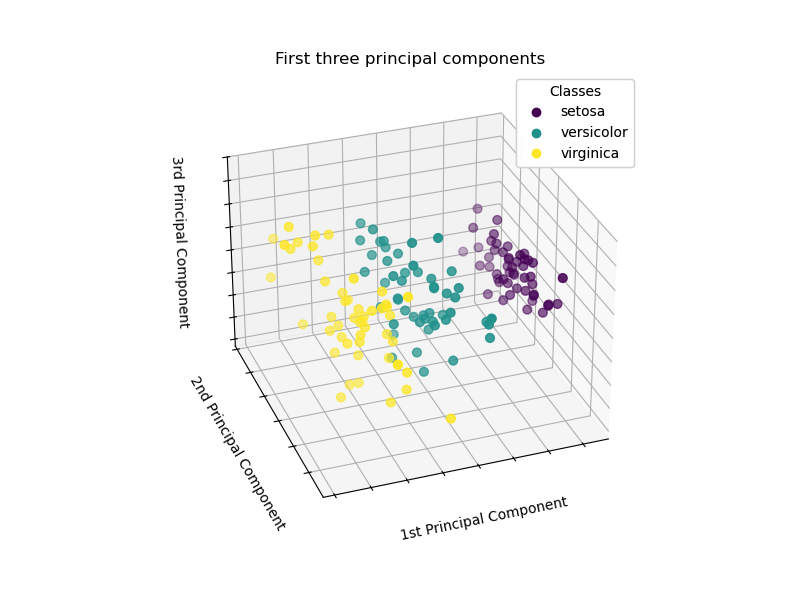
PCA erstellt 3 neue Merkmale, die eine lineare Kombination der 4 ursprünglichen Merkmale sind. Darüber hinaus maximiert diese Transformation die Varianz. Mit dieser Transformation können wir jede Spezies anhand der ersten Hauptkomponente identifizieren.
Gesamtlaufzeit des Skripts: (0 Minuten 1,923 Sekunden)
Verwandte Beispiele
Vergleich von LDA und PCA 2D-Projektion des Iris-Datensatzes
Entscheidungsfläche von Entscheidungsbäumen, trainiert auf dem Iris-Datensatz, plotten