Hinweis
Zum Ende springen, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Underfitting vs. Overfitting#
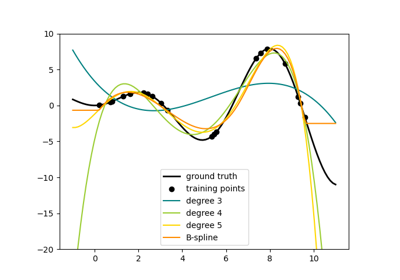
Dieses Beispiel demonstriert die Probleme von Underfitting und Overfitting und wie wir lineare Regression mit polynomialen Merkmalen verwenden können, um nichtlineare Funktionen zu approximieren. Die Grafik zeigt die Funktion, die wir approximieren wollen, welche ein Teil der Kosinusfunktion ist. Zusätzlich werden die Stichproben aus der realen Funktion und die Approximationen verschiedener Modelle angezeigt. Die Modelle haben polynomiale Merkmale unterschiedlichen Grades. Wir können sehen, dass eine lineare Funktion (Polynom vom Grad 1) nicht ausreicht, um die Trainingsstichproben anzupassen. Dies wird als Underfitting bezeichnet. Ein Polynom vom Grad 4 approximiert die wahre Funktion nahezu perfekt. Bei höheren Graden wird das Modell jedoch die Trainingsdaten overfitten, d. h. es lernt das Rauschen der Trainingsdaten. Wir bewerten Overfitting / Underfitting quantitativ mithilfe von Kreuzvalidierung. Wir berechnen den mittleren quadratischen Fehler (MSE) auf dem Validierungsdatensatz. Je höher dieser ist, desto unwahrscheinlicher ist es, dass das Modell korrekt von den Trainingsdaten generalisiert.

# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
import matplotlib.pyplot as plt
import numpy as np
from sklearn.linear_model import LinearRegression
from sklearn.model_selection import cross_val_score
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import PolynomialFeatures
def true_fun(X):
return np.cos(1.5 * np.pi * X)
np.random.seed(0)
n_samples = 30
degrees = [1, 4, 15]
X = np.sort(np.random.rand(n_samples))
y = true_fun(X) + np.random.randn(n_samples) * 0.1
plt.figure(figsize=(14, 5))
for i in range(len(degrees)):
ax = plt.subplot(1, len(degrees), i + 1)
plt.setp(ax, xticks=(), yticks=())
polynomial_features = PolynomialFeatures(degree=degrees[i], include_bias=False)
linear_regression = LinearRegression()
pipeline = Pipeline(
[
("polynomial_features", polynomial_features),
("linear_regression", linear_regression),
]
)
pipeline.fit(X[:, np.newaxis], y)
# Evaluate the models using crossvalidation
scores = cross_val_score(
pipeline, X[:, np.newaxis], y, scoring="neg_mean_squared_error", cv=10
)
X_test = np.linspace(0, 1, 100)
plt.plot(X_test, pipeline.predict(X_test[:, np.newaxis]), label="Model")
plt.plot(X_test, true_fun(X_test), label="True function")
plt.scatter(X, y, edgecolor="b", s=20, label="Samples")
plt.xlabel("x")
plt.ylabel("y")
plt.xlim((0, 1))
plt.ylim((-2, 2))
plt.legend(loc="best")
plt.title(
"Degree {}\nMSE = {:.2e}(+/- {:.2e})".format(
degrees[i], -scores.mean(), scores.std()
)
)
plt.show()
Gesamtlaufzeit des Skripts: (0 Minuten 0,162 Sekunden)
Verwandte Beispiele
Klassifikationsgrenzen mit verschiedenen SVM-Kernen plotten