Hinweis
Gehen Sie zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Visualisierung des Kreuzvalidierungsverhaltens in scikit-learn#
Die Wahl des richtigen Kreuzvalidierungsobjekts ist ein entscheidender Teil der ordnungsgemäßen Anpassung eines Modells. Es gibt viele Möglichkeiten, Daten in Trainings- und Testdatensätze aufzuteilen, um Modell-Overfitting zu vermeiden, die Anzahl der Gruppen in Testdatensätzen zu standardisieren usw.
Dieses Beispiel visualisiert das Verhalten mehrerer gängiger scikit-learn-Objekte zum Vergleich.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
import matplotlib.pyplot as plt
import numpy as np
from matplotlib.patches import Patch
from sklearn.model_selection import (
GroupKFold,
GroupShuffleSplit,
KFold,
ShuffleSplit,
StratifiedGroupKFold,
StratifiedKFold,
StratifiedShuffleSplit,
TimeSeriesSplit,
)
rng = np.random.RandomState(1338)
cmap_data = plt.cm.Paired
cmap_cv = plt.cm.coolwarm
n_splits = 4
Visualisierung unserer Daten#
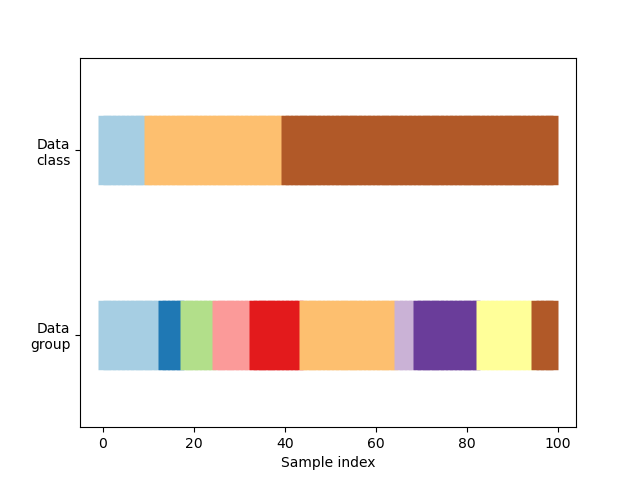
Zuerst müssen wir die Struktur unserer Daten verstehen. Sie enthält 100 zufällig generierte Eingabedatenpunkte, 3 Klassen, die ungleichmäßig auf die Datenpunkte verteilt sind, und 10 "Gruppen", die gleichmäßig auf die Datenpunkte verteilt sind.
Wie wir sehen werden, tun einige Kreuzvalidierungsobjekte bestimmte Dinge mit gelabelten Daten, andere verhalten sich anders mit gruppierten Daten, und wieder andere verwenden diese Informationen nicht.
Um zu beginnen, visualisieren wir unsere Daten.
# Generate the class/group data
n_points = 100
X = rng.randn(100, 10)
percentiles_classes = [0.1, 0.3, 0.6]
y = np.hstack([[ii] * int(100 * perc) for ii, perc in enumerate(percentiles_classes)])
# Generate uneven groups
group_prior = rng.dirichlet([2] * 10)
groups = np.repeat(np.arange(10), rng.multinomial(100, group_prior))
def visualize_groups(classes, groups, name):
# Visualize dataset groups
fig, ax = plt.subplots()
ax.scatter(
range(len(groups)),
[0.5] * len(groups),
c=groups,
marker="_",
lw=50,
cmap=cmap_data,
)
ax.scatter(
range(len(groups)),
[3.5] * len(groups),
c=classes,
marker="_",
lw=50,
cmap=cmap_data,
)
ax.set(
ylim=[-1, 5],
yticks=[0.5, 3.5],
yticklabels=["Data\ngroup", "Data\nclass"],
xlabel="Sample index",
)
visualize_groups(y, groups, "no groups")
Definieren einer Funktion zur Visualisierung des Kreuzvalidierungsverhaltens#
Wir definieren eine Funktion, die es uns ermöglicht, das Verhalten jedes Kreuzvalidierungsobjekts zu visualisieren. Wir werden 4 Aufteilungen der Daten durchführen. Bei jeder Aufteilung visualisieren wir die für den Trainingsdatensatz (in Blau) und den Testdatensatz (in Rot) ausgewählten Indizes.
def plot_cv_indices(cv, X, y, group, ax, n_splits, lw=10):
"""Create a sample plot for indices of a cross-validation object."""
use_groups = "Group" in type(cv).__name__
groups = group if use_groups else None
# Generate the training/testing visualizations for each CV split
for ii, (tr, tt) in enumerate(cv.split(X=X, y=y, groups=groups)):
# Fill in indices with the training/test groups
indices = np.array([np.nan] * len(X))
indices[tt] = 1
indices[tr] = 0
# Visualize the results
ax.scatter(
range(len(indices)),
[ii + 0.5] * len(indices),
c=indices,
marker="_",
lw=lw,
cmap=cmap_cv,
vmin=-0.2,
vmax=1.2,
)
# Plot the data classes and groups at the end
ax.scatter(
range(len(X)), [ii + 1.5] * len(X), c=y, marker="_", lw=lw, cmap=cmap_data
)
ax.scatter(
range(len(X)), [ii + 2.5] * len(X), c=group, marker="_", lw=lw, cmap=cmap_data
)
# Formatting
yticklabels = list(range(n_splits)) + ["class", "group"]
ax.set(
yticks=np.arange(n_splits + 2) + 0.5,
yticklabels=yticklabels,
xlabel="Sample index",
ylabel="CV iteration",
ylim=[n_splits + 2.2, -0.2],
xlim=[0, 100],
)
ax.set_title("{}".format(type(cv).__name__), fontsize=15)
return ax
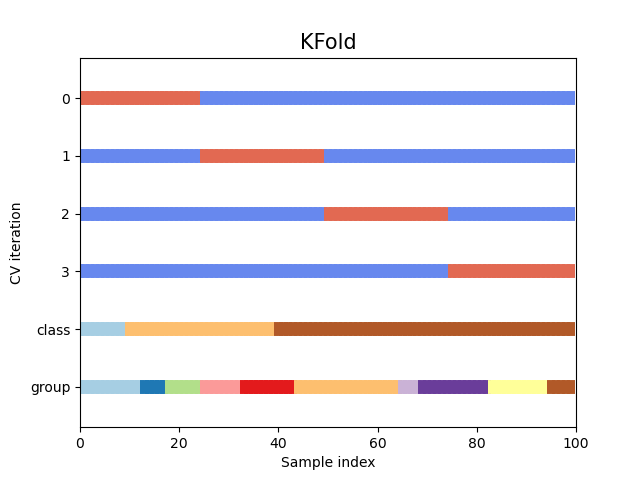
Sehen wir uns an, wie es für das KFold-Kreuzvalidierungsobjekt aussieht
fig, ax = plt.subplots()
cv = KFold(n_splits)
plot_cv_indices(cv, X, y, groups, ax, n_splits)
<Axes: title={'center': 'KFold'}, xlabel='Sample index', ylabel='CV iteration'>
Wie Sie sehen können, berücksichtigt der KFold-Kreuzvalidierungs-Iterator standardmäßig weder die Datenpunktklasse noch die Gruppe. Wir können dies ändern, indem wir entweder
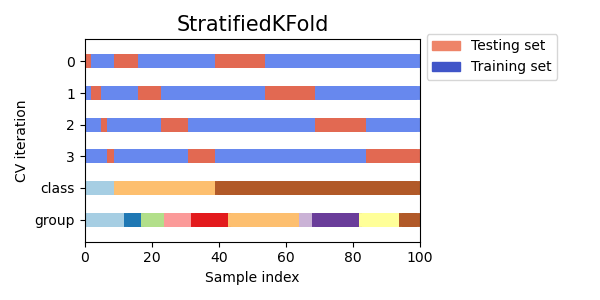
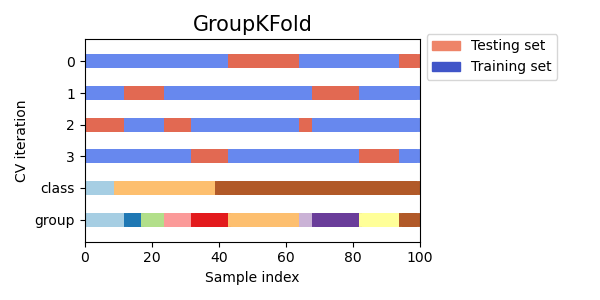
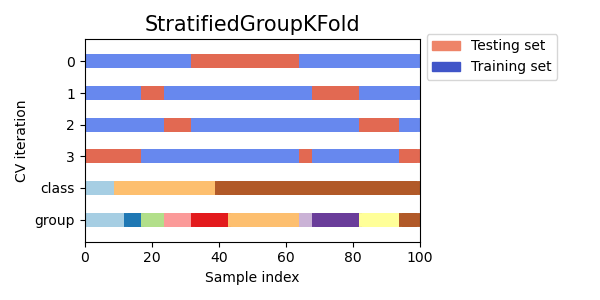
StratifiedKFoldverwenden, um den Prozentsatz der Stichproben für jede Klasse zu erhalten.GroupKFoldverwenden, um sicherzustellen, dass dieselbe Gruppe nicht in zwei verschiedenen Folds erscheint.StratifiedGroupKFoldverwenden, um die Einschränkung vonGroupKFoldbeizubehalten und gleichzeitig zu versuchen, stratifizierte Folds zurückzugeben.
cvs = [StratifiedKFold, GroupKFold, StratifiedGroupKFold]
for cv in cvs:
fig, ax = plt.subplots(figsize=(6, 3))
plot_cv_indices(cv(n_splits), X, y, groups, ax, n_splits)
ax.legend(
[Patch(color=cmap_cv(0.8)), Patch(color=cmap_cv(0.02))],
["Testing set", "Training set"],
loc=(1.02, 0.8),
)
# Make the legend fit
plt.tight_layout()
fig.subplots_adjust(right=0.7)
Als Nächstes visualisieren wir dieses Verhalten für eine Reihe von CV-Iteratoren.
Visualisierung von Kreuzvalidierungsindizes für viele CV-Objekte#
Vergleichen wir visuell das Kreuzvalidierungsverhalten vieler scikit-learn-Kreuzvalidierungsobjekte. Im Folgenden durchlaufen wir mehrere gängige Kreuzvalidierungsobjekte und visualisieren das Verhalten jedes einzelnen.
Beachten Sie, wie einige die Gruppen-/Informationsklassen verwenden und andere nicht.
cvs = [
KFold,
GroupKFold,
ShuffleSplit,
StratifiedKFold,
StratifiedGroupKFold,
GroupShuffleSplit,
StratifiedShuffleSplit,
TimeSeriesSplit,
]
for cv in cvs:
this_cv = cv(n_splits=n_splits)
fig, ax = plt.subplots(figsize=(6, 3))
plot_cv_indices(this_cv, X, y, groups, ax, n_splits)
ax.legend(
[Patch(color=cmap_cv(0.8)), Patch(color=cmap_cv(0.02))],
["Testing set", "Training set"],
loc=(1.02, 0.8),
)
# Make the legend fit
plt.tight_layout()
fig.subplots_adjust(right=0.7)
plt.show()
Gesamtlaufzeit des Skripts: (0 Minuten 0,998 Sekunden)
Verwandte Beispiele
Receiver Operating Characteristic (ROC) mit Kreuzvalidierung
Rekursive Merkmalseliminierung mit Kreuzvalidierung
Verschachtelte vs. nicht verschachtelte Kreuzvalidierung