Hinweis
Gehen Sie zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Fehlende Werte imputieren, bevor ein Schätzer aufgebaut wird#
Fehlende Werte können durch den Mittelwert, den Median oder den häufigsten Wert mithilfe des einfachen SimpleImputer ersetzt werden.
In diesem Beispiel werden wir verschiedene Imputationstechniken untersuchen
Imputation durch den konstanten Wert 0
Imputation durch den Mittelwert jeder Merkmalvariable
k-Nächste-Nachbarn-Imputation
Iterative Imputation
In allen Fällen fügen wir für jede Merkmalvariable eine neue Variable hinzu, die das Fehlen angibt.
Wir werden zwei Datensätze verwenden: den Diabetes-Datensatz, der aus 10 Merkmalvariablen von Diabetespatienten besteht, mit dem Ziel, das Fortschreiten der Krankheit vorherzusagen, und den California Housing-Datensatz, bei dem das Ziel der mittlere Hauswert für kalifornische Bezirke ist.
Da keiner dieser Datensätze fehlende Werte aufweist, werden wir einige Werte entfernen, um neue Versionen mit künstlich fehlenden Daten zu erstellen. Die Leistung von RandomForestRegressor auf dem vollständigen Originaldatensatz wird dann mit der Leistung auf den veränderten Datensätzen verglichen, bei denen die künstlich fehlenden Werte mit verschiedenen Techniken imputiert wurden.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Daten herunterladen und Datensätze mit fehlenden Werten erstellen#
Zuerst laden wir die beiden Datensätze herunter. Der Diabetes-Datensatz ist bei scikit-learn enthalten. Er hat 442 Einträge mit jeweils 10 Merkmalen. Der California Housing-Datensatz ist mit 20640 Einträgen und 8 Merkmalen wesentlich größer. Er muss heruntergeladen werden. Wir verwenden nur die ersten 300 Einträge, um die Berechnungen zu beschleunigen, aber Sie können gerne den gesamten Datensatz verwenden.
import numpy as np
from sklearn.datasets import fetch_california_housing, load_diabetes
X_diabetes, y_diabetes = load_diabetes(return_X_y=True)
X_california, y_california = fetch_california_housing(return_X_y=True)
X_diabetes = X_diabetes[:300]
y_diabetes = y_diabetes[:300]
X_california = X_california[:300]
y_california = y_california[:300]
def add_missing_values(X_full, y_full, rng):
n_samples, n_features = X_full.shape
# Add missing values in 75% of the lines
missing_rate = 0.75
n_missing_samples = int(n_samples * missing_rate)
missing_samples = np.zeros(n_samples, dtype=bool)
missing_samples[:n_missing_samples] = True
rng.shuffle(missing_samples)
missing_features = rng.randint(0, n_features, n_missing_samples)
X_missing = X_full.copy()
X_missing[missing_samples, missing_features] = np.nan
y_missing = y_full.copy()
return X_missing, y_missing
rng = np.random.RandomState(42)
X_miss_diabetes, y_miss_diabetes = add_missing_values(X_diabetes, y_diabetes, rng)
X_miss_california, y_miss_california = add_missing_values(
X_california, y_california, rng
)
Fehlende Daten imputieren und bewerten#
Nun schreiben wir eine Funktion, die die Ergebnisse auf den unterschiedlich imputierten Daten bewertet, einschließlich des Falls keiner Imputation für vollständige Daten. Wir verwenden RandomForestRegressor für die Zielregression.
from sklearn.ensemble import RandomForestRegressor
# To use the experimental IterativeImputer, we need to explicitly ask for it:
from sklearn.experimental import enable_iterative_imputer # noqa: F401
from sklearn.impute import IterativeImputer, KNNImputer, SimpleImputer
from sklearn.model_selection import cross_val_score
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import RobustScaler
N_SPLITS = 4
def get_score(X, y, imputer=None):
regressor = RandomForestRegressor(random_state=0)
if imputer is not None:
estimator = make_pipeline(imputer, regressor)
else:
estimator = regressor
scores = cross_val_score(
estimator, X, y, scoring="neg_mean_squared_error", cv=N_SPLITS
)
return scores.mean(), scores.std()
x_labels = []
mses_diabetes = np.zeros(5)
stds_diabetes = np.zeros(5)
mses_california = np.zeros(5)
stds_california = np.zeros(5)
Ergebnisse schätzen#
Zuerst möchten wir die Ergebnisse auf den Originaldaten schätzen
mses_diabetes[0], stds_diabetes[0] = get_score(X_diabetes, y_diabetes)
mses_california[0], stds_california[0] = get_score(X_california, y_california)
x_labels.append("Full Data")
Fehlende Werte durch 0 ersetzen#
Nun schätzen wir die Ergebnisse auf den Daten, bei denen die fehlenden Werte durch 0 ersetzt wurden
imputer = SimpleImputer(strategy="constant", fill_value=0, add_indicator=True)
mses_diabetes[1], stds_diabetes[1] = get_score(
X_miss_diabetes, y_miss_diabetes, imputer
)
mses_california[1], stds_california[1] = get_score(
X_miss_california, y_miss_california, imputer
)
x_labels.append("Zero Imputation")
Fehlende Werte mit dem Mittelwert imputieren#
imputer = SimpleImputer(strategy="mean", add_indicator=True)
mses_diabetes[2], stds_diabetes[2] = get_score(
X_miss_diabetes, y_miss_diabetes, imputer
)
mses_california[2], stds_california[2] = get_score(
X_miss_california, y_miss_california, imputer
)
x_labels.append("Mean Imputation")
kNN-Imputation der fehlenden Werte#
KNNImputer imputiert fehlende Werte anhand des gewichteten oder ungewichteten Mittels der gewünschten Anzahl von nächsten Nachbarn. Wenn Ihre Merkmalvariablen stark unterschiedliche Skalen aufweisen (wie im California Housing-Datensatz), sollten Sie sie neu skalieren, um die Leistung möglicherweise zu verbessern.
imputer = KNNImputer(add_indicator=True)
mses_diabetes[3], stds_diabetes[3] = get_score(
X_miss_diabetes, y_miss_diabetes, imputer
)
mses_california[3], stds_california[3] = get_score(
X_miss_california, y_miss_california, make_pipeline(RobustScaler(), imputer)
)
x_labels.append("KNN Imputation")
Iterative Imputation der fehlenden Werte#
Eine weitere Option ist der IterativeImputer. Dieser verwendet Round-Robin-Regression, bei der jede Merkmalvariable mit fehlenden Werten nacheinander als Funktion anderer Merkmale modelliert wird. Wir verwenden die Standardwahl des Regressionsmodells der Klasse (BayesianRidge), um fehlende Merkmalwerte vorherzusagen. Die Leistung des Prädiktors kann durch stark unterschiedliche Skalen der Merkmale negativ beeinflusst werden, daher skalieren wir die Merkmale im California Housing-Datensatz neu.
imputer = IterativeImputer(add_indicator=True)
mses_diabetes[4], stds_diabetes[4] = get_score(
X_miss_diabetes, y_miss_diabetes, imputer
)
mses_california[4], stds_california[4] = get_score(
X_miss_california, y_miss_california, make_pipeline(RobustScaler(), imputer)
)
x_labels.append("Iterative Imputation")
mses_diabetes = mses_diabetes * -1
mses_california = mses_california * -1
Ergebnisse plotten#
Schließlich werden wir die Ergebnisse visualisieren
import matplotlib.pyplot as plt
n_bars = len(mses_diabetes)
xval = np.arange(n_bars)
colors = ["r", "g", "b", "orange", "black"]
# plot diabetes results
plt.figure(figsize=(12, 6))
ax1 = plt.subplot(121)
for j in xval:
ax1.barh(
j,
mses_diabetes[j],
xerr=stds_diabetes[j],
color=colors[j],
alpha=0.6,
align="center",
)
ax1.set_title("Imputation Techniques with Diabetes Data")
ax1.set_xlim(left=np.min(mses_diabetes) * 0.9, right=np.max(mses_diabetes) * 1.1)
ax1.set_yticks(xval)
ax1.set_xlabel("MSE")
ax1.invert_yaxis()
ax1.set_yticklabels(x_labels)
# plot california dataset results
ax2 = plt.subplot(122)
for j in xval:
ax2.barh(
j,
mses_california[j],
xerr=stds_california[j],
color=colors[j],
alpha=0.6,
align="center",
)
ax2.set_title("Imputation Techniques with California Data")
ax2.set_yticks(xval)
ax2.set_xlabel("MSE")
ax2.invert_yaxis()
ax2.set_yticklabels([""] * n_bars)
plt.show()
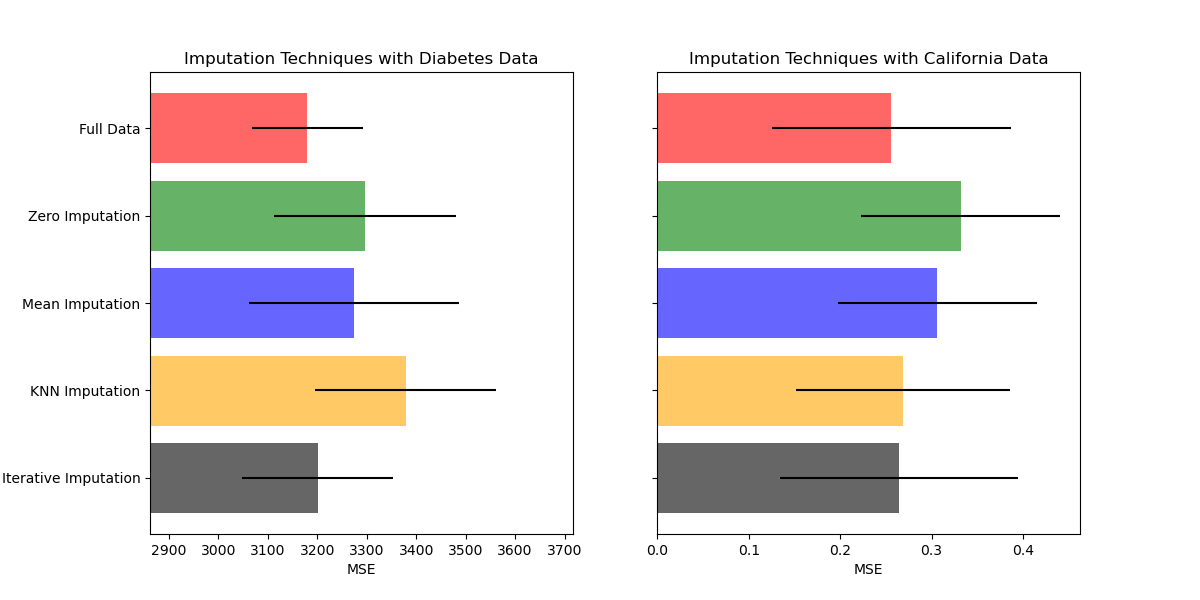
Sie können auch andere Techniken ausprobieren. Zum Beispiel ist der Median ein robusterer Schätzer für Daten mit Variablen mit hoher Amplitude, die die Ergebnisse dominieren könnten (auch bekannt als "lange Enden").
Gesamtlaufzeit des Skripts: (0 Minuten 8,208 Sekunden)
Verwandte Beispiele
Fehlende Werte mit Varianten von IterativeImputer imputieren
Analyse des Konzentrations-Prior-Typs der Variation im Bayes'schen Gaußschen Gemisch