Hinweis
Zum Ende springen, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Release Highlights für scikit-learn 1.8#
Wir freuen uns, die Veröffentlichung von scikit-learn 1.8 bekannt zu geben! Viele Fehlerbehebungen und Verbesserungen wurden hinzugefügt, ebenso wie einige wichtige neue Funktionen. Nachfolgend erläutern wir die Highlights dieser Version. **Für eine vollständige Liste aller Änderungen** konsultieren Sie bitte die Release Notes.
Um die neueste Version zu installieren (mit pip)
pip install --upgrade scikit-learn
oder mit conda
conda install -c conda-forge scikit-learn
Array API-Unterstützung (ermöglicht GPU-Berechnungen)#
Die schrittweise Einführung des Python Array API-Standards in scikit-learn bedeutet, dass PyTorch und CuPy Eingangsarrays direkt verwendet werden. Dies bedeutet, dass in scikit-learn-Estimators und -Funktionen Nicht-CPU-Geräte, wie z. B. GPUs, zur Durchführung der Berechnungen verwendet werden können. Dies führt zu einer verbesserten Leistung und einer einfacheren Integration mit diesen Bibliotheken.
In scikit-learn 1.8 wurden mehrere Estimators und Funktionen aktualisiert, um Array API-kompatible Eingaben zu unterstützen, z. B. PyTorch-Tensoren und CuPy-Arrays.
Die Array API-Unterstützung wurde zu den folgenden Estimators hinzugefügt: preprocessing.StandardScaler, preprocessing.PolynomialFeatures, linear_model.RidgeCV, linear_model.RidgeClassifierCV, mixture.GaussianMixture und calibration.CalibratedClassifierCV.
Die Array API-Unterstützung wurde auch zu mehreren Metriken im sklearn.metrics-Modul hinzugefügt. Weitere Details finden Sie unter Unterstützung für Array API-kompatible Eingaben.
Bitte beachten Sie die Seite Array API-Unterstützung für Anleitungen zur Verwendung von scikit-learn mit Array API-kompatiblen Bibliotheken wie PyTorch oder CuPy. Hinweis: Die Array API-Unterstützung ist experimentell und muss sowohl in SciPy als auch in scikit-learn explizit aktiviert werden.
Hier ist ein Auszug aus der Verwendung eines Feature-Engineering-Präprozessors auf der CPU, gefolgt von calibration.CalibratedClassifierCV und linear_model.RidgeCV zusammen auf einer GPU mit Hilfe von PyTorch.
ridge_pipeline_gpu = make_pipeline(
# Ensure that all features (including categorical features) are preprocessed
# on the CPU and mapped to a numerical representation.
feature_preprocessor,
# Move the results to the GPU and perform computations there
FunctionTransformer(
lambda x: torch.tensor(x.to_numpy().astype(np.float32), device="cuda"))
,
CalibratedClassifierCV(
RidgeClassifierCV(alphas=alphas), method="temperature"
),
)
with sklearn.config_context(array_api_dispatch=True):
cv_results = cross_validate(ridge_pipeline_gpu, features, target)
Sehen Sie sich das vollständige Notebook auf Google Colab für weitere Details an. In diesem speziellen Beispiel führt die Verwendung der Colab GPU gegenüber einem einzelnen CPU-Kern zu einer 10-fachen Beschleunigung, was für solche Workloads recht typisch ist.
Unterstützung für Free-Threaded CPython 3.14#
scikit-learn unterstützt free-threaded CPython, insbesondere sind free-threaded Wheels für alle unsere unterstützten Plattformen unter Python 3.14 verfügbar.
Wir wären sehr an Benutzerfeedback interessiert. Hier sind einige Dinge, die Sie ausprobieren können:
Installieren Sie free-threaded CPython 3.14, führen Sie Ihr bevorzugtes scikit-learn-Skript aus und überprüfen Sie, ob nichts unerwartet kaputt geht. Beachten Sie, dass CPython 3.14 (anstelle von 3.13) dringend empfohlen wird, da seit CPython 3.13 einige Fehler bei free-threaded behoben wurden.
Wenn Sie Estimators mit einem
n_jobs-Parameter verwenden, versuchen Sie, das Standard-Backend mitjoblib.parallel_configwie im folgenden Snippet zu ändern. Dies könnte Ihren Code potenziell beschleunigen, da das Standard-Joblib-Backend prozessbasiert ist und mehr Overhead als Threads verursacht.grid_search = GridSearchCV(clf, param_grid=param_grid, n_jobs=4) with joblib.parallel_config(backend="threading"): grid_search.fit(X, y)
Zögern Sie nicht, Probleme oder unerwartetes Leistungsverhalten zu melden, indem Sie ein GitHub-Issue eröffnen!
Free-threaded (auch bekannt als nogil) CPython ist eine Version von CPython, die darauf abzielt, effiziente Multi-Threading-Anwendungsfälle zu ermöglichen, indem der Global Interpreter Lock (GIL) entfernt wird.
Weitere Details zu free-threaded CPython finden Sie in der py-free-threading-Dokumentation, insbesondere wie man ein free-threaded CPython installiert und Ecosystem-Kompatibilitätsverfolgung.
In scikit-learn hoffen wir mit free-threaded Python, Multi-Core-CPUs effizienter nutzen zu können, indem wir Thread-Worker anstelle von Subprozess-Workern für parallele Berechnungen verwenden, wenn n_jobs>1 in Funktionen oder Estimators übergeben wird. Effizienzsteigerungen werden durch die Vermeidung der Notwendigkeit der Interprozesskommunikation erwartet. Beachten Sie, dass die Änderung des Standard-Joblib-Backends und das Testen, ob alles gut mit free-threaded Python funktioniert, eine fortlaufende langfristige Anstrengung ist.
Temperaturskalierung in CalibratedClassifierCV#
Die Wahrscheinlichkeitskalibrierung von Klassifikatoren mit Temperaturskalierung ist in calibration.CalibratedClassifierCV durch Setzen von method="temperature" verfügbar. Diese Methode eignet sich besonders gut für Multiklassenprobleme, da sie (besser) kalibrierte Wahrscheinlichkeiten mit einem einzigen freien Parameter liefert. Dies steht im Gegensatz zu allen anderen verfügbaren Kalibrierungsmethoden, die ein „One-vs-Rest“-Schema verwenden, das zusätzliche Parameter für jede Klasse hinzufügt.
from sklearn.calibration import CalibratedClassifierCV
from sklearn.datasets import make_classification
from sklearn.naive_bayes import GaussianNB
X, y = make_classification(n_classes=3, n_informative=8, random_state=42)
clf = GaussianNB().fit(X, y)
sig = CalibratedClassifierCV(clf, method="sigmoid", ensemble=False).fit(X, y)
ts = CalibratedClassifierCV(clf, method="temperature", ensemble=False).fit(X, y)
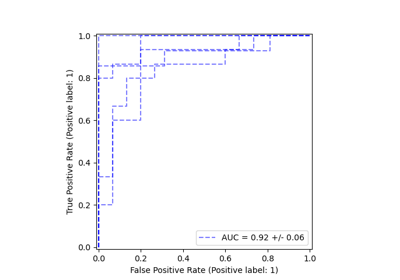
Das folgende Beispiel zeigt, dass die Temperaturskalierung bei Multiklassen-Klassifizierungsproblemen mit 3 Klassen zu besser kalibrierten Wahrscheinlichkeiten führen kann als die Sigmoid-Kalibrierung.
import matplotlib.pyplot as plt
from sklearn.calibration import CalibrationDisplay
fig, axes = plt.subplots(
figsize=(8, 4.5),
ncols=3,
sharey=True,
)
for i, c in enumerate(ts.classes_):
CalibrationDisplay.from_predictions(
y == c, clf.predict_proba(X)[:, i], name="Uncalibrated", ax=axes[i], marker="s"
)
CalibrationDisplay.from_predictions(
y == c,
ts.predict_proba(X)[:, i],
name="Temperature scaling",
ax=axes[i],
marker="o",
)
CalibrationDisplay.from_predictions(
y == c, sig.predict_proba(X)[:, i], name="Sigmoid", ax=axes[i], marker="v"
)
axes[i].set_title(f"Class {c}")
axes[i].set_xlabel(None)
axes[i].set_ylabel(None)
axes[i].get_legend().remove()
fig.suptitle("Reliability Diagrams per Class")
fig.supxlabel("Mean Predicted Probability")
fig.supylabel("Fraction of Class")
fig.legend(*axes[0].get_legend_handles_labels(), loc=(0.72, 0.5))
plt.subplots_adjust(right=0.7)
_ = fig.show()
Effizienzverbesserungen bei linearen Modellen#
Die Trainingszeit wurde für Schätzer, die auf quadratischen Fehlern basieren und eine L1-Strafe verwenden, massiv reduziert: ElasticNet, Lasso, MultiTaskElasticNet, MultiTaskLasso und ihre CV-Varianten. Die Verbesserung der Trainingszeit wird hauptsächlich durch **Gap Safe Screening Rules** erreicht. Diese ermöglichen es dem Coordinate Descent-Solver, Koeffizienten von Merkmalen frühzeitig auf Null zu setzen und sie nicht erneut zu betrachten. Je stärker die L1-Strafe, desto früher können Merkmale von weiteren Aktualisierungen ausgeschlossen werden.
from time import time
from sklearn.datasets import make_regression
from sklearn.linear_model import ElasticNetCV
X, y = make_regression(n_features=10_000, random_state=0)
model = ElasticNetCV()
tic = time()
model.fit(X, y)
toc = time()
print(f"Fitting ElasticNetCV took {toc - tic:.3} seconds.")
Fitting ElasticNetCV took 12.9 seconds.
HTML-Darstellung von Estimators#
Hyperparameter in der Dropdown-Tabelle der HTML-Darstellung enthalten jetzt Links zur Online-Dokumentation. Docstring-Beschreibungen werden auch als Tooltips beim Überfahren angezeigt.
from sklearn.linear_model import LogisticRegression
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import StandardScaler
clf = make_pipeline(StandardScaler(), LogisticRegression(random_state=0, C=10))
Erweitern Sie das untenstehende Estimator-Diagramm, indem Sie auf „LogisticRegression“ und dann auf „Parameters“ klicken.
clf
DecisionTreeRegressor mit criterion="absolute_error"#
tree.DecisionTreeRegressor mit criterion="absolute_error" läuft jetzt viel schneller. Es hat jetzt eine Komplexität von O(n * log(n)) im Vergleich zu O(n**2) zuvor, was die Skalierung auf Millionen von Datenpunkten ermöglicht.
Zur Veranschaulichung: Bei einem Datensatz mit 100.000 Samples und 1 Merkmal dauert ein einzelner Split etwa 100 ms, verglichen mit ca. 20 Sekunden zuvor.
import time
from sklearn.datasets import make_regression
from sklearn.tree import DecisionTreeRegressor
X, y = make_regression(n_samples=100_000, n_features=1)
tree = DecisionTreeRegressor(criterion="absolute_error", max_depth=1)
tic = time.time()
tree.fit(X, y)
elapsed = time.time() - tic
print(f"Fit took {elapsed:.2f} seconds")
Fit took 0.13 seconds
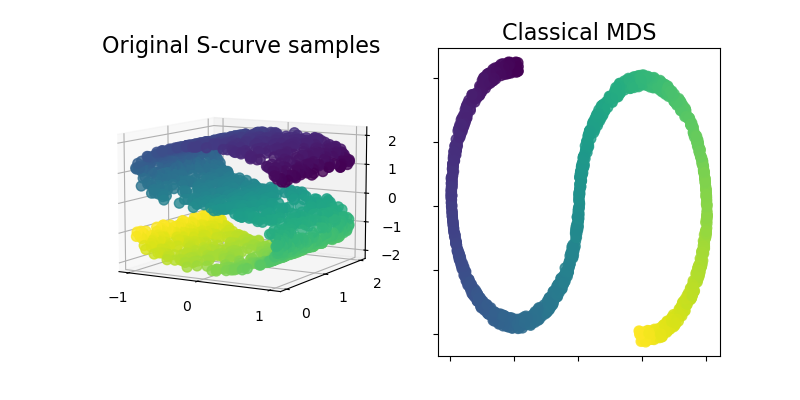
ClassicalMDS#
Classical MDS, auch bekannt als „Principal Coordinates Analysis“ (PCoA) oder „Torgerson’s Scaling“, ist jetzt im Modul sklearn.manifold verfügbar. Classical MDS ist ähnlich wie PCA und approximiert anstelle von Abständen paarweise Skalarprodukte, was eine exakte analytische Lösung in Form einer Eigenzerlegung hat.
Lassen Sie uns diese neue Ergänzung veranschaulichen, indem wir sie auf einem S-Kurven-Datensatz verwenden, um eine niedrigdimensionale Darstellung der Daten zu erhalten.
import matplotlib.pyplot as plt
from matplotlib import ticker
from sklearn import datasets, manifold
n_samples = 1500
S_points, S_color = datasets.make_s_curve(n_samples, random_state=0)
md_classical = manifold.ClassicalMDS(n_components=2)
S_scaling = md_classical.fit_transform(S_points)
fig = plt.figure(figsize=(8, 4))
ax1 = fig.add_subplot(1, 2, 1, projection="3d")
x, y, z = S_points.T
ax1.scatter(x, y, z, c=S_color, s=50, alpha=0.8)
ax1.set_title("Original S-curve samples", size=16)
ax1.view_init(azim=-60, elev=9)
for axis in (ax1.xaxis, ax1.yaxis, ax1.zaxis):
axis.set_major_locator(ticker.MultipleLocator(1))
ax2 = fig.add_subplot(1, 2, 2)
x2, y2 = S_scaling.T
ax2.scatter(x2, y2, c=S_color, s=50, alpha=0.8)
ax2.set_title("Classical MDS", size=16)
for axis in (ax2.xaxis, ax2.yaxis):
axis.set_major_formatter(ticker.NullFormatter())
plt.show()
Gesamtlaufzeit des Skripts: (0 Minuten 13,717 Sekunden)
Verwandte Beispiele