Hinweis
Zum Ende gehen, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Vergleich der K-Means- und MiniBatchKMeans-Clustering-Algorithmen#
Wir wollen die Leistung von MiniBatchKMeans und KMeans vergleichen: MiniBatchKMeans ist schneller, liefert aber leicht unterschiedliche Ergebnisse (siehe Mini Batch K-Means).
Wir werden einen Datensatz clustern, zuerst mit KMeans und dann mit MiniBatchKMeans, und die Ergebnisse plotten. Wir werden auch die Punkte plotten, die von den beiden Algorithmen unterschiedlich bezeichnet werden.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Generieren Sie die Daten#
Wir beginnen mit der Erzeugung der zu clusterten Daten-Blobs.
import numpy as np
from sklearn.datasets import make_blobs
np.random.seed(0)
batch_size = 45
centers = [[1, 1], [-1, -1], [1, -1]]
n_clusters = len(centers)
X, labels_true = make_blobs(n_samples=3000, centers=centers, cluster_std=0.7)
Clustering mit KMeans berechnen#
Clustering mit MiniBatchKMeans berechnen#
from sklearn.cluster import MiniBatchKMeans
mbk = MiniBatchKMeans(
init="k-means++",
n_clusters=3,
batch_size=batch_size,
n_init=10,
max_no_improvement=10,
verbose=0,
)
t0 = time.time()
mbk.fit(X)
t_mini_batch = time.time() - t0
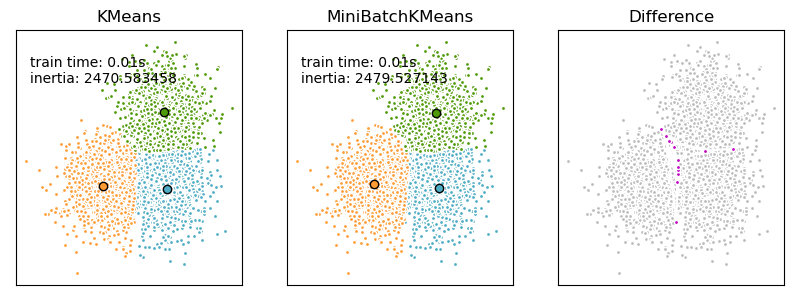
Festlegung der Übereinstimmung zwischen Clustern#
Wir möchten, dass die Cluster aus beiden Algorithmen (MiniBatchKMeans und KMeans) dieselbe Farbe haben. Lassen Sie uns die Clusterzentren nach dem nächstgelegenen zuordnen.
from sklearn.metrics.pairwise import pairwise_distances_argmin
k_means_cluster_centers = k_means.cluster_centers_
order = pairwise_distances_argmin(k_means.cluster_centers_, mbk.cluster_centers_)
mbk_means_cluster_centers = mbk.cluster_centers_[order]
k_means_labels = pairwise_distances_argmin(X, k_means_cluster_centers)
mbk_means_labels = pairwise_distances_argmin(X, mbk_means_cluster_centers)
Ergebnisse plotten#
import matplotlib.pyplot as plt
fig = plt.figure(figsize=(8, 3))
fig.subplots_adjust(left=0.02, right=0.98, bottom=0.05, top=0.9)
colors = ["#4EACC5", "#FF9C34", "#4E9A06"]
# KMeans
ax = fig.add_subplot(1, 3, 1)
for k, col in zip(range(n_clusters), colors):
my_members = k_means_labels == k
cluster_center = k_means_cluster_centers[k]
ax.plot(X[my_members, 0], X[my_members, 1], "w", markerfacecolor=col, marker=".")
ax.plot(
cluster_center[0],
cluster_center[1],
"o",
markerfacecolor=col,
markeredgecolor="k",
markersize=6,
)
ax.set_title("KMeans")
ax.set_xticks(())
ax.set_yticks(())
plt.text(-3.5, 1.8, "train time: %.2fs\ninertia: %f" % (t_batch, k_means.inertia_))
# MiniBatchKMeans
ax = fig.add_subplot(1, 3, 2)
for k, col in zip(range(n_clusters), colors):
my_members = mbk_means_labels == k
cluster_center = mbk_means_cluster_centers[k]
ax.plot(X[my_members, 0], X[my_members, 1], "w", markerfacecolor=col, marker=".")
ax.plot(
cluster_center[0],
cluster_center[1],
"o",
markerfacecolor=col,
markeredgecolor="k",
markersize=6,
)
ax.set_title("MiniBatchKMeans")
ax.set_xticks(())
ax.set_yticks(())
plt.text(-3.5, 1.8, "train time: %.2fs\ninertia: %f" % (t_mini_batch, mbk.inertia_))
# Initialize the different array to all False
different = mbk_means_labels == 4
ax = fig.add_subplot(1, 3, 3)
for k in range(n_clusters):
different += (k_means_labels == k) != (mbk_means_labels == k)
identical = np.logical_not(different)
ax.plot(X[identical, 0], X[identical, 1], "w", markerfacecolor="#bbbbbb", marker=".")
ax.plot(X[different, 0], X[different, 1], "w", markerfacecolor="m", marker=".")
ax.set_title("Difference")
ax.set_xticks(())
ax.set_yticks(())
plt.show()
Gesamtlaufzeit des Skripts: (0 Minuten 0,164 Sekunden)
Verwandte Beispiele
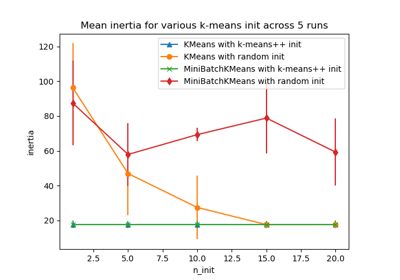
Empirische Auswertung des Einflusses der K-Means Initialisierung
Demo des Affinity Propagation Clustering Algorithmus