Hinweis
Zum Ende springen, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Sparsere Schätzung der inversen Kovarianz#
Verwendung des GraphicalLasso-Schätzers, um eine Kovarianz und eine sparsere Präzision aus einer kleinen Anzahl von Stichproben zu lernen.
Um ein probabilistisches Modell (z.B. ein Gaußsches Modell) zu schätzen, ist die Schätzung der Präzisionsmatrix, d.h. der inversen Kovarianzmatrix, ebenso wichtig wie die Schätzung der Kovarianzmatrix. Tatsächlich wird ein Gaußsches Modell durch die Präzisionsmatrix parametrisiert.
Um günstige Wiederherstellungsbedingungen zu haben, werden die Daten aus einem Modell mit einer sparsen inversen Kovarianzmatrix abgetastet. Zusätzlich stellen wir sicher, dass die Daten nicht zu stark korreliert sind (was den größten Koeffizienten der Präzisionsmatrix begrenzt) und dass keine kleinen Koeffizienten in der Präzisionsmatrix vorhanden sind, die nicht wiederhergestellt werden können. Darüber hinaus ist es bei einer kleinen Anzahl von Beobachtungen einfacher, eine Korrelationsmatrix anstelle einer Kovarianz wiederherzustellen, daher skalieren wir die Zeitreihen.
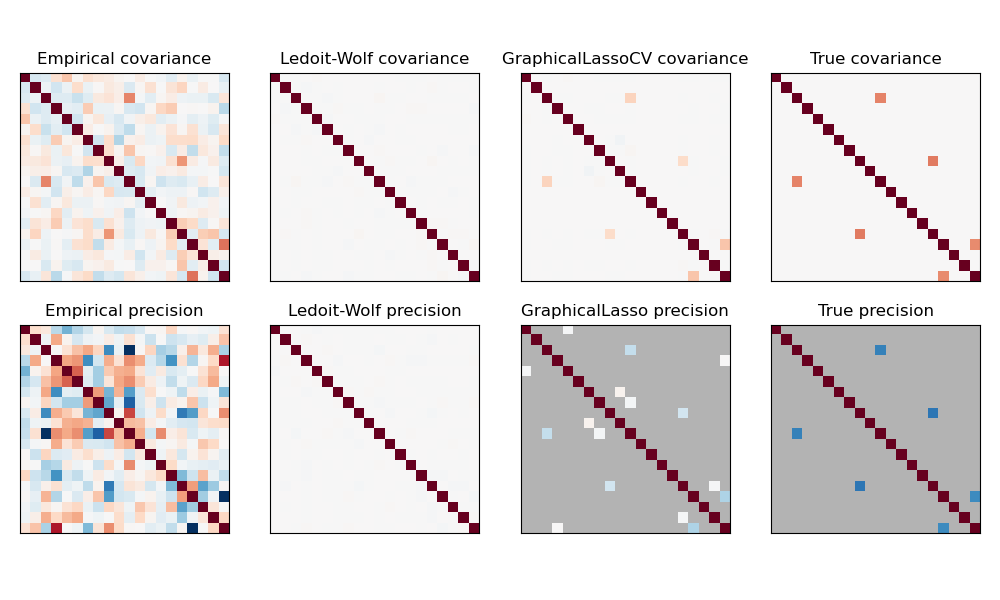
Hier ist die Anzahl der Stichproben etwas größer als die Anzahl der Dimensionen, daher ist die empirische Kovarianz noch invertierbar. Da die Beobachtungen jedoch stark korreliert sind, ist die empirische Kovarianzmatrix schlecht konditioniert und folglich ist ihre Inverse – die empirische Präzisionsmatrix – sehr weit von der Grundwahrheit entfernt.
Wenn wir L2-Shrinkage verwenden, wie beim Ledoit-Wolf-Schätzer, müssen wir, da die Anzahl der Stichproben klein ist, stark schrumpfen. Infolgedessen ist die Ledoit-Wolf-Präzision ziemlich nahe an der tatsächlichen Präzision, die nicht weit davon entfernt ist, diagonal zu sein, aber die Struktur der Nicht-Diagonale geht verloren.
Der L1-penalisierte Schätzer kann einen Teil dieser Nicht-Diagonalstruktur wiederherstellen. Er lernt eine sparsere Präzision. Er ist nicht in der Lage, das exakte Sparsity-Muster wiederherzustellen: Er erkennt zu viele Nicht-Null-Koeffizienten. Die höchsten Nicht-Null-Koeffizienten der L1-Schätzung entsprechen jedoch den Nicht-Null-Koeffizienten in der Grundwahrheit. Schließlich sind die Koeffizienten der L1-Präzisionsschätzung gegen Null verzerrt: Aufgrund der Strafe sind sie alle kleiner als der entsprechende Wert der Grundwahrheit, wie in der Abbildung zu sehen ist.
Beachten Sie, dass der Farbbereich der Präzisionsmatrizen zur besseren Lesbarkeit der Abbildung angepasst wurde. Der gesamte Wertebereich der empirischen Präzision wird nicht angezeigt.
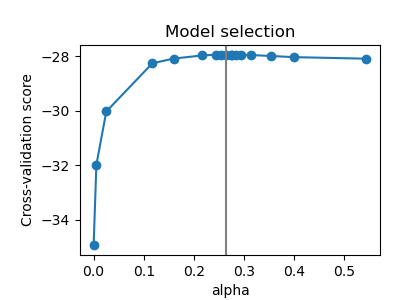
Der Alpha-Parameter von GraphicalLasso, der die Sparsity des Modells festlegt, wird durch interne Kreuzvalidierung in GraphicalLassoCV festgelegt. Wie in Abbildung 2 zu sehen ist, wird das Gitter zur Berechnung des Kreuzvalidierungs-Scores iterativ in der Nähe des Maximums verfeinert.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Generieren Sie die Daten#
import numpy as np
from scipy import linalg
from sklearn.datasets import make_sparse_spd_matrix
n_samples = 60
n_features = 20
prng = np.random.RandomState(1)
prec = make_sparse_spd_matrix(
n_features, alpha=0.98, smallest_coef=0.4, largest_coef=0.7, random_state=prng
)
cov = linalg.inv(prec)
d = np.sqrt(np.diag(cov))
cov /= d
cov /= d[:, np.newaxis]
prec *= d
prec *= d[:, np.newaxis]
X = prng.multivariate_normal(np.zeros(n_features), cov, size=n_samples)
X -= X.mean(axis=0)
X /= X.std(axis=0)
Schätzen Sie die Kovarianz#
from sklearn.covariance import GraphicalLassoCV, ledoit_wolf
emp_cov = np.dot(X.T, X) / n_samples
model = GraphicalLassoCV()
model.fit(X)
cov_ = model.covariance_
prec_ = model.precision_
lw_cov_, _ = ledoit_wolf(X)
lw_prec_ = linalg.inv(lw_cov_)
Ergebnisse plotten#
import matplotlib.pyplot as plt
plt.figure(figsize=(10, 6))
plt.subplots_adjust(left=0.02, right=0.98)
# plot the covariances
covs = [
("Empirical", emp_cov),
("Ledoit-Wolf", lw_cov_),
("GraphicalLassoCV", cov_),
("True", cov),
]
vmax = cov_.max()
for i, (name, this_cov) in enumerate(covs):
plt.subplot(2, 4, i + 1)
plt.imshow(
this_cov, interpolation="nearest", vmin=-vmax, vmax=vmax, cmap=plt.cm.RdBu_r
)
plt.xticks(())
plt.yticks(())
plt.title("%s covariance" % name)
# plot the precisions
precs = [
("Empirical", linalg.inv(emp_cov)),
("Ledoit-Wolf", lw_prec_),
("GraphicalLasso", prec_),
("True", prec),
]
vmax = 0.9 * prec_.max()
for i, (name, this_prec) in enumerate(precs):
ax = plt.subplot(2, 4, i + 5)
plt.imshow(
np.ma.masked_equal(this_prec, 0),
interpolation="nearest",
vmin=-vmax,
vmax=vmax,
cmap=plt.cm.RdBu_r,
)
plt.xticks(())
plt.yticks(())
plt.title("%s precision" % name)
if hasattr(ax, "set_facecolor"):
ax.set_facecolor(".7")
else:
ax.set_axis_bgcolor(".7")
# plot the model selection metric
plt.figure(figsize=(4, 3))
plt.axes([0.2, 0.15, 0.75, 0.7])
plt.plot(model.cv_results_["alphas"], model.cv_results_["mean_test_score"], "o-")
plt.axvline(model.alpha_, color=".5")
plt.title("Model selection")
plt.ylabel("Cross-validation score")
plt.xlabel("alpha")
plt.show()
Gesamtlaufzeit des Skripts: (0 Minuten 0,569 Sekunden)
Verwandte Beispiele
Schrumpfkovarianzschätzung: LedoitWolf vs OAS und Maximum-Likelihood
Lineare und Quadratische Diskriminanzanalyse mit Kovarianzellipsoid