Hinweis
Gehen Sie zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
Kurvenanpassung mit Bayes'scher Ridge-Regression#
Berechnet eine Bayes'sche Ridge-Regression von Sinusoiden.
Weitere Informationen zur Regressionsmethode finden Sie unter Bayes'sche Ridge-Regression.
Im Allgemeinen kann bei der Anpassung einer Kurve mit einem Polynom durch Bayes'sche Ridge-Regression die Auswahl der Anfangswerte der Regularisierungsparameter (alpha, lambda) wichtig sein. Dies liegt daran, dass die Regularisierungsparameter durch ein iteratives Verfahren bestimmt werden, das von den Anfangswerten abhängt.
In diesem Beispiel wird die Sinusoide mithilfe verschiedener Paare von Anfangswerten durch ein Polynom approximiert.
Wenn man von den Standardwerten (alpha_init = 1.90, lambda_init = 1.) ausgeht, ist der Bias der resultierenden Kurve groß und die Varianz klein. Daher sollte lambda_init relativ klein (1.e-3) sein, um den Bias zu reduzieren.
Durch die Auswertung der logarithmischen marginalen Likelihood (L) dieser Modelle können wir auch bestimmen, welches besser ist. Es kann daraus geschlossen werden, dass das Modell mit größerem L wahrscheinlicher ist.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
Generieren von Sinusoid-Daten mit Rauschen#
import numpy as np
def func(x):
return np.sin(2 * np.pi * x)
size = 25
rng = np.random.RandomState(1234)
x_train = rng.uniform(0.0, 1.0, size)
y_train = func(x_train) + rng.normal(scale=0.1, size=size)
x_test = np.linspace(0.0, 1.0, 100)
Anpassung durch kubisches Polynom#
from sklearn.linear_model import BayesianRidge
n_order = 3
X_train = np.vander(x_train, n_order + 1, increasing=True)
X_test = np.vander(x_test, n_order + 1, increasing=True)
reg = BayesianRidge(tol=1e-6, fit_intercept=False, compute_score=True)
Plot der wahren und vorhergesagten Kurven mit logarithmischer marginaler Likelihood (L)#
import matplotlib.pyplot as plt
fig, axes = plt.subplots(1, 2, figsize=(8, 4))
for i, ax in enumerate(axes):
# Bayesian ridge regression with different initial value pairs
if i == 0:
init = [1 / np.var(y_train), 1.0] # Default values
elif i == 1:
init = [1.0, 1e-3]
reg.set_params(alpha_init=init[0], lambda_init=init[1])
reg.fit(X_train, y_train)
ymean, ystd = reg.predict(X_test, return_std=True)
ax.plot(x_test, func(x_test), color="blue", label="sin($2\\pi x$)")
ax.scatter(x_train, y_train, s=50, alpha=0.5, label="observation")
ax.plot(x_test, ymean, color="red", label="predict mean")
ax.fill_between(
x_test, ymean - ystd, ymean + ystd, color="pink", alpha=0.5, label="predict std"
)
ax.set_ylim(-1.3, 1.3)
ax.legend()
title = "$\\alpha$_init$={:.2f},\\ \\lambda$_init$={}$".format(init[0], init[1])
if i == 0:
title += " (Default)"
ax.set_title(title, fontsize=12)
text = "$\\alpha={:.1f}$\n$\\lambda={:.3f}$\n$L={:.1f}$".format(
reg.alpha_, reg.lambda_, reg.scores_[-1]
)
ax.text(0.05, -1.0, text, fontsize=12)
plt.tight_layout()
plt.show()

Gesamtlaufzeit des Skripts: (0 Minuten 0,229 Sekunden)
Verwandte Beispiele

Verwendung von KBinsDiscretizer zur Diskretisierung kontinuierlicher Merkmale
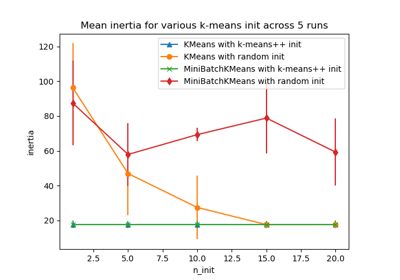
Empirische Auswertung des Einflusses der K-Means Initialisierung