Hinweis
Gehen Sie zum Ende, um den vollständigen Beispielcode herunterzuladen oder dieses Beispiel über JupyterLite oder Binder in Ihrem Browser auszuführen.
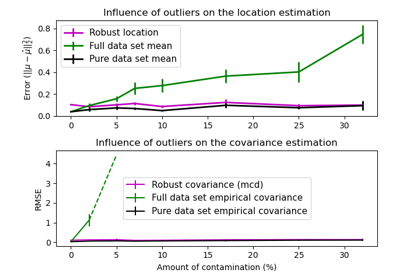
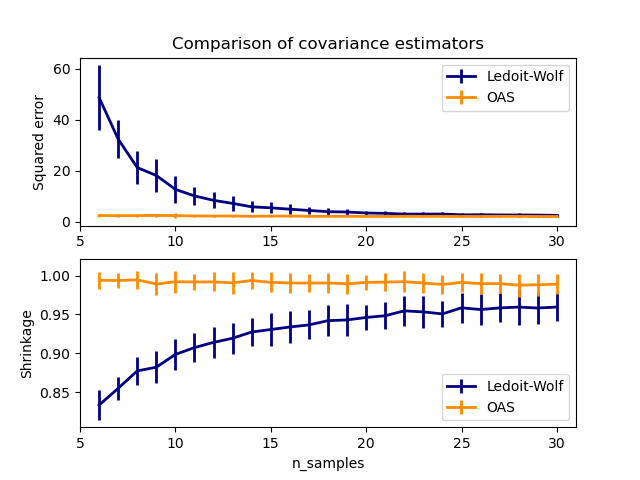
Ledoit-Wolf vs OAS-Schätzung#
Die übliche Maximum-Likelihood-Schätzung der Kovarianz kann durch Schrumpfung regularisiert werden. Ledoit und Wolf schlugen eine geschlossene Formel vor, um den asymptotisch optimalen Schrumpfungsparameter zu berechnen (der ein MSE-Kriterium minimiert), was zu der Ledoit-Wolf-Kovarianzschätzung führt.
Chen et al. [1] schlugen eine Verbesserung des Ledoit-Wolf-Schrumpfungsparameters vor, den OAS-Koeffizienten, dessen Konvergenz unter der Annahme, dass die Daten Gaußsch verteilt sind, signifikant besser ist.
Dieses Beispiel, inspiriert von Chen's Veröffentlichung [1], zeigt einen Vergleich der geschätzten MSE der LW- und OAS-Methoden unter Verwendung von Gaußsch verteilten Daten.
Referenzen
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
import matplotlib.pyplot as plt
import numpy as np
from scipy.linalg import cholesky, toeplitz
from sklearn.covariance import OAS, LedoitWolf
np.random.seed(0)
n_features = 100
# simulation covariance matrix (AR(1) process)
r = 0.1
real_cov = toeplitz(r ** np.arange(n_features))
coloring_matrix = cholesky(real_cov)
n_samples_range = np.arange(6, 31, 1)
repeat = 100
lw_mse = np.zeros((n_samples_range.size, repeat))
oa_mse = np.zeros((n_samples_range.size, repeat))
lw_shrinkage = np.zeros((n_samples_range.size, repeat))
oa_shrinkage = np.zeros((n_samples_range.size, repeat))
for i, n_samples in enumerate(n_samples_range):
for j in range(repeat):
X = np.dot(np.random.normal(size=(n_samples, n_features)), coloring_matrix.T)
lw = LedoitWolf(store_precision=False, assume_centered=True)
lw.fit(X)
lw_mse[i, j] = lw.error_norm(real_cov, scaling=False)
lw_shrinkage[i, j] = lw.shrinkage_
oa = OAS(store_precision=False, assume_centered=True)
oa.fit(X)
oa_mse[i, j] = oa.error_norm(real_cov, scaling=False)
oa_shrinkage[i, j] = oa.shrinkage_
# plot MSE
plt.subplot(2, 1, 1)
plt.errorbar(
n_samples_range,
lw_mse.mean(1),
yerr=lw_mse.std(1),
label="Ledoit-Wolf",
color="navy",
lw=2,
)
plt.errorbar(
n_samples_range,
oa_mse.mean(1),
yerr=oa_mse.std(1),
label="OAS",
color="darkorange",
lw=2,
)
plt.ylabel("Squared error")
plt.legend(loc="upper right")
plt.title("Comparison of covariance estimators")
plt.xlim(5, 31)
# plot shrinkage coefficient
plt.subplot(2, 1, 2)
plt.errorbar(
n_samples_range,
lw_shrinkage.mean(1),
yerr=lw_shrinkage.std(1),
label="Ledoit-Wolf",
color="navy",
lw=2,
)
plt.errorbar(
n_samples_range,
oa_shrinkage.mean(1),
yerr=oa_shrinkage.std(1),
label="OAS",
color="darkorange",
lw=2,
)
plt.xlabel("n_samples")
plt.ylabel("Shrinkage")
plt.legend(loc="lower right")
plt.ylim(plt.ylim()[0], 1.0 + (plt.ylim()[1] - plt.ylim()[0]) / 10.0)
plt.xlim(5, 31)
plt.show()
Gesamtlaufzeit des Skripts: (0 Minuten 2,252 Sekunden)
Verwandte Beispiele
Schrumpfkovarianzschätzung: LedoitWolf vs OAS und Maximum-Likelihood
Normale, Ledoit-Wolf und OAS Lineare Diskriminanzanalyse zur Klassifikation