SpectralClustering#
- class sklearn.cluster.SpectralClustering(n_clusters=8, *, eigen_solver=None, n_components=None, random_state=None, n_init=10, gamma=1.0, affinity='rbf', n_neighbors=10, eigen_tol='auto', assign_labels='kmeans', degree=3, coef0=1, kernel_params=None, n_jobs=None, verbose=False)[Quelle]#
Wendet Clustering auf eine Projektion des normalisierten Laplace-Operators an.
In der Praxis ist Spectral Clustering sehr nützlich, wenn die Struktur der einzelnen Cluster stark nicht-konvex ist oder allgemeiner, wenn ein Maß für das Zentrum und die Streuung des Clusters keine geeignete Beschreibung des vollständigen Clusters darstellt, z. B. wenn sich die Cluster in verschachtelten Kreisen auf der 2D-Ebene befinden.
Wenn die Affinitätsmatrix die Adjazenzmatrix eines Graphen ist, kann diese Methode verwendet werden, um normalisierte Graph-Schnitte zu finden [1], [2].
Beim Aufruf von
fitwird eine Affinitätsmatrix konstruiert, entweder mittels einer Kernel-Funktion wie dem Gaußschen (auch RBF) Kernel mit Euklidischem Abstandd(X, X)np.exp(-gamma * d(X,X) ** 2)
oder einer k-nächsten-Nachbarn-Konnektivitätsmatrix.
Alternativ kann eine vom Benutzer bereitgestellte Affinitätsmatrix durch Setzen von
affinity='precomputed'angegeben werden.Lesen Sie mehr im Benutzerhandbuch.
- Parameter:
- n_clustersint, default=8
Die Dimension des Projektionsunterraums.
- eigen_solver{‘arpack’, ‘lobpcg’, ‘amg’}, default=None
Die Strategie für die Eigenwertzerlegung. AMG erfordert die Installation von pyamg. Es kann bei sehr großen, dünn besetzten Problemen schneller sein, kann aber auch zu Instabilitäten führen. Wenn None, wird
'arpack'verwendet. Siehe [4] für weitere Details zu'lobpcg'.- n_componentsint, default=None
Anzahl der zu verwendenden Eigenvektoren für das spektrale Embedding. Wenn None, wird standardmäßig
n_clustersverwendet.- random_stateint, RandomState instance, default=None
Ein pseudo-zufälliger Generator zur Initialisierung der lobpcg-Eigenvektoren-Zerlegung, wenn
eigen_solver == 'amg'und für die K-Means-Initialisierung. Verwenden Sie eine Ganzzahl, um die Ergebnisse über Aufrufe hinweg deterministisch zu machen (siehe Glossar).Hinweis
Bei Verwendung von
eigen_solver == 'amg'ist es notwendig, auch den globalen Numpy-Seed mitnp.random.seed(int)festzulegen, um deterministische Ergebnisse zu erzielen. Siehe pyamg/pyamg#139 für weitere Informationen.- n_initint, Standard=10
Anzahl der Durchläufe des K-Means-Algorithmus mit unterschiedlichen Zentroid-Seeds. Die Endergebnisse sind die besten Ausgaben von n_init aufeinanderfolgenden Durchläufen in Bezug auf die Trägheit. Wird nur verwendet, wenn
assign_labels='kmeans'.- gammafloat, default=1.0
Kernel-Koeffizient für rbf, poly, sigmoid, laplacian und chi2-Kernel. Ignoriert für
affinity='nearest_neighbors',affinity='precomputed'oderaffinity='precomputed_nearest_neighbors'.- affinitystr oder callable, default=’rbf’
- Wie die Affinitätsmatrix konstruiert wird.
‘nearest_neighbors’: Konstruiert die Affinitätsmatrix durch Berechnung eines Graphen der nächsten Nachbarn.
‘rbf’: Konstruiert die Affinitätsmatrix unter Verwendung eines Radial Basis Function (RBF) Kernels.
‘precomputed’: Interpretiert
Xals eine vorab berechnete Affinitätsmatrix, bei der größere Werte eine größere Ähnlichkeit zwischen Instanzen anzeigen.‘precomputed_nearest_neighbors’: Interpretiert
Xals einen dünn besetzten Graphen von vorab berechneten Entfernungen und konstruiert eine binäre Affinitätsmatrix aus denn_neighborsnächsten Nachbarn jeder Instanz.einer der von
pairwise_kernelsunterstützten Kernel.
Es sollten nur Kernel verwendet werden, die Ähnlichkeitswerte liefern (nicht-negative Werte, die mit zunehmender Ähnlichkeit steigen). Diese Eigenschaft wird vom Clustering-Algorithmus nicht überprüft.
- n_neighborsint, default=10
Anzahl der Nachbarn, die bei der Konstruktion der Affinitätsmatrix unter Verwendung der Methode der nächsten Nachbarn verwendet werden. Ignoriert für
affinity='rbf'.- eigen_tolfloat, default=”auto”
Abbruchkriterium für die Eigenzerlegung der Laplace-Matrix. Wenn
eigen_tol="auto", dann hängt die übergebene Toleranz vomeigen_solverab.Wenn
eigen_solver="arpack", dann isteigen_tol=0.0;Wenn
eigen_solver="lobpcg"odereigen_solver="amg", dann isteigen_tol=None, was den zugrunde liegendenlobpcg-Solver so konfiguriert, dass der Wert gemäß seinen Heuristiken automatisch aufgelöst wird. Siehescipy.sparse.linalg.lobpcgfür Details.
Beachten Sie, dass bei Verwendung von
eigen_solver="lobpcg"odereigen_solver="amg"Werte vontol<1e-5zu Konvergenzproblemen führen können und vermieden werden sollten.Hinzugefügt in Version 1.2: Option 'auto' hinzugefügt.
- assign_labels{‘kmeans’, ‘discretize’, ‘cluster_qr’}, default=’kmeans’
Die Strategie für die Zuweisung von Labels im Embedding-Raum. Es gibt zwei Möglichkeiten, Labels nach dem Laplace-Embedding zuzuweisen. k-Means ist eine beliebte Wahl, kann aber empfindlich auf die Initialisierung reagieren. Diskretisierung ist ein weiterer Ansatz, der weniger empfindlich auf zufällige Initialisierung reagiert [3]. Die cluster_qr-Methode [5] extrahiert direkt Cluster aus Eigenvektoren im spektralen Clustering. Im Gegensatz zu k-Means und Diskretisierung hat cluster_qr keine Abstimmungsparameter und führt keine Iterationen durch, kann aber k-Means und Diskretisierung in Bezug auf Qualität und Geschwindigkeit übertreffen.
Geändert in Version 1.1: Neue Labeling-Methode 'cluster_qr' hinzugefügt.
- degreefloat, Standard=3
Grad des Polynom-Kernels. Ignoriert von anderen Kerneln.
- coef0float, Standard=1
Null-Koeffizient für Polynom- und Sigmoid-Kernel. Ignoriert von anderen Kerneln.
- kernel_paramsdict of str to any, default=None
Parameter (Schlüsselwortargumente) und Werte für den als callable übergebenen Kernel. Ignoriert von anderen Kerneln.
- n_jobsint, default=None
Die Anzahl der parallelen Jobs, die ausgeführt werden, wenn
affinity='nearest_neighbors'oderaffinity='precomputed_nearest_neighbors'. Die Nachbarschaftssuche wird parallel durchgeführt.Nonebedeutet 1, es sei denn, Sie befinden sich in einemjoblib.parallel_backendKontext.-1bedeutet die Verwendung aller Prozessoren. Siehe Glossar für weitere Details.- verbosebool, default=False
Ausführlichkeitsmodus.
Hinzugefügt in Version 0.24.
- Attribute:
- affinity_matrix_array-like of shape (n_samples, n_samples)
Affinitätsmatrix, die für das Clustering verwendet wird. Nur verfügbar, nachdem
fitaufgerufen wurde.- labels_ndarray der Form (n_samples,)
Labels jedes Punkts
- n_features_in_int
Anzahl der während des fits gesehenen Merkmale.
Hinzugefügt in Version 0.24.
- feature_names_in_ndarray mit Form (
n_features_in_,) Namen der während fit gesehenen Merkmale. Nur definiert, wenn
XMerkmalnamen hat, die alle Zeichenketten sind.Hinzugefügt in Version 1.0.
Siehe auch
sklearn.cluster.KMeansK-Means-Clustering.
sklearn.cluster.DBSCANDichtebasierte räumliche Clusterbildung von Anwendungen mit Rauschen.
Anmerkungen
Eine Distanzmatrix, bei der 0 identische Elemente kennzeichnet und hohe Werte sehr unähnliche Elemente anzeigen, kann durch Anwendung des Gaußschen (auch RBF, Hitze-) Kernels in eine für den Algorithmus gut geeignete Affinitäts-/Ähnlichkeitsmatrix umgewandelt werden.
np.exp(- dist_matrix ** 2 / (2. * delta ** 2))
wobei
deltaein freier Parameter ist, der die Breite des Gaußschen Kernels darstellt.Eine Alternative ist die Verwendung einer symmetrischen Version der k-nächsten-Nachbarn-Konnektivitätsmatrix der Punkte.
Wenn das pyamg-Paket installiert ist, wird es verwendet: dies beschleunigt die Berechnung erheblich.
Referenzen
[4]Beispiele
>>> from sklearn.cluster import SpectralClustering >>> import numpy as np >>> X = np.array([[1, 1], [2, 1], [1, 0], ... [4, 7], [3, 5], [3, 6]]) >>> clustering = SpectralClustering(n_clusters=2, ... assign_labels='discretize', ... random_state=0).fit(X) >>> clustering.labels_ array([1, 1, 1, 0, 0, 0]) >>> clustering SpectralClustering(assign_labels='discretize', n_clusters=2, random_state=0)
Einen Vergleich von spektraler Clusterbildung mit anderen Clustering-Algorithmen finden Sie unter Vergleich verschiedener Clustering-Algorithmen auf Spielzeugdatensätzen
- fit(X, y=None)[Quelle]#
Führt spektrale Clusterbildung aus Merkmalen oder einer Affinitätsmatrix durch.
- Parameter:
- X{array-artig, spärs matrix} der Form (n_samples, n_features) oder (n_samples, n_samples)
Trainingsinstanzen zum Clustern, Ähnlichkeiten / Affinitäten zwischen Instanzen, wenn
affinity='precomputed', oder Distanzen zwischen Instanzen, wennaffinity='precomputed_nearest_neighbors. Wenn eine dünn besetzte Matrix in einem anderen Format alscsr_matrix,csc_matrixodercoo_matrixbereitgestellt wird, wird sie in eine dünn besetztecsr_matrixkonvertiert.- yIgnoriert
Nicht verwendet, hier zur API-Konsistenz durch Konvention vorhanden.
- Gibt zurück:
- selfobject
Eine trainierte Instanz des Schätzers.
- fit_predict(X, y=None)[Quelle]#
Führt spektrale Clusterbildung auf
Xdurch und gibt Cluster-Labels zurück.- Parameter:
- X{array-artig, spärs matrix} der Form (n_samples, n_features) oder (n_samples, n_samples)
Trainingsinstanzen zum Clustern, Ähnlichkeiten / Affinitäten zwischen Instanzen, wenn
affinity='precomputed', oder Distanzen zwischen Instanzen, wennaffinity='precomputed_nearest_neighbors. Wenn eine dünn besetzte Matrix in einem anderen Format alscsr_matrix,csc_matrixodercoo_matrixbereitgestellt wird, wird sie in eine dünn besetztecsr_matrixkonvertiert.- yIgnoriert
Nicht verwendet, hier zur API-Konsistenz durch Konvention vorhanden.
- Gibt zurück:
- labelsndarray der Form (n_samples,)
Clusterbeschriftungen.
- get_metadata_routing()[Quelle]#
Holt das Metadaten-Routing dieses Objekts.
Bitte prüfen Sie im Benutzerhandbuch, wie der Routing-Mechanismus funktioniert.
- Gibt zurück:
- routingMetadataRequest
Ein
MetadataRequest, der Routing-Informationen kapselt.
- get_params(deep=True)[Quelle]#
Holt Parameter für diesen Schätzer.
- Parameter:
- deepbool, default=True
Wenn True, werden die Parameter für diesen Schätzer und die enthaltenen Unterobjekte, die Schätzer sind, zurückgegeben.
- Gibt zurück:
- paramsdict
Parameternamen, zugeordnet ihren Werten.
- set_params(**params)[Quelle]#
Setzt die Parameter dieses Schätzers.
Die Methode funktioniert sowohl bei einfachen Schätzern als auch bei verschachtelten Objekten (wie
Pipeline). Letztere haben Parameter der Form<component>__<parameter>, so dass es möglich ist, jede Komponente eines verschachtelten Objekts zu aktualisieren.- Parameter:
- **paramsdict
Schätzer-Parameter.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
Galeriebeispiele#
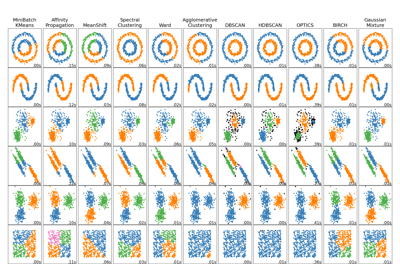
Vergleich verschiedener Clustering-Algorithmen auf Toy-Datensätzen