FeatureAgglomeration#
- class sklearn.cluster.FeatureAgglomeration(n_clusters=2, *, metric='euclidean', memory=None, connectivity=None, compute_full_tree='auto', linkage='ward', pooling_func=<function mean>, distance_threshold=None, compute_distances=False)[Quelle]#
Mergt Merkmale.
Fügt rekursiv Merkmalscluster zusammen.
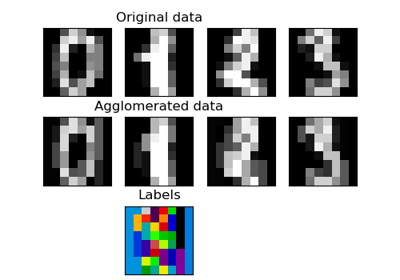
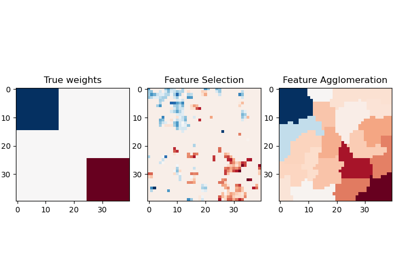
Siehe Feature Agglomeration im Vergleich zur univariaten Auswahl für ein Beispiel für die Strategie
FeatureAgglomerationim Vergleich zu einer univariaten Merkmalsauswahlstrategie (basierend auf ANOVA).Lesen Sie mehr im Benutzerhandbuch.
- Parameter:
- n_clustersint oder None, Standard=2
Die Anzahl der zu findenden Cluster. Muss
Nonesein, wenndistance_thresholdnichtNoneist.- metricstr oder callable, Standard=”euclidean”
Metrik zur Berechnung der Verknüpfung. Kann "euclidean", "l1", "l2", "manhattan", "cosine" oder "precomputed" sein. Wenn linkage "ward" ist, wird nur "euclidean" akzeptiert. Wenn "precomputed", wird eine Distanzmatrix als Eingabe für die fit-Methode benötigt.
Hinzugefügt in Version 1.2.
- memoryString oder Objekt mit dem joblib.Memory-Interface, Standard=None
Wird verwendet, um die Ausgabe der Baumkalkulation zu cachen. Standardmäßig erfolgt keine Zwischenspeicherung. Wenn ein String angegeben wird, ist dies der Pfad zum Caching-Verzeichnis.
- connectivityarray-like, sparse matrix oder callable, Standard=None
Konnetivitätsmatrix. Definiert für jedes Merkmal die benachbarten Merkmale, die einer bestimmten Struktur der Daten folgen. Dies kann eine Konnetivitätsmatrix selbst sein oder eine callable, die die Daten in eine Konnetivitätsmatrix umwandelt, wie z. B. aus
kneighbors_graphabgeleitet. Standard istNone, d.h. der hierarchische Clustering-Algorithmus ist unstrukturiert.- compute_full_tree‘auto’ oder bool, Standard=’auto’
Stoppt frühzeitig den Aufbau des Baumes bei
n_clusters. Dies ist nützlich, um die Berechnungszeit zu verkürzen, wenn die Anzahl der Cluster nicht klein im Verhältnis zur Anzahl der Merkmale ist. Diese Option ist nur nützlich, wenn eine Konnetivitätsmatrix angegeben wird. Beachten Sie auch, dass es bei Variation der Anzahl der Cluster und Verwendung von Caching vorteilhaft sein kann, den vollständigen Baum zu berechnen. MussTruesein, wenndistance_thresholdnichtNoneist. Standardmäßig istcompute_full_tree"auto", was äquivalent zuTrueist, wenndistance_thresholdnichtNoneist odern_clusterskleiner als das Maximum von 100 oder0.02 * n_samplesist. Andernfalls ist "auto" äquivalent zuFalse.- linkage{“ward”, “complete”, “average”, “single”}, Standard=”ward”
Welches Verknüpfungskriterium verwendet werden soll. Das Verknüpfungskriterium bestimmt, welche Distanz zwischen Merkmalsmengen verwendet wird. Der Algorithmus wird die Clusterpaare zusammenführen, die dieses Kriterium minimieren.
„ward“ minimiert die Varianz der zusammengeführten Cluster.
„complete“ oder maximale Verknüpfung verwendet die maximalen Distanzen zwischen allen Merkmalen der beiden Mengen.
„average“ verwendet den Durchschnitt der Distanzen jedes Merkmals der beiden Mengen.
„single“ verwendet das Minimum der Distanzen zwischen allen Merkmalen der beiden Mengen.
- pooling_funccallable, Standard=np.mean
Kombiniert die Werte der agglomerierten Merkmale zu einem einzigen Wert und sollte ein Array der Form [M, N] und das Schlüsselwortargument
axis=1akzeptieren und es auf ein Array der Größe [M] reduzieren.- distance_thresholdfloat, Standard=None
Die Verknüpfungsdistanzschwelle, bei oder über der Cluster nicht mehr zusammengeführt werden. Wenn nicht
None, mussn_clustersNonesein undcompute_full_treemussTruesein.Hinzugefügt in Version 0.21.
- compute_distancesbool, Standard=False
Berechnet die Distanzen zwischen Clustern, auch wenn
distance_thresholdnicht verwendet wird. Dies kann zur Visualisierung von Dendrogrammen verwendet werden, verursacht aber einen Rechen- und Speicheraufwand.Hinzugefügt in Version 0.24.
- Attribute:
- n_clusters_int
Die vom Algorithmus gefundene Anzahl von Clustern. Wenn
distance_threshold=None, ist sie gleich dem gegebenenn_clusters.- labels_array-like von (n_features,)
Cluster-Labels für jedes Merkmal.
- n_leaves_int
Anzahl der Blätter im hierarchischen Baum.
- n_connected_components_int
Die geschätzte Anzahl von verbundenen Komponenten im Graphen.
Hinzugefügt in Version 0.21:
n_connected_components_wurde hinzugefügt, umn_components_zu ersetzen.- n_features_in_int
Anzahl der während des fits gesehenen Merkmale.
Hinzugefügt in Version 0.24.
- feature_names_in_ndarray mit Form (
n_features_in_,) Namen der während fit gesehenen Merkmale. Nur definiert, wenn
XMerkmalnamen hat, die alle Zeichenketten sind.Hinzugefügt in Version 1.0.
- children_array-like der Form (n_nodes-1, 2)
Die Kinder jedes Nicht-Blatt-Knotens. Werte kleiner als
n_featuresentsprechen Blättern des Baumes, die die ursprünglichen Samples sind. Ein Knotenigrößer oder gleichn_featuresist ein Nicht-Blatt-Knoten und hat die Kinderchildren_[i - n_features]. Alternativ werden in der i-ten Iteration children[i][0] und children[i][1] zusammengeführt, um den Knotenn_features + izu bilden.- distances_array-like der Form (n_nodes-1,)
Distanzen zwischen den Knoten an der entsprechenden Stelle in
children_. Nur berechnet, wenndistance_thresholdverwendet wird odercompute_distancesaufTruegesetzt ist.
Siehe auch
AgglomerativeClusteringAgglomerative Clustering wendet dies auf Samples anstelle von Merkmalen an.
ward_treeHierarchisches Clustering mit Ward-Verknüpfung.
Beispiele
>>> import numpy as np >>> from sklearn import datasets, cluster >>> digits = datasets.load_digits() >>> images = digits.images >>> X = np.reshape(images, (len(images), -1)) >>> agglo = cluster.FeatureAgglomeration(n_clusters=32) >>> agglo.fit(X) FeatureAgglomeration(n_clusters=32) >>> X_reduced = agglo.transform(X) >>> X_reduced.shape (1797, 32)
- fit(X, y=None)[Quelle]#
Passt das hierarchische Clustering an die Daten an.
- Parameter:
- Xarray-like der Form (n_samples, n_features)
Die Daten.
- yIgnoriert
Nicht verwendet, hier zur API-Konsistenz durch Konvention vorhanden.
- Gibt zurück:
- selfobject
Gibt den Transformer zurück.
- property fit_predict#
Passt an und gibt das Ergebnis der Clustering-Zuweisung jedes Samples zurück.
- fit_transform(X, y=None, **fit_params)[Quelle]#
An Daten anpassen, dann transformieren.
Passt den Transformer an
Xundymit optionalen Parameternfit_paramsan und gibt eine transformierte Version vonXzurück.- Parameter:
- Xarray-like der Form (n_samples, n_features)
Eingabestichproben.
- yarray-like der Form (n_samples,) oder (n_samples, n_outputs), Standardwert=None
Zielwerte (None für unüberwachte Transformationen).
- **fit_paramsdict
Zusätzliche Fit-Parameter. Nur übergeben, wenn der Estimator zusätzliche Parameter in seiner
fit-Methode akzeptiert.
- Gibt zurück:
- X_newndarray array der Form (n_samples, n_features_new)
Transformiertes Array.
- get_feature_names_out(input_features=None)[Quelle]#
Holt die Ausgabemerkmale für die Transformation.
Die Feature-Namen werden mit dem kleingeschriebenen Klassennamen präfixiert. Wenn der Transformer z.B. 3 Features ausgibt, dann sind die Feature-Namen:
["klassenname0", "klassenname1", "klassenname2"].- Parameter:
- input_featuresarray-like von str oder None, default=None
Wird nur verwendet, um die Feature-Namen mit den in
fitgesehenen Namen zu validieren.
- Gibt zurück:
- feature_names_outndarray von str-Objekten
Transformierte Merkmalnamen.
- get_metadata_routing()[Quelle]#
Holt das Metadaten-Routing dieses Objekts.
Bitte prüfen Sie im Benutzerhandbuch, wie der Routing-Mechanismus funktioniert.
- Gibt zurück:
- routingMetadataRequest
Ein
MetadataRequest, der Routing-Informationen kapselt.
- get_params(deep=True)[Quelle]#
Holt Parameter für diesen Schätzer.
- Parameter:
- deepbool, default=True
Wenn True, werden die Parameter für diesen Schätzer und die enthaltenen Unterobjekte, die Schätzer sind, zurückgegeben.
- Gibt zurück:
- paramsdict
Parameternamen, zugeordnet ihren Werten.
- inverse_transform(X)[Quelle]#
Kehrt die Transformation um und gibt einen Vektor der Größe
n_featureszurück.- Parameter:
- Xarray-like der Form (n_samples, n_clusters) oder (n_clusters,)
Die Werte, die jedem Cluster von Samples zugewiesen werden sollen.
- Gibt zurück:
- X_originalndarray der Form (n_samples, n_features) oder (n_features,)
Ein Vektor der Größe
n_samplesmit den Werten vonX, die jedem der Sample-Cluster zugewiesen sind.
- set_output(*, transform=None)[Quelle]#
Ausgabecontainer festlegen.
Siehe Einführung in die set_output API für ein Beispiel zur Verwendung der API.
- Parameter:
- transform{“default”, “pandas”, “polars”}, default=None
Konfiguriert die Ausgabe von
transformundfit_transform."default": Standardausgabeformat eines Transformers"pandas": DataFrame-Ausgabe"polars": Polars-AusgabeNone: Die Transformationskonfiguration bleibt unverändert
Hinzugefügt in Version 1.4: Die Option
"polars"wurde hinzugefügt.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
- set_params(**params)[Quelle]#
Setzt die Parameter dieses Schätzers.
Die Methode funktioniert sowohl bei einfachen Schätzern als auch bei verschachtelten Objekten (wie
Pipeline). Letztere haben Parameter der Form<component>__<parameter>, so dass es möglich ist, jede Komponente eines verschachtelten Objekts zu aktualisieren.- Parameter:
- **paramsdict
Schätzer-Parameter.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
- transform(X)[Quelle]#
Transformiert eine neue Matrix mithilfe des erstellten Clusterings.
- Parameter:
- Xarray-ähnlich der Form (n_samples, n_features) oder (n_samples, n_samples)
Ein M mal N-Array von M Beobachtungen in N Dimensionen oder ein Array der Länge M von M eindimensionalen Beobachtungen.
- Gibt zurück:
- Yndarray der Form (n_samples, n_clusters) oder (n_clusters,)
Die gepoolten Werte für jeden Merkmalscluster.