SGDOneClassSVM#
- class sklearn.linear_model.SGDOneClassSVM(nu=0.5, fit_intercept=True, max_iter=1000, tol=0.001, shuffle=True, verbose=0, random_state=None, learning_rate='optimal', eta0=0.01, power_t=0.5, warm_start=False, average=False)[source]#
Löst lineare One-Class-SVM mit stochastischem Gradientenabstieg.
Diese Implementierung ist für die Verwendung mit einer Kernel-Approximationstechnik (z. B.
sklearn.kernel_approximation.Nystroem) gedacht, um ähnliche Ergebnisse wiesklearn.svm.OneClassSVMzu erzielen, das standardmäßig einen Gaußschen Kernel verwendet.Lesen Sie mehr im Benutzerhandbuch.
Hinzugefügt in Version 1.0.
- Parameter:
- nufloat, Standard=0.5
Der Parameter nu des One-Class-SVM: eine obere Schranke für den Anteil der Trainingsfehler und eine untere Schranke für den Anteil der Stützvektoren. Sollte im Intervall (0, 1] liegen. Standardmäßig wird 0.5 verwendet.
- fit_interceptbool, Standardwert=True
Ob der Achsenabschnitt geschätzt werden soll oder nicht. Standardmäßig True.
- max_iterint, default=1000
Die maximale Anzahl von Durchläufen über die Trainingsdaten (auch Epochen genannt). Beeinflusst nur das Verhalten in der
fitMethode und nicht diepartial_fit. Standardmäßig 1000. Werte müssen im Bereich[1, inf)liegen.- tolfloat oder None, default=1e-3
Das Abbruchkriterium. Wenn es nicht None ist, werden die Iterationen gestoppt, wenn (loss > previous_loss - tol). Standardmäßig 1e-3. Werte müssen im Bereich
[0.0, inf)liegen.- shufflebool, Standard=True
Ob die Trainingsdaten nach jeder Epoche gemischt werden sollen oder nicht. Standardmäßig True.
- verboseint, default=0
Die Ausführlichkeitsstufe.
- random_stateint, RandomState-Instanz oder None, default=None
Der Seed des Pseudo-Zufallszahlengenerators, der beim Mischen der Daten verwendet werden soll. Wenn int, ist random_state der Seed, der vom Zufallszahlengenerator verwendet wird; Wenn RandomState-Instanz, ist random_state der Zufallszahlengenerator; Wenn None, ist der Zufallszahlengenerator die RandomState-Instanz, die von
np.randomverwendet wird.- learning_rate{‘constant’, ‘optimal’, ‘invscaling’, ‘adaptive’}, Standard=’optimal’
Der Lernratenschedule, der mit
fitverwendet werden soll. (Wennpartial_fitverwendet wird, muss die Lernrate direkt gesteuert werden).‘constant’:
eta = eta0‘optimal’:
eta = 1.0 / (alpha * (t + t0)), wobei t0 durch eine von Leon Bottou vorgeschlagene Heuristik gewählt wird.‘invscaling’:
eta = eta0 / pow(t, power_t)‘adaptive’: eta = eta0, solange das Training abnimmt. Jedes Mal, wenn n_iter_no_change aufeinanderfolgende Epochen die Trainingsverluste um tol nicht verringern oder den Validierungs-Score um tol nicht erhöhen, wenn early_stopping True ist, wird die aktuelle Lernrate durch 5 geteilt.
- eta0float, default=0.01
Die anfängliche Lernrate für die Schedules ‘constant’, ‘invscaling’ oder ‘adaptive’. Der Standardwert ist 0.0, aber beachten Sie, dass eta0 nicht von der Standard-Lernrate ‘optimal’ verwendet wird. Werte müssen im Bereich
(0.0, inf)liegen.- power_tfloat, default=0.5
Der Exponent für die umgekehrt proportionale Lernrate. Werte müssen im Bereich
[0.0, inf)liegen.Veraltet seit Version 1.8: Negative Werte für
power_tsind in Version 1.8 veraltet und lösen in 1.10 einen Fehler aus. Verwenden Sie stattdessen Werte im Bereich [0.0, inf).- warm_startbool, Standard=False
Wenn auf True gesetzt, wird die Lösung des vorherigen Aufrufs von fit als Initialisierung wiederverwendet, andernfalls wird die vorherige Lösung einfach verworfen. Siehe das Glossar.
Wiederholtes Aufrufen von fit oder partial_fit, wenn warm_start True ist, kann zu einer anderen Lösung führen als beim einmaligen Aufrufen von fit, aufgrund der Art und Weise, wie die Daten gemischt werden. Wenn eine dynamische Lernrate verwendet wird, wird die Lernrate in Abhängigkeit von der Anzahl der bereits gesehenen Stichproben angepasst. Das Aufrufen von
fitsetzt diesen Zähler zurück, währendpartial_fitdazu führt, dass der vorhandene Zähler erhöht wird.- averagebool oder int, default=False
Wenn True gesetzt, werden die gemittelten SGD-Gewichte berechnet und im Attribut
coef_gespeichert. Wenn auf eine Ganzzahl größer als 1 gesetzt, beginnt die Mittelung, sobald die Gesamtzahl der gesehenen Stichproben die `average` erreicht. Also wirdaverage=10nach dem Sehen von 10 Stichproben mit der Mittelung beginnen.
- Attribute:
- coef_ndarray der Form (1, n_features)
Den Merkmalen zugeordnete Gewichte.
- offset_ndarray der Form (1,)
Offset, der verwendet wird, um die Entscheidungsgrenze aus den Rohwerten zu definieren. Wir haben die Beziehung: decision_function = score_samples - offset.
- n_iter_int
Die tatsächliche Anzahl der Iterationen, um das Abbruchkriterium zu erreichen.
- t_int
Anzahl der während des Trainings durchgeführten Gewichtungsaktualisierungen. Gleich wie
(n_iter_ * n_samples + 1).- n_features_in_int
Anzahl der während des fits gesehenen Merkmale.
Hinzugefügt in Version 0.24.
- feature_names_in_ndarray mit Form (
n_features_in_,) Namen der während fit gesehenen Merkmale. Nur definiert, wenn
XMerkmalnamen hat, die alle Zeichenketten sind.Hinzugefügt in Version 1.0.
Siehe auch
sklearn.svm.OneClassSVMUnüberwachte Ausreißererkennung.
Anmerkungen
Dieser Schätzer hat eine lineare Komplexität in Bezug auf die Anzahl der Trainingsstichproben und ist daher besser für Datensätze mit einer großen Anzahl von Trainingsstichproben (z. B. > 10.000) geeignet als die
sklearn.svm.OneClassSVMImplementierung.Beispiele
>>> import numpy as np >>> from sklearn import linear_model >>> X = np.array([[-1, -1], [-2, -1], [1, 1], [2, 1]]) >>> clf = linear_model.SGDOneClassSVM(random_state=42, tol=None) >>> clf.fit(X) SGDOneClassSVM(random_state=42, tol=None)
>>> print(clf.predict([[4, 4]])) [1]
- decision_function(X)[source]#
Signierte Entfernung zur trennenden Hyperebene.
Die signierte Entfernung ist positiv für ein Inlier und negativ für ein Outlier.
- Parameter:
- X{array-like, sparse matrix}, shape (n_samples, n_features)
Testdaten.
- Gibt zurück:
- decarray-like, Form (n_samples,)
Entscheidungsfunktionwerte der Stichproben.
- densify()[source]#
Konvertiert die Koeffizientenmatrix in ein dichtes Array-Format.
Konvertiert das Mitglied
coef_(zurück) in ein numpy.ndarray. Dies ist das Standardformat voncoef_und wird für das Training benötigt, daher muss diese Methode nur auf Modellen aufgerufen werden, die zuvor verknappt wurden; andernfalls ist sie eine No-Op.- Gibt zurück:
- self
Angepasster Schätzer.
- fit(X, y=None, coef_init=None, offset_init=None, sample_weight=None)[source]#
Linearen One-Class-SVM mit Stochastic Gradient Descent anpassen.
Dies löst ein äquivalentes Optimierungsproblem des primalen Optimierungsproblems des One-Class-SVM und gibt einen Gewichtsvektor w und einen Offset rho zurück, sodass die Entscheidungsgrenze durch <w, x> - rho gegeben ist.
- Parameter:
- X{array-like, sparse matrix}, shape (n_samples, n_features)
Trainingsdaten.
- yIgnoriert
Wird nicht verwendet, ist aber aus Gründen der API-Konsistenz per Konvention vorhanden.
- coef_initarray, Form (n_classes, n_features)
Die anfänglichen Koeffizienten zum Aufwärmen der Optimierung.
- offset_initarray, Form (n_classes,)
Der anfängliche Offset zum Warm-Starten der Optimierung.
- sample_weightarray-like, Form (n_samples,), optional
Gewichte, die auf einzelne Stichproben angewendet werden. Wenn nicht angegeben, werden einheitliche Gewichte angenommen. Diese Gewichte werden mit class_weight (über den Konstruktor übergeben) multipliziert, wenn class_weight angegeben ist.
- Gibt zurück:
- selfobject
Gibt eine angepasste Instanz von self zurück.
- fit_predict(X, y=None, **kwargs)[source]#
Führt die Anpassung an X durch und gibt Labels für X zurück.
Gibt -1 für Ausreißer und 1 für Inlier zurück.
- Parameter:
- X{array-like, sparse matrix} der Form (n_samples, n_features)
Die Eingabestichproben.
- yIgnoriert
Wird nicht verwendet, ist aber aus Gründen der API-Konsistenz per Konvention vorhanden.
- **kwargsdict
Argumente, die an
fitübergeben werden sollen.Hinzugefügt in Version 1.4.
- Gibt zurück:
- yndarray der Form (n_samples,)
1 für Inlier, -1 für Ausreißer.
- get_metadata_routing()[source]#
Holt das Metadaten-Routing dieses Objekts.
Bitte prüfen Sie im Benutzerhandbuch, wie der Routing-Mechanismus funktioniert.
- Gibt zurück:
- routingMetadataRequest
Ein
MetadataRequest, der Routing-Informationen kapselt.
- get_params(deep=True)[source]#
Holt Parameter für diesen Schätzer.
- Parameter:
- deepbool, default=True
Wenn True, werden die Parameter für diesen Schätzer und die enthaltenen Unterobjekte, die Schätzer sind, zurückgegeben.
- Gibt zurück:
- paramsdict
Parameternamen, zugeordnet ihren Werten.
- partial_fit(X, y=None, sample_weight=None)[source]#
Linearen One-Class-SVM mit Stochastic Gradient Descent anpassen.
- Parameter:
- X{array-like, sparse matrix}, shape (n_samples, n_features)
Teilmenge der Trainingsdaten.
- yIgnoriert
Wird nicht verwendet, ist aber aus Gründen der API-Konsistenz per Konvention vorhanden.
- sample_weightarray-like, Form (n_samples,), optional
Gewichte, die auf einzelne Stichproben angewendet werden. Wenn nicht angegeben, werden einheitliche Gewichte angenommen.
- Gibt zurück:
- selfobject
Gibt eine angepasste Instanz von self zurück.
- predict(X)[source]#
Gibt Labels (1 Inlier, -1 Outlier) der Stichproben zurück.
- Parameter:
- X{array-like, sparse matrix}, shape (n_samples, n_features)
Testdaten.
- Gibt zurück:
- yarray, Form (n_samples,)
Labels der Stichproben.
- score_samples(X)[source]#
Rohe Scoring-Funktion der Stichproben.
- Parameter:
- X{array-like, sparse matrix}, shape (n_samples, n_features)
Testdaten.
- Gibt zurück:
- score_samplesarray-like, Form (n_samples,)
Unverschobene Scoring-Funktionswerte der Stichproben.
- set_fit_request(*, coef_init: bool | None | str = '$UNCHANGED$', offset_init: bool | None | str = '$UNCHANGED$', sample_weight: bool | None | str = '$UNCHANGED$') SGDOneClassSVM[source]#
Konfiguriert, ob Metadaten für die
fit-Methode angefordert werden sollen.Beachten Sie, dass diese Methode nur relevant ist, wenn dieser Schätzer als Unter-Schätzer innerhalb eines Meta-Schätzers verwendet wird und Metadaten-Routing mit
enable_metadata_routing=Trueaktiviert ist (siehesklearn.set_config). Bitte lesen Sie das Benutzerhandbuch, um zu erfahren, wie der Routing-Mechanismus funktioniert.Die Optionen für jeden Parameter sind
True: Metadaten werden angefordert und, falls vorhanden, anfitübergeben. Die Anforderung wird ignoriert, wenn keine Metadaten vorhanden sind.False: Metadaten werden nicht angefordert und der Meta-Schätzer übergibt sie nicht anfit.None: Metadaten werden nicht angefordert und der Meta-Schätzer löst einen Fehler aus, wenn der Benutzer sie bereitstellt.str: Metadaten sollten mit diesem Alias an den Meta-Schätzer übergeben werden und nicht mit dem ursprünglichen Namen.
Der Standardwert (
sklearn.utils.metadata_routing.UNCHANGED) behält die bestehende Anforderung bei. Dies ermöglicht es Ihnen, die Anforderung für einige Parameter zu ändern und für andere nicht.Hinzugefügt in Version 1.3.
- Parameter:
- coef_initstr, True, False, oder None, default=sklearn.utils.metadata_routing.UNCHANGED
Metadaten-Routing für den Parameter
coef_initinfit.- offset_initstr, True, False, oder None, Standard=sklearn.utils.metadata_routing.UNCHANGED
Metadaten-Routing für den Parameter
offset_initinfit.- sample_weightstr, True, False, oder None, Standardwert=sklearn.utils.metadata_routing.UNCHANGED
Metadaten-Routing für den Parameter
sample_weightinfit.
- Gibt zurück:
- selfobject
Das aktualisierte Objekt.
- set_params(**params)[source]#
Setzt die Parameter dieses Schätzers.
Die Methode funktioniert sowohl bei einfachen Schätzern als auch bei verschachtelten Objekten (wie
Pipeline). Letztere haben Parameter der Form<component>__<parameter>, so dass es möglich ist, jede Komponente eines verschachtelten Objekts zu aktualisieren.- Parameter:
- **paramsdict
Schätzer-Parameter.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
- set_partial_fit_request(*, sample_weight: bool | None | str = '$UNCHANGED$') SGDOneClassSVM[source]#
Konfiguriert, ob Metadaten für die
partial_fit-Methode angefordert werden sollen.Beachten Sie, dass diese Methode nur relevant ist, wenn dieser Schätzer als Unter-Schätzer innerhalb eines Meta-Schätzers verwendet wird und Metadaten-Routing mit
enable_metadata_routing=Trueaktiviert ist (siehesklearn.set_config). Bitte lesen Sie das Benutzerhandbuch, um zu erfahren, wie der Routing-Mechanismus funktioniert.Die Optionen für jeden Parameter sind
True: Metadaten werden angefordert und anpartial_fitübergeben, wenn sie bereitgestellt werden. Die Anforderung wird ignoriert, wenn keine Metadaten bereitgestellt werden.False: Metadaten werden nicht angefordert und der Meta-Schätzer übergibt sie nicht anpartial_fit.None: Metadaten werden nicht angefordert und der Meta-Schätzer löst einen Fehler aus, wenn der Benutzer sie bereitstellt.str: Metadaten sollten mit diesem Alias an den Meta-Schätzer übergeben werden und nicht mit dem ursprünglichen Namen.
Der Standardwert (
sklearn.utils.metadata_routing.UNCHANGED) behält die bestehende Anforderung bei. Dies ermöglicht es Ihnen, die Anforderung für einige Parameter zu ändern und für andere nicht.Hinzugefügt in Version 1.3.
- Parameter:
- sample_weightstr, True, False, oder None, Standardwert=sklearn.utils.metadata_routing.UNCHANGED
Metadaten-Routing für den Parameter
sample_weightinpartial_fit.
- Gibt zurück:
- selfobject
Das aktualisierte Objekt.
- sparsify()[source]#
Koeffizientenmatrix in Sparse-Format konvertieren.
Konvertiert das
coef_-Mitglied in eine scipy.sparse-Matrix, die für Modelle mit L1-Regularisierung speicher- und speichereffizienter sein kann als die übliche numpy.ndarray-Darstellung.Das
intercept_-Mitglied wird nicht konvertiert.- Gibt zurück:
- self
Angepasster Schätzer.
Anmerkungen
Für nicht-sparse Modelle, d.h. wenn nicht viele Nullen in
coef_vorhanden sind, kann dies tatsächlich den Speicherverbrauch *erhöhen*, also verwenden Sie diese Methode mit Vorsicht. Eine Faustregel besagt, dass die Anzahl der Nullelemente, die mit(coef_ == 0).sum()berechnet werden kann, mehr als 50 % betragen muss, damit dies signifikante Vorteile bringt.Nach dem Aufruf dieser Methode funktioniert die weitere Anpassung mit der Methode
partial_fit(falls vorhanden) nicht mehr, bis Siedensifyaufrufen.
Galeriebeispiele#
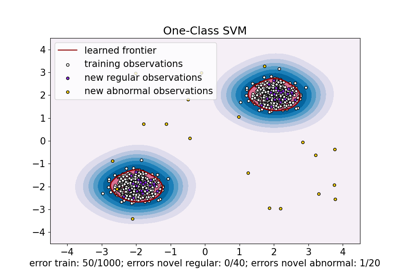
One-Class SVM vs. One-Class SVM mittels Stochastic Gradient Descent
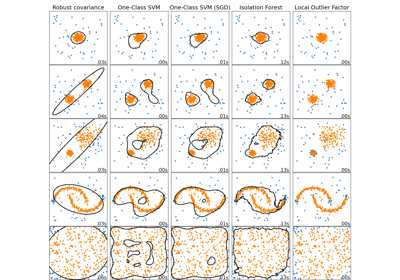
Vergleich von Anomalieerkennungsalgorithmen zur Ausreißererkennung auf Toy-Datensätzen