f1_score#
- sklearn.metrics.f1_score(y_true, y_pred, *, labels=None, pos_label=1, average='binary', sample_weight=None, zero_division='warn')[source]#
Berechne den F1-Score, auch bekannt als Balanced F-Score oder F-Maß.
Der F1-Score kann als harmonisches Mittel aus Präzision und Recall interpretiert werden, wobei ein F1-Score seinen besten Wert bei 1 und seinen schlechtesten Wert bei 0 erreicht. Der relative Beitrag von Präzision und Recall zum F1-Score ist gleich. Die Formel für den F1-Score lautet:
\[\text{F1} = \frac{2 * \text{TP}}{2 * \text{TP} + \text{FP} + \text{FN}}\]Dabei ist \(\text{TP}\) die Anzahl der wahren Positive, \(\text{FN}\) die Anzahl der falschen Negative und \(\text{FP}\) die Anzahl der falschen Positive. Der F1-Score wird standardmäßig als 0,0 berechnet, wenn keine wahren Positive, falschen Negative oder falschen Positive vorhanden sind.
Unterstützung über binäre Ziele hinaus wird erreicht, indem multiclass- und multilabel-Daten als Sammlung von binären Problemen behandelt werden, eines für jedes Label. Im binären Fall gibt die Einstellung
average='binary'den F1-Score fürpos_labelzurück. Wennaveragenicht'binary'ist, wirdpos_labelignoriert und der F1-Score für beide Klassen berechnet, dann gemittelt oder beide zurückgegeben (wennaverage=None). Ebenso werden für multiclass- und multilabel-Ziele die F1-Scores für allelabelsentweder zurückgegeben oder gemittelt, abhängig vomaverage-Parameter. Verwenden Sielabels, um die Menge der Labels anzugeben, für die der F1-Score berechnet werden soll.Lesen Sie mehr im Benutzerhandbuch.
- Parameter:
- y_true1D Array-ähnlich oder Label-Indikator-Array / Sparse Matrix
Wahre (korrekte) Zielwerte. Sparse Matrix wird nur unterstützt, wenn die Ziele vom Typ Multilabel sind.
- y_pred1D Array-ähnlich oder Label-Indikator-Array / Sparse Matrix
Geschätzte Ziele, wie sie von einem Klassifikator zurückgegeben werden. Sparse Matrix wird nur unterstützt, wenn die Ziele vom Typ Multilabel sind.
- labelsarray-artig, Standardwert=None
Die Menge der Bezeichnungen, die eingeschlossen werden sollen, wenn
average != 'binary', und ihre Reihenfolge, wennaverage is None. Bezeichnungen, die in den Daten vorhanden sind, können ausgeschlossen werden, z. B. bei der Multiklassenklassifizierung, um eine „negative Klasse“ auszuschließen. Bezeichnungen, die nicht in den Daten vorhanden sind, können eingeschlossen werden und erhalten 0 Stichproben. Bei Multilabel-Zielen sind Bezeichnungen Spaltenindizes. Standardmäßig werden alle Bezeichnungen iny_trueundy_predin sortierter Reihenfolge verwendet.Geändert in Version 0.17: Der Parameter
labelswurde für Multiklassenprobleme verbessert.- pos_labelint, float, bool oder str, Standard=1
Die Klasse, für die berichtet werden soll, wenn
average='binary'und die Daten binär sind, andernfalls wird dieser Parameter ignoriert. Für Multiklassen- oder Multilabel-Ziele setzen Sielabels=[pos_label]undaverage != 'binary', um Metriken für nur eine Bezeichnung zu berichten.- average{‘micro’, ‘macro’, ‘samples’, ‘weighted’, ‘binary’} oder None, Standard=’binary’
Dieser Parameter ist für Multiklassen-/Multilabel-Ziele erforderlich. Wenn
None, werden die Metriken für jede Klasse zurückgegeben. Andernfalls bestimmt dieser Parameter die Art der Mittelung, die auf den Daten durchgeführt wird.'binary':Nur Ergebnisse für die durch
pos_labelangegebene Klasse berichten. Dies ist nur anwendbar, wenn die Ziele (y_{true,pred}) binär sind.'micro':Metriken global berechnen, indem die gesamten wahren Positive, falschen Negative und falschen Positive gezählt werden.
'macro':Metriken für jede Bezeichnung berechnen und deren ungewichtetes Mittel finden. Dies berücksichtigt keine unausgeglichenen Bezeichnungen.
'weighted':Berechnet Metriken für jedes Label und ermittelt deren Durchschnitt, gewichtet nach dem Support (die Anzahl der tatsächlichen Instanzen für jedes Label). Dies verändert „macro“, um die Ungleichheit der Labels zu berücksichtigen; es kann zu einem F-Score führen, der nicht zwischen Präzision und Recall liegt.
'samples':Metriken für jede Instanz berechnen und deren Mittel finden (nur sinnvoll für Multilabel-Klassifizierung, wo dies von
accuracy_scoreabweicht).
- sample_weightarray-like der Form (n_samples,), Standardwert=None
Stichprobengewichte.
- zero_division{“warn”, 0.0, 1.0, np.nan}, Standard=”warn”
Legt den Wert fest, der zurückgegeben wird, wenn eine Division durch Null auftritt, d. h. wenn alle Vorhersagen und Labels negativ sind.
Hinweise: - Wenn auf "warn" gesetzt, verhält sich dies wie 0, aber es wird auch eine Warnung ausgegeben. - Wenn auf
np.nangesetzt, werden solche Werte vom Durchschnitt ausgeschlossen.Hinzugefügt in Version 1.3: Option
np.nanhinzugefügt.
- Gibt zurück:
- f1_scorefloat oder Array von float, Form = [n_unique_labels]
F1-Score der positiven Klasse in der binären Klassifizierung oder gewichteter Durchschnitt der F1-Scores jeder Klasse für die Multiclass-Aufgabe.
Siehe auch
fbeta_scoreBerechne den F-beta-Score.
precision_recall_fscore_supportBerechnet Präzision, Recall, F-Score und Support.
jaccard_scoreBerechnet den Jaccard-Ähnlichkeitskoeffizienten-Score.
multilabel_confusion_matrixBerechne eine Konfusionsmatrix für jede Klasse oder Stichprobe.
Anmerkungen
Wenn
wahre positive + falsche positive + falsche negative == 0(d. h. eine Klasse ist vollständig ausy_trueodery_predabwesend), ist der F-Score undefiniert. In solchen Fällen wird der F-Score standardmäßig auf 0,0 gesetzt, undUndefinedMetricWarningwird ausgelöst. Dieses Verhalten kann durch Setzen des Parameterszero_divisiongeändert werden.Referenzen
Beispiele
>>> import numpy as np >>> from sklearn.metrics import f1_score >>> y_true = [0, 1, 2, 0, 1, 2] >>> y_pred = [0, 2, 1, 0, 0, 1] >>> f1_score(y_true, y_pred, average='macro') 0.267 >>> f1_score(y_true, y_pred, average='micro') 0.33 >>> f1_score(y_true, y_pred, average='weighted') 0.267 >>> f1_score(y_true, y_pred, average=None) array([0.8, 0. , 0. ])
>>> # binary classification >>> y_true_empty = [0, 0, 0, 0, 0, 0] >>> y_pred_empty = [0, 0, 0, 0, 0, 0] >>> f1_score(y_true_empty, y_pred_empty) 0.0... >>> f1_score(y_true_empty, y_pred_empty, zero_division=1.0) 1.0... >>> f1_score(y_true_empty, y_pred_empty, zero_division=np.nan) nan...
>>> # multilabel classification >>> y_true = [[0, 0, 0], [1, 1, 1], [0, 1, 1]] >>> y_pred = [[0, 0, 0], [1, 1, 1], [1, 1, 0]] >>> f1_score(y_true, y_pred, average=None) array([0.66666667, 1. , 0.66666667])
Galeriebeispiele#
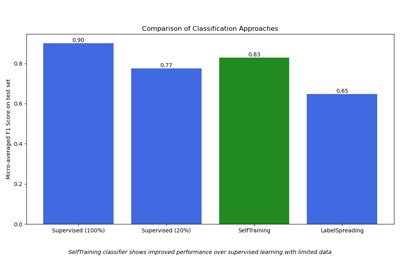
Semi-überwachte Klassifikation auf einem Textdatensatz