HDBSCAN#
- class sklearn.cluster.HDBSCAN(min_cluster_size=5, min_samples=None, cluster_selection_epsilon=0.0, max_cluster_size=None, metric='euclidean', metric_params=None, alpha=1.0, algorithm='auto', leaf_size=40, n_jobs=None, cluster_selection_method='eom', allow_single_cluster=False, store_centers=None, copy='warn')[Quelle]#
Cluster Daten mittels hierarchischem dichte-basiertem Clustering.
HDBSCAN - Hierarchical Density-Based Spatial Clustering of Applications with Noise. Führt
DBSCANüber verschiedene epsilon-Werte aus und integriert das Ergebnis, um eine Clusterbildung zu finden, die die beste Stabilität über epsilon liefert. Dies ermöglicht HDBSCAN, Cluster unterschiedlicher Dichten zu finden (im Gegensatz zuDBSCAN) und robuster gegenüber der Parameterauswahl zu sein. Lesen Sie mehr im Benutzerhandbuch.Hinzugefügt in Version 1.3.
- Parameter:
- min_cluster_sizeint, default=5
Die Mindestanzahl von Samples in einer Gruppe, damit diese Gruppe als Cluster betrachtet wird; Gruppierungen kleiner als diese Größe werden als Rauschen behandelt.
- min_samplesint, default=None
Der Parameter
k, der zur Berechnung des Abstands zwischen einem Punktx_pund seinem k-nächsten Nachbarn verwendet wird. WennNone, wird standardmäßigmin_cluster_sizeverwendet.- cluster_selection_epsilonfloat, default=0.0
Eine Abstandsschwelle. Cluster unterhalb dieses Wertes werden zusammengeführt. Siehe [5] für weitere Informationen.
- max_cluster_sizeint, default=None
Eine Grenze für die Größe von Clustern, die vom
"eom"-Cluster-Auswahlalgorithmus zurückgegeben werden. Es gibt keine Grenze, wennmax_cluster_size=Noneist. Hat keine Auswirkung, wenncluster_selection_method="leaf"ist.- metricstr oder callable, Standard=’euclidean’
Die Metrik, die bei der Berechnung des Abstands zwischen Instanzen in einem Feature-Array verwendet wird.
Wenn die Metrik ein String oder eine aufrufbare Funktion ist, muss sie eine der von
pairwise_distancesfür ihren Metrikparameter erlaubten Optionen sein.Wenn die Metrik „precomputed“ ist, wird X als Distanzmatrix angenommen und muss quadratisch sein.
- metric_paramsdict, Standard=None
Argumente, die an die Distanzmetrik übergeben werden.
- alphafloat, default=1.0
Ein Parameter zur Skalierung von Abständen, wie er bei robuster Single-Linkage-Analyse verwendet wird. Siehe [3] für weitere Informationen.
- algorithm{“auto”, “brute”, “kd_tree”, “ball_tree”}, default=”auto”
Genau welcher Algorithmus zur Berechnung von Kerndistanzen verwendet werden soll; Standardmäßig ist dies auf
"auto"gesetzt, was versucht, einenKDTree-Baum zu verwenden, wenn möglich, andernfalls einenBallTree-Baum. Sowohl die Algorithmen"kd_tree"als auch"ball_tree"verwenden denNearestNeighbors-Schätzer.Wenn das bei
fitübergebeneXdünnbesetzt (sparse) ist oder diemetricsowohl fürKDTreeals auch fürBallTreeungültig ist, wird auf den"brute"-Algorithmus zurückgegriffen.- leaf_sizeint, default=40
Blattgröße für Bäume, die für schnelle Abfrage nächster Nachbarn verantwortlich sind, wenn ein KDTree oder ein BallTree als Kerndistanzalgorithmen verwendet werden. Eine große Datensatzgröße und eine kleine
leaf_sizekönnen zu übermäßigem Speicherverbrauch führen. Wenn Ihnen der Speicher ausgeht, erwägen Sie die Erhöhung desleaf_size-Parameters. Ignoriert füralgorithm="brute".- n_jobsint, default=None
Anzahl der Jobs, die parallel ausgeführt werden, um Distanzen zu berechnen.
Nonebedeutet 1, es sei denn, Sie befinden sich in einemjoblib.parallel_backend-Kontext.-1bedeutet, alle Prozessoren zu verwenden. Weitere Details finden Sie im Glossar.- cluster_selection_method{“eom”, “leaf”}, default=”eom”
Die Methode, die zur Auswahl von Clustern aus dem verdichteten Baum verwendet wird. Der Standardansatz für HDBSCAN* ist die Verwendung eines Excess of Mass (
"eom")-Algorithmus, um die persistentesten Cluster zu finden. Alternativ können Sie stattdessen die Cluster an den Blättern des Baumes auswählen – dies liefert die feinkörnigsten und homogensten Cluster.- allow_single_clusterbool, default=False
Standardmäßig erzeugt HDBSCAN* keinen einzelnen Cluster. Wenn Sie dies auf True setzen, wird dies überschrieben und es werden einzelne Cluster-Ergebnisse zugelassen, falls Sie dies für Ihren Datensatz als gültiges Ergebnis betrachten.
- store_centersstr, default=None
Welche Clusterzentren, falls vorhanden, berechnet und gespeichert werden sollen. Die Optionen sind:
None, was keine Zentren berechnet oder speichert."centroid", was das Zentrum durch den gewichteten Durchschnitt ihrer Positionen berechnet. Beachten Sie, dass der Algorithmus die Euklidische Metrik verwendet und nicht garantiert, dass das Ergebnis ein beobachteter Datenpunkt ist."medoid", was das Zentrum durch den Punkt in den angepassten Daten berechnet, der die Distanz zu allen anderen Punkten im Cluster minimiert. Dies ist langsamer als „centroid“, da zusätzliche paarweise Distanzen zwischen Punkten desselben Clusters berechnet werden müssen, aber es garantiert, dass das Ergebnis ein beobachteter Datenpunkt ist. Der Medoid ist auch für beliebige Metriken gut definiert und hängt nicht von einer Euklidischen Metrik ab."both", was beide Formen von Zentren berechnet und speichert.
- copybool, default=False
Wenn
copy=True, wird bei jeder In-Place-Modifikation, die Daten überschreiben würde, die an fit übergeben wurden, zuerst eine Kopie erstellt, was garantiert, dass die Originaldaten unverändert bleiben. Derzeit gilt dies nur, wennmetric="precomputed"ist, wenn ein dichtes Array oder eine CSR-Sparse-Matrix übergeben wird und wennalgorithm="brute"ist.Geändert in Version 1.10: Der Standardwert für
copyändert sich vonFalsezuTruein Version 1.10.
- Attribute:
- labels_ndarray der Form (n_samples,)
Cluster-Labels für jeden Punkt im Datensatz, der an fit übergeben wurde. Ausreißer werden wie folgt gekennzeichnet:
Rauschproben erhalten das Label -1.
Proben mit unendlichen Elementen (+/- np.inf) erhalten das Label -2.
Proben mit fehlenden Daten erhalten das Label -3, auch wenn sie unendliche Elemente haben.
- probabilities_ndarray von Form (n_samples,)
Die Stärke, mit der ein Sample Mitglied seines zugewiesenen Clusters ist.
Clusterte Samples haben Wahrscheinlichkeiten, die proportional dazu sind, wie stark sie als Teil des Clusters bestehen bleiben.
Rauschproben haben eine Wahrscheinlichkeit von Null.
Samples mit unendlichen Elementen (+/- np.inf) haben eine Wahrscheinlichkeit von 0.
Samples mit fehlenden Daten haben die Wahrscheinlichkeit
np.nan.
- n_features_in_int
Anzahl der während des fits gesehenen Merkmale.
- feature_names_in_ndarray mit Form (
n_features_in_,) Namen der während fit gesehenen Merkmale. Nur definiert, wenn
XMerkmalnamen hat, die alle Zeichenketten sind.- centroids_ndarray von Form (n_clusters, n_features)
Eine Sammlung, die die Mittelpunkte jedes Clusters enthält, berechnet unter der Standard-Euklidischen Metrik. Die Mittelpunkte können „außerhalb“ ihrer jeweiligen Cluster liegen, wenn die Cluster selbst nicht konvex sind.
Beachten Sie, dass
n_clustersnur Nicht-Ausreißer-Cluster zählt. Das heißt, die Labels-1, -2, -3für die Ausreißer-Cluster sind ausgeschlossen.- medoids_ndarray von Form (n_clusters, n_features)
Eine Sammlung, die die Medoide jedes Clusters enthält, berechnet unter der Metrik, die an den
metric-Parameter übergeben wurde. Die Medoide sind Punkte in den ursprünglichen Clustern, die die durchschnittliche Distanz zu allen anderen Punkten in diesem Cluster unter der gewählten Metrik minimieren. Diese können als das Ergebnis der Projektion desmetric-basierten Zentrums zurück auf den Cluster betrachtet werden.Beachten Sie, dass
n_clustersnur Nicht-Ausreißer-Cluster zählt. Das heißt, die Labels-1, -2, -3für die Ausreißer-Cluster sind ausgeschlossen.
Siehe auch
Anmerkungen
Der Parameter
min_samplesschließt den Punkt selbst ein, während die Implementierung in scikit-learn-contrib/hdbscan dies nicht tut. Um die gleichen Ergebnisse in beiden Versionen zu erzielen, muss der Wert vonmin_sampleshier um 1 größer sein als der Wert, der in scikit-learn-contrib/hdbscan verwendet wird.Referenzen
Beispiele
>>> import numpy as np >>> from sklearn.cluster import HDBSCAN >>> from sklearn.datasets import load_digits >>> X, _ = load_digits(return_X_y=True) >>> hdb = HDBSCAN(copy=True, min_cluster_size=20) >>> hdb.fit(X) HDBSCAN(copy=True, min_cluster_size=20) >>> hdb.labels_.shape == (X.shape[0],) True >>> np.unique(hdb.labels_).tolist() [-1, 0, 1, 2, 3, 4, 5, 6, 7]
- dbscan_clustering(cut_distance, min_cluster_size=5)[Quelle]#
Gibt eine durch DBSCAN ohne Randpunkte erzielte Clusterbildung zurück.
Gibt eine Clusterbildung zurück, die für einen bestimmten cut_distance (oder epsilon) dem Ausführen von DBSCAN* entsprechen würde. DBSCAN* kann als DBSCAN ohne die Randpunkte betrachtet werden. Daher können diese Ergebnisse leicht von
cluster.DBSCANabweichen, aufgrund des Implementierungsunterschieds bei den nicht-Kernpunkten.Dies kann auch als eine flache Clusterbildung betrachtet werden, die aus einem Schnitt mit konstanter Höhe durch den Single-Linkage-Baum abgeleitet wurde.
Dies stellt das Ergebnis der Auswahl eines Schnittwerts für die robuste Single-Linkage-Clusterbildung dar.
min_cluster_sizeermöglicht es der flachen Clusterbildung, Rauschpunkte zu deklarieren (und Cluster kleiner alsmin_cluster_size).- Parameter:
- cut_distancefloat
Der gegenseitige Erreichbarkeitsabstandswert (mutual reachability distance), der zur Erzeugung einer flachen Clusterbildung verwendet wird.
- min_cluster_sizeint, default=5
Cluster, die kleiner als dieser Wert sind, werden als „Rauschen“ bezeichnet und bleiben in der resultierenden flachen Clusterbildung ungeclustert.
- Gibt zurück:
- labelsndarray der Form (n_samples,)
Ein Array von Cluster-Labels, eines pro Datenpunkt. Ausreißer werden wie folgt gekennzeichnet:
Rauschproben erhalten das Label -1.
Proben mit unendlichen Elementen (+/- np.inf) erhalten das Label -2.
Proben mit fehlenden Daten erhalten das Label -3, auch wenn sie unendliche Elemente haben.
- fit(X, y=None)[Quelle]#
Cluster basierend auf hierarchischer dichte-basierter Clusterbildung finden.
- Parameter:
- X{array-like, sparse matrix} von Form (n_samples, n_features), oder ndarray von Form (n_samples, n_samples)
Ein Feature-Array oder ein Array von Abständen zwischen Samples, wenn
metric='precomputed'ist.- yNone
Ignoriert.
- Gibt zurück:
- selfobject
Gibt sich selbst zurück.
- fit_predict(X, y=None)[Quelle]#
Cluster X und gibt die zugehörigen Cluster-Labels zurück.
- Parameter:
- X{array-like, sparse matrix} von Form (n_samples, n_features), oder ndarray von Form (n_samples, n_samples)
Ein Feature-Array oder ein Array von Abständen zwischen Samples, wenn
metric='precomputed'ist.- yNone
Ignoriert.
- Gibt zurück:
- yndarray der Form (n_samples,)
Clusterbeschriftungen.
- get_metadata_routing()[Quelle]#
Holt das Metadaten-Routing dieses Objekts.
Bitte prüfen Sie im Benutzerhandbuch, wie der Routing-Mechanismus funktioniert.
- Gibt zurück:
- routingMetadataRequest
Ein
MetadataRequest, der Routing-Informationen kapselt.
- get_params(deep=True)[Quelle]#
Holt Parameter für diesen Schätzer.
- Parameter:
- deepbool, default=True
Wenn True, werden die Parameter für diesen Schätzer und die enthaltenen Unterobjekte, die Schätzer sind, zurückgegeben.
- Gibt zurück:
- paramsdict
Parameternamen, zugeordnet ihren Werten.
- set_params(**params)[Quelle]#
Setzt die Parameter dieses Schätzers.
Die Methode funktioniert sowohl bei einfachen Schätzern als auch bei verschachtelten Objekten (wie
Pipeline). Letztere haben Parameter der Form<component>__<parameter>, so dass es möglich ist, jede Komponente eines verschachtelten Objekts zu aktualisieren.- Parameter:
- **paramsdict
Schätzer-Parameter.
- Gibt zurück:
- selfestimator instance
Schätzer-Instanz.
Galeriebeispiele#
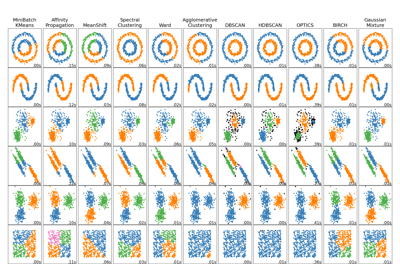
Vergleich verschiedener Clustering-Algorithmen auf Toy-Datensätzen